CHICKEN- AND GOOSE- TYPE LYSOZYME GENES IN THE JAPANESE EEL ANGUILLA JAPONICA
-
摘要: 研究以日本鳗鲡(Anguilla japonica Temminck et Schlegel)为研究对象, 根据其基因组数据库, 预测并扩增出2类, 共5个溶菌酶基因, 包括1个C-型溶菌酶和4个G-型溶菌酶, 分别命名为AJLysC、AJLysG1、AJLysG2、AJLysG3和AJLysG4。它们的cDNA全长分别为811、749、1352、1175和733 bp, 编码143、193、185、185和187个氨基酸。SignalP预测表明, AJLysC和AJLysG1的N-端分别包括15和19氨基酸的信号肽, 另外3种溶菌酶没有信号肽。基因组分析显示, AJLysC、AJLysG2、AJLysG3和AJLysG4的基因结构与其他鱼类的同类溶菌酶的基因结构相似, C-型溶菌酶具有4个外显子, G-型则具有5个。但是, AJLysG1的基因结构与其他鱼类G-型溶菌酶不同, 具有6个外显子, 与其他鱼类溶菌酶的蛋白序列比较, 发现AJLysG1缺失其他G-型溶菌酶存在的第2个酶活性位点氨基酸, 即天冬氨酸Asp。AJLysC与其他很多物种的C-型溶菌酶具有较高的同一性, 如与牙鲆的同一性为72.7%。G-型溶菌酶中AJLysG2、AJLysG3、AJLysG4彼此之间以及与其他物种G-型溶菌酶的同一性相对较高; 而AJLysG1与其他物种以及与其他3种G-型溶菌酶的同一性均不高, 且都在50%以下。组织表达分析显示, 所有5个溶菌酶基因在12种检测的组织中均有表达。C-型溶菌酶在胃及免疫相关组织的表达量较高; G-型溶菌酶在各组织/器官中的表达则差异较大, AJLysG1在皮肤和肌肉中的表达量最高, AJLysG2在免疫组织/器官如血液、头肾、体肾和鳃中表达量较高。经迟缓爱德华氏菌(Edwardsiella tarda)刺激48h后, 这5个溶菌酶基因在组织/器官中的表达量均有上调, 其中在血液、肠道和头肾等的上调较为显著。此外, 研究尝试重组表达这些抗菌肽, 获得了AJLysG2、AJLysG3和AJLysG4基因在鲤上皮瘤细胞(Epithelioma papulosum cyprinid, EPC)细胞中的表达, 重组蛋白表现出对溶壁微球菌(Micrococcus lyso-deikticus)生长的明显抑制作用。文章较全面地研究了日本鳗鲡溶菌酶基因的组成和类型及其表达变化, 并重组表达了部分基因, 这为进一步研究这些溶菌酶的功能, 特别是对病原微生物的作用奠定了基础。Abstract: Based on genomic database of the Japanese eel (Anguilla japonica), two types of lysozyme genes including one C-type and four G-type lysozyme genes were found and named as AJLysC, AJLysG1, AJLysG2, AJLysG3, and AJLysG4, which consisted of 809, 732, 1352, 1177 and 731 nucleotides that encode 143, 193, 185, 185 and 187 amino acids, respectively. AJLysC and AJLysG1 have 15 and 19 amino acids signal peptides, respectively, but other three lysozymes have no signal peptide. The gene structure of AJLysC, AJLysG2, AJLysG3 and AJLysG4 is similar to their corresponding lysozyme genes in other species in terms of exon-intron organization, with 4 exons for the C-type and 5 exons for the G-type. However, the gene structure of AJLysG1 differed from other fishes, with 6 exons, representing the first report in fish. Compared with other species, second catalytic residue, Asp, in AJLysG1 is missing. AJLysC shares high identity with C-type lysozymes of other species, having 72.7% identity with Japanese flounder (Paralichthys olivaceus). AJLysG2, AJLysG3 and AJLysG4 share high identity with each other and with those in other species, while AJLysG1 does not share high identity with neither G-type lysozymes of other species nor the other three, with the identity lower than 50%. The real-time quantitative PCR analysis showed that all five genes were expressed in all examined organs/tissues of eels. The C-type lysozyme gene expressed highly in stomach and modestly in immune related tissues. The expression of the four G-type lysozymes was tissue-dependent. AJLysG1 highly expressed in skin and muscle, while AJLysG2 highly expressed in blood, head kidney, trunk kidney and gills. All five genes were up-regulated in many organs/tissues by Edwardsiella tarda infection, especially in blood, intestine and head kidney. In addition, the recombinant proteins of AJLysG2, AJLysG3 and AJLysG4 obviously repressed the growth of Micrococcus lysodeikticus, however, their roles in inhibiting pathogenic bacteria of the Japanese eel need further investigation.
-
Keywords:
- Japanese eel /
- Lysozyme /
- Expression /
- Recombinant protein /
- Antimicrobial activity
-
溶菌酶广泛存在于有机体, 能水解细菌的细胞壁肽聚糖中由N-乙酰葡萄糖胺(N-acetylglucosa-mine)和N-乙酰胞壁酸(N-acetylmuramic acid)形成的β-1,4糖苷键, 造成细胞壁裂解, 引起细菌死亡, 从而在机体的防御系统中起着重要作用[1]。脊椎动物中有C-型(Chicken-type)和G-型(Goose-type)两种溶菌酶[2, 3]。C-型溶菌酶是最早发现并且研究最多的一类溶菌酶。最早被测序的鸡蛋蛋白溶菌酶和人的溶菌酶都是C-型溶菌酶, 它们也是应用较广的溶菌酶[4—6]。鱼类C-型溶菌酶的研究涉及许多种类, 包括一些重要的经济种类和模式种类, 如罗非鱼(Oreochromis niloticus Linnaeus)、虹鳟(Oncorhyn-chus mykiss Walbaum)、牙鲆(Paralichthys olivaceus Temminck et Schlegel)、鲤(Cyprinus carpio L.)、红鳍东方鲀(Fugu rubripes Temminck et Schlegel)、斑马鱼(Danio rerio Buchanan-Hamilton)等[7—11]。所有已发现的C-型溶菌酶均含有2个酶活性位点, 即第35个氨基酸谷氨酸(Glu)和第52个氨基酸天冬氨酸(Asp); 同时也存在8个保守的半胱氨酸(Cys), 这些半胱氨酸能形成4个二硫键, 维持蛋白结构的稳定[12, 13]。C-型溶菌酶的基因结构较为保守, 脊椎动物中已报道的C-型溶菌酶基因均为4个外显子和3个内含子的结构, 其中第一个外显子编码的蛋白质含有信号肽[14]。
G-型溶菌酶最初由Canfield等[15]于1967年在鹅蛋蛋白中发现, 因此命名为G-型溶菌酶。之后, 在很多其他鸟类中也报道了G-型溶菌酶, 如鸵鸟(Struthio camelus L.)、黑天鹅(Cygnus atratus L.)和鸡(Gallus domesticus L.)等[16—19]。然而, 直到2001年才在鱼类中报道了G-型溶菌酶[2]。目前, 已在很多鱼类中发现了G-型溶菌酶的存在, 包括斜带石斑鱼(Epinephelus coioides Temminck et Schlegel)、鳜(Siniperca chuatsi Basilewsky)、大西洋鲑(Salmo salar L.)、大西洋鳕(Gadus morhua L.)等[20—23]。目前, 对于哺乳动物G-型溶菌酶的研究相对较少。2003年, Irwin等[24]通过对基因组的分析, 首次在人(Homo sapiens L.)、鼠(Mus musculus L.)等物种中发现了G-型溶菌酶基因。在不同物种中, G-型溶菌酶外显子的数目差异较大。人的G-型溶菌酶基因有7个外显子[24]; 其他哺乳动物以及鸟类的G-型溶菌酶均为6个外显子, 这些溶菌酶均含有信号肽[24, 25]。在鱼类中, 已经报道的G型溶菌酶基因均为5个外显子, 并且很多鱼类的G-型溶菌酶没有信号肽, 因此曾认为鱼类G-型溶菌酶基因较少的外显子数目是造成鱼类溶菌酶中没有信号肽的原因[24]。然而, 在大西洋鲑和大西洋鳕中的G-型溶菌酶尽管只有5个外显子, 但经过变换转录起始位点(Transcription start site)可以影响其是否分泌到胞外[22, 23]。
鱼类C-型溶菌酶和G-型溶菌酶在鱼体的所有组织/器官中都有表达, 如草鱼(Ctenopharyngodon idella Valenciennes)[26]和菱鲆(Scophthalmus rhombus L.)[12]的肝脏、头肾、肾、脾、皮肤、肌肉、心脏、脑、肠道和鳃等组织/器官中均有溶菌酶基因的表达。经过病原或病原相关分子刺激后, 这两类溶菌酶基因在检测的组织/器官中出现上调表达[12, 26]。已有的研究显示, 鱼类的溶菌酶对一些病原细菌具有明显的抑制作用。例如, 草鱼的C-型溶菌酶和G-型溶菌酶蛋白对溶壁微球菌(Micrococcus lysodeikticus)、大肠杆菌(Escherichia coli)及嗜水气单胞菌(Aeromonas hydrophila)、副溶血弧菌(Vibrio parahaemolyticus)、迟缓爱德华氏菌(Edwardsiella tarda)、铜绿假单胞菌(Pseudomonas aeruginosa)等细菌均表现出溶菌活性[26]。重组的鳜G-型溶菌酶对溶壁微球菌的生长有明显的抑制作用[21]。斜带石斑鱼的G-型溶菌酶对大肠杆菌、溶藻弧菌(V. alginolyticus)、创伤弧菌(V. vulnificus)、副溶血弧菌、嗜水气单胞菌、荧光假单胞菌(P. fluorescens)等多种病原菌均有溶菌活性[20]。
日本鳗鲡(Anguilla japonica)属于鳗鲡目(Anguilliformes)、鳗鲡科(Anguillidae), 是重要的经济种类, 也是我国重要的水产养殖对象[27]。目前, 我国东南沿海分布的日本鳗鲡的苗种捕捞量逐年降低, 资源量不断减少, 无法满足养殖生产需求[28]。在日本, 日本鳗鲡已被列为国际濒危物种红色目录(IUCN Red List of Threatened Species)[29]。我国鳗鲡养殖业也曾一度依赖进口的欧洲鳗鲡(Anguilla anguilla L.), 但其也被列为国际濒危物种红色目录, 其苗种的出口受到了严格限制[30]。因此, 研究日本鳗鲡的生物学和养殖对于其种质资源保护、养殖生产和相关产业的发展都有重要意义。本研究通过分析日本鳗鲡的基因组数据库, 发现了5个溶菌酶基因, 在此基础上克隆了它们的基因序列, 揭示了它们的表达变化规律, 并在鱼类细胞系中表达了其中的3个溶菌酶基因, 为进一步研究这些溶菌酶的功能奠定了基础。
1. 材料与方法
1.1 目的基因的部分序列
根据日本鳗鲡基因组数据, 以其他鱼类的C-型和G-型溶菌酶基因作为参照, 用Blast软件进行分析, 获得日本鳗鲡可能的C-型和G-型溶菌酶基因的部分序列。
1.2 扩增目的基因cDNA全长
为确认预测的目的基因是否正确以及获得目的基因完整的cDNA全长序列, 本研究根据SMARTTM RACE cDNA Amplification Kit (Clontech)的说明, 以预测的基因序列为模板设计并合成引物, 并对目的基因的3′及5′端分别进行扩增。实验用鱼取自于集美大学海水养殖场, 体重约250 g。经暂养后, 采集头肾及肝脏, 采用Trizol® Reagent (Invitrogen)提取总RNA, 然后采用SMARTTM RACE cDNA Amplification Kit合成模板。基因的扩增采用槽式PCR, 即第一轮采用降落PCR (Touchdown PCR)经UPM (Universal Primer Mix)和外引物进行扩增; 第二轮采用常规PCR经NUP (Nested Universal Primer)和内引物进行扩增。其中, AJLysC、AJLysG2、AJLysG3和AJLysG4使用Ex taq (TaKaRa)进行扩增; AJLysG1采用扩增性能较好的Advantage 2(Clontech)进行实验。PCR扩增产物经1%琼脂糖凝胶电泳后切胶回收。回收产物连接至pMD19-T (TaKaRa), 然后转化至大肠杆菌(Escherichia coli)TOP10感受态细胞; 转化细菌的阳性克隆被送测序公司(金斯瑞生物科技或生工生物)进行测序。将5′-RACE和3′-RACE测序结果拼接, 即获得cDNA序列全长。本研究所有使用的引物见表 1。
表 1 本研究使用的引物Table 1. Primers used in the current study引物Primer 序列Sequence (5′-3′) 用途Usage AJLysCF-out ACCGACGGATCGACGGACTA 扩增开放阅读框 AJLysCF-in AGTCGCTGGTGGTGTAACGA AJLysCR-out AGTGGTTCCGCCATGCCAC AJLysCR-in TGGGATCTCTGACCACACGC AJLysG1F-out CACAAAGACCGAATCAACAGGG AJLysG1F-in GTTCCATTTCCCGTGAGTCCAG AJLysG1R-out CAGAAGTTCAGTGCCTTGACAGTT AJLysG1R-in TGCTGCTCGCTATTCCACTGTCC AJLysG2F-out GTGGGACGGGCTCAGCAAAT AJLysG2F-in CAAGCGATACCACACGCCAAG AJLysG2R-in TGGCGTGTGGTATCGCTTGTC AJLysG3F-out GCATCCCACAAGTTGGCACAG AJLysG3F-in CGGAGTTCTTACAAACGGCTGG AJLysG3R-out TTCTTGTTGAACCACTGGGCTC AJLysG3R-in TGTCCATTCGCTCATAAGTCCG AJLysG4F-out TCACAACAAACCGCTAATCAGGA AJLysG4F-in CTGTGGTCGCCGCTATCATTTC AJLysG4R-out CAACATCATTGGAGTAGTCACGCC AJLysG4R-in ATTGTAGGCTGCTATTCCACCCAT CF AATCATCTGTCACTTCAGAATG 扩增全基因 CR GAAAAGGTGCTTCTCTCTGG G1F TTGCTAGAAAATTACTAAAAGTAGAA G1R GATTGCATTTCACCCTGTGT G2F CCACAGGGAAGAATAACAAC G2R TCCAAGGCTTATTTTTCTCT G3F AGCCAGAAGACAGAACATTAC G3R TTACAAGGTTTTCATCATATTTT G4F CTGAAGAGTTTGTGGAAGTTC G4R AAGATTTCATTCACTCCTTTTA ActinF CCCAAGACATCAGGGCGTGA 定量分析 ActinR CTCAAACATGATCTGCGTCATC CQF CCTGGGAGACTGGGTCTG CQR CCACCCATGCTCTGATCC G1QF TGGAATAGCGAGCAGCACAACT G1QR CCACGGTCAGAAGCCAGATTGA G2QF GGACTCATGCAGATTGACAAGC G2QR TATCCCACCTTTAAATTGGTGCT G3QF GGACTCATGCAGGTTGACAA G3QR GATATTCCTCCTTTTAATTGTTGC G4QF GGCTTATGCAGGTTGACAAG G4QR CTGCTATTCCACCTTTAAGTTGT EUAJLysG2F CGGAATTCGCCACCATGGCAATGACCTGCATTTATGGGGA 构建表达载体 EUAJLysG2R GCGGATCC AAAGCCATTCCGCTTGAACC EUAJLysG3F GCGGATCCGCCACCATGGACGGCATTTATGGCGA EUAJLysG3R CCAAGCTTAAAACCATTCTTGTTGAACCACTGG EUAJLysG4F GCGGATCCGCCACCATGGCCTGCATTTACGGCGA EUAJLysG4R CCAAGCTTAAAACCATTCCTTTTGTAGAACTGG 1.3 目的基因DNA的扩增
为研究分析目的基因, 根据所获得的cDNA序列设计并合成了扩增DNA全长的引物(表 1)。采用QIAamp DNA Mini Kit (QIAGEN)从肌肉组织中纯化DNA, 目的基因DNA使用LA Taq (TaKaRa)进行扩增, 经测序获DNA序列。
1.4 生物信息学分析
目的基因的预测采用本地Blast (blast-2.2.22)。引物的设计采用Primer Premier 5.0和6.0。序列经查找重叠区域而拼接完整后采用ORF Finder (http://www.ncbi.nlm.nih.gov/gorf/gorf.htmL)查找开放阅读框。使用ExPASy网站(http://www.expasy.org)中的Translate软件推导氨基酸序列。信号肽的分析采用SignalP 4.1 Server (http://www.cbs.dtu.dk/ser-vices/SignalP/)。推导出的蛋白的参数分析采用ExPASy网站中ProtParam tool软件进行分析。序列同源性比对分析采用软件MatGat 2.02[31]。蛋白质二硫键的预测采用Predict Protein (https://www.predictprotein.org/)。进化树使用MEGA 5.2经邻接法(Neighbor-Joining, NJ)构建, bootstraps设置为1000。基因结构采用Spidey (http://www.ncbi.nlm. nih.gov/IEB/Research/Ostell/Spidey/spideyweb.cgi)进行分析。
1.5 溶菌酶基因在正常鱼和受病原刺激鱼体的表达分析
使用大豆酪蛋白消化液培养基(Tryptic soytone broth medium, TSB), 30℃培养迟缓爱德华氏菌, 待菌液浑浊后, 测定OD540值(Spectrumlab 721N型, 上海精密科学仪器有限公司)。按照比例使用TSB将E. tarda梯度稀释, 将稀释后的菌液涂布在大豆琼脂培养基(Tryptic soy agar, TSA)平板上, 生化培养箱中30℃培养过夜, 第2天对菌落进行计数。经计数分析显示, 当OD540为0.5时, 每毫升菌液对应的细菌菌落总数约为2.5×108。2200×g离心15min回收菌体, 弃培养基后用合适体积的PBS缓冲液(Phosphate buffer saline, pH 7.4)悬浮菌体; 将菌体再次2200×g离心15min, 更换PBS, 将菌液浓度调节至1.0×107 cfu/mL用于感染鱼体。
将经过暂养2周的日本鳗鲡(购自广东省江门市, 个体体质量约为500 g)随机分成4组: 空白组、对照组和两个刺激组, 每组均为4条鱼。空白组不注射, 对照组每条鱼腹腔注射1.0 mL灭菌后的PBS缓冲液; 刺激组每条鱼注射1 mL浓度调节至1.0×107 cfu/mL的迟缓爱德华氏菌菌液。对照组在注射PBS缓冲液后24h采样, 刺激组分别在注射菌液24h和48h后采样。采样时, 将日本鳗鲡经丁香酚醇溶液麻醉, 分别采集血液、肠道、头肾、体肾、胃、肝、脾、鳃、脑、心、皮肤和肌肉, 共计12个组织/器官。然后, 将样品浸入冰上预冷的1 mL Trizol® Reagent中, 提取样品总RNA或者将样品保存于超低温冰箱中备用。
提取总RNA后, 用Nano Drop 2000(Thermo)测定浓度, 取总RNA 500—1000 ng使用DNaseⅠ (Thermo)处理, 然后用M-MuLV酶逆转录试剂盒(Thermo)合成cDNA。为降低或消除定量分析时提取总RNA过程中残留的基因组DNA的影响, 实验引物采用跨内含子的方法设计, 采用定量分析引物(表 1)扩增目的基因, 连接至pMD18-T, 然后测序确认。将测序正确的克隆扩大培养, 提取质粒(Plasmid Mini Kit, Omega), 使用分光光度计(NanoDrop 2000, Thermo)测定质粒的浓度。质粒拷贝数的计算参照已报道的方法[32]。将测定浓度后的质粒按照10倍梯度稀释, 然后使用Bio-rad公司定量PCR系统(CFX96TM Real-Time System)构建标准曲线。实时荧光定量分析(Real-time quantitative reverse transcription-PCR, rt-qPCR)的反应体系为20 μL, 包括iQTM SYBR Green Super Mix (2×) 10 μL, 正向引物(10 μmol/L) 1 μL, 反向引物(10 μmol/L) 1 μL, 模板1 μL, 和ddH2O 7 μL。rt-qPCR的程序如下: 预变性95℃ 3min; 95℃变性10s, 60℃退火20s, 72℃延伸20s, 80℃读板5s, 共41个循环。
定量分析时, 所有样品均重复3次。健康组各组织/器官目的基因和拷贝数根据标准曲线获得; 经过β-actin进行校正后即获得各目的基因在不同组织/器官中的相对表达量。日本鳗鲡经迟缓爱德华氏菌刺激后目的基因在各组织/器官中的变化采用2–∆∆Ct方法进行分析, 其中–∆∆Ct=(Ct目的基因–Ctβ-actin)实验组–(Ct目的基因在脑中的表达–Ctβ-actin)24h对照组[33, 34]。数据采用SPSS进行One-way ANOVA分析, 若P<0.05即表示差异显著。
1.6 溶菌酶基因的重组表达与重组蛋白的活性分析
分别使用EcoRⅠ/BamHⅠ和BamHⅠ/Hind Ⅲ酶切位点将AJLysG2、AJLysG3和AJLysG4的开放阅读框序列(去掉终止密码子及信号肽编码序列)连入pcDNATM 3.1/myc-His(-) A (Invitrogen)真核表达质粒。
采用鲤上皮瘤细胞(Epithelioma papulosum cyprinid, EPC)进行重组表达。利用M199培养基+10%胎牛血清(Fetal bovine serum, FBS), 于28℃在CO2培养箱培养。将构建成功的pcDNA3.1-AJLysG2、-AJLysG3、-AJLysG4以及pcDNA3.1空载体质粒分别使用Lipofectamine 2000(Invitrogen)转染75 cm2培养瓶中的EPC细胞。转染24h后, 去掉上清培养基并用含有蛋白酶抑制剂cocktail (Roche)的RIPA细胞裂解液裂解细胞。裂解产物经12000×g, 4℃离心10min, 获取上清液用于后续实验。
转染后的EPC细胞裂解产物上清液经SDS-PAGE电泳后转PVDF膜(孔径0.2 μm), 用含有3%脱脂奶粉的TBS缓冲液常温封闭1h, 然后使用含鼠抗Myc单抗(Sigma, 1鲶2000)及1.5%脱脂牛奶的TBS缓冲液4℃孵育过夜。洗膜后, 二抗(羊抗鼠IgG-HP多抗, Pierce, 1鲶5000)孵育1h。TBST缓冲液洗膜后, 在化学发光仪(Bio-Rad)中显色和拍照。根据蛋白标准品(MagicMarkTM XP Western Protein Standard, Novex)估测目的蛋白浓度。
溶菌酶抑菌实验参照Hikima等[2]以及Sun等[21]的方法进行。磷酸盐缓冲液(pH 6.5)、TSB培养基和琼脂粉, 以100鲶3鲶1 (v鲶w鲶w)比例混合灭菌。灭菌后, 固体培养基在50℃水浴保温。TSB液体培养基培养溶壁微球菌, 当菌液OD600为0.4时, 以1鲶200 (v鲶v)的比例将菌液混入50℃固体培养基中, 倒在9 cm平板上。培养基凝固后打孔并分别加入转染pcDNA3.1-AJLysG2、-AJLysG3、-AJLysG4和pcDNA3.1空载体质粒的EPC细胞裂解产物上清液以及鸡蛋蛋白溶菌酶(Hen egg white lysozyme, HEWL)。然后将平板置于28℃培养约18h。观察样品在平板上产生的溶菌环的大小并拍照记录。
2. 结果
2.1 C-型和G-型溶菌酶基因的cDNA和DNA序列分析
通过分析基因组数据库以及PCR扩增, 鉴定了日本鳗鲡的1个C-型和4个G-型溶菌酶基因, 将它们分别命名为AJLysC、AJLysG1、AJLysG2、AJLysG3和AJLysG4。它们的cDNA全长分别为811、749、1352、1175和733 bp, 编码143、193、185、185和187个氨基酸。SignalP预测表明, AJLysC和AJLysG1的N-端含有信号肽, 成熟肽分别为128和174氨基酸; 另外3种溶菌酶没有信号肽(表 2)。在这些基因的3′端非编码区, 其cDNA序列均有多腺苷酸化信号(AATAAA), 其中AJLysG1和AJLysG2在ploy A之前分别含有1和4个不稳定信号(ATTTA)(未显示, 可见GenBank序列)。
表 2 日本鳗鲡溶菌酶基因cDNA序列及其氨基酸序列Table 2. The cDNA sequences and deduced amino acids of lysozyme genes in the Japanese eel基因名称Gene name 登录号GenBank Acc. No. 总长度Length (bp) 5′端非编码区5′ UTR* 3′端非编码区3′ URT* 氨基酸数目No. of aa 成熟肽氨基酸数目No. of aa in mature peptide 等电点pI C-type AJLysC KM454476 811 83 296 143 128 8.48 G-type AJLysG1 KM454477 749 90 77 193 174 9.42 AJLysG2 KM454478 1352 35 759 185 185 8.89 AJLysG3 KM454479 1175 82 529 185 185 9.00 AJLysG4 KM454480 733 56 113 187 187 7.05 注: * UTR表示untranslated region 氨基酸序列比对结果显示, AJLysC与菱鲆的同一性最高, 为76.9%, 其次是牙鲆、红鳍东方鲀和塞内加尔鳎, 分别为72.7%、69.2%和67.8%, 与鲤和草鱼的比较低, 只有38.6%和37.4%。G-型溶菌酶中AJLysG2、AJLysG3和AJLysG4之间的同一性相对较高, 在50%以上; 而AJLysG1与其他溶菌酶的则较低, 均在50%以下(表 3)。与其他鱼类的相对应的溶菌酶序列比较发现, 日本鳗鲡的C-型溶菌酶和其他鱼类的C-型溶菌酶都有信号肽, 有8个保守的半胱氨酸和2个保守的催化残基Glu35和Asp52; 这8个半胱氨酸能形成4个二硫键(图 1)。日本鳗鲡的G-型溶菌酶AJLysG2、AJLysG3和AJLysG4均具有3个保守的催化残基, 即Glu73、Asp86和Asp97, 但AJLysG1的第二个催化残基, 即Asp86缺失(图 2)。此外, AJLysG1存在2个半胱氨酸, 但预测结果显示, 这2个半胱氨酸并不形成二硫键(图中未显示)。
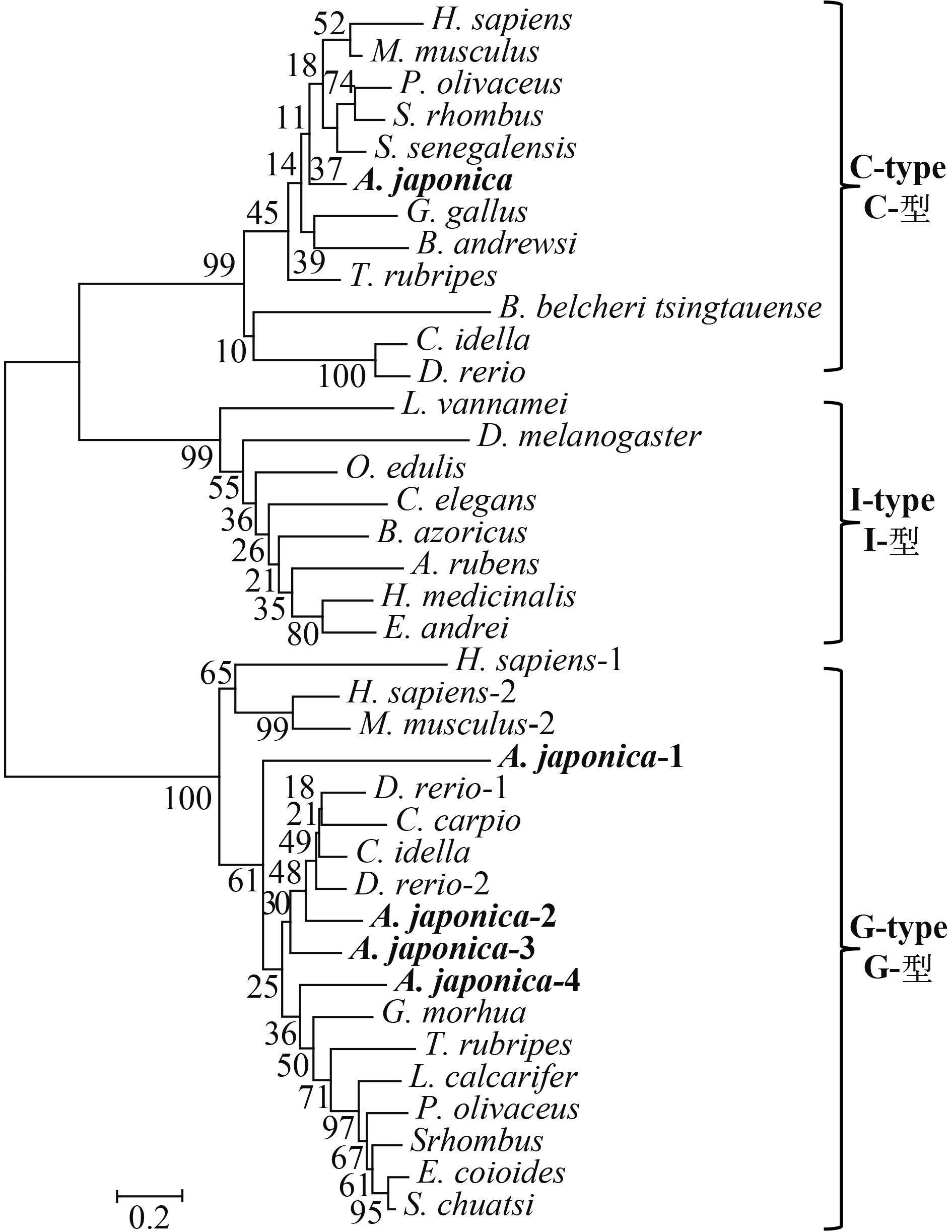
表 3 日本鳗鲡G-型溶菌酶之间以及与其他鱼类G-型溶菌酶的同一性分析Table 3. Comparison among G-type lysozymes of the Japanese eel and other species of fish鱼类的G-型溶菌酶G-type lysozymes in fish 日本鳗鲡G-型溶菌酶G-type lysozymes in Japanese eel AJLysG1 AJLysG2 AJLysG3 AJLysG4 日本鳗鲡AJLysG1 38.9 41.8 34.5 日本鳗鲡AJLysG2 38.9 68.4 58.8 日本鳗鲡AJLysG3 41.8 68.4 63.6 日本鳗鲡AJLysG4 34.5 58.8 63.6 草鱼Grass carp 36.9 76.2 65.8 59.4 鲤Common carp 34.5 65.4 64.7 54.5 斑马鱼Zebrafish 37.9 73 68.4 57.2 红鳍东方鲀Fugu rubripes 31.1 54.7 49.5 49.5 尖吻鲈Barramundi perch 31.5 57.1 59.7 59.2 牙鲆Japanese flounder 34.1 52.8 53.3 51.8 菱鲆Brill 30.2 56.4 55.3 53.8 斜带石斑鱼Orange-spotted grouper 30 55.1 52.5 52 鳜Mandarin fish 29.7 55.1 54.5 53.5 注: AJLysG1、AJLysG2、AJLysG3、AJLysG4为日本鳗鲡的4个G-型溶菌酶, 其GenBank序列号见表 2。草鱼grass carp Ctenopharyngodon idella: 代表草鱼G-型溶菌酶, 其GenBank登录号为ACF41166; 鲤common carp Cyprinus carpio: BAA95698; 斑马鱼zebrafish Danio rerio: AAI34160; 红鳍东方鲀Fugu rubripes Takifugu rubripes: NP_001027764; 尖吻鲈barramundi perch Lates calcarifer: ABV66069; 牙鲆Japanese flounder Paralichthys olivaceus: BAB17215; 菱鲆brill Scophthalmus rhombus: BAF75845; 斜带石斑鱼orange-spotted grouper Epinephelus coioides: AAL08021; 鳜mandarin fish Siniperca chuatsi: AAU86896 Note: AJLysG1, AJLysG2, AJLysG3 and AJLysG4 are four G-type lysozymes in the Japanese eel with their GenBank accession numbers indicated in Tab. 2 本研究采用邻接法构建了系统进化树(图 3)。从图中可以看出, 日本鳗鲡的AJLysC与其他脊椎动物的C-型溶菌酶聚为一大支。鱼类的G-型溶菌酶聚为一大支, 而日本鳗鲡的AJLysG2、AJLysG3、AJLysG4与其他硬骨鱼类G-型溶菌酶聚为一支, 再与AJLysG1聚在一起。
2.2 C-型和G-型溶菌酶的基因结构
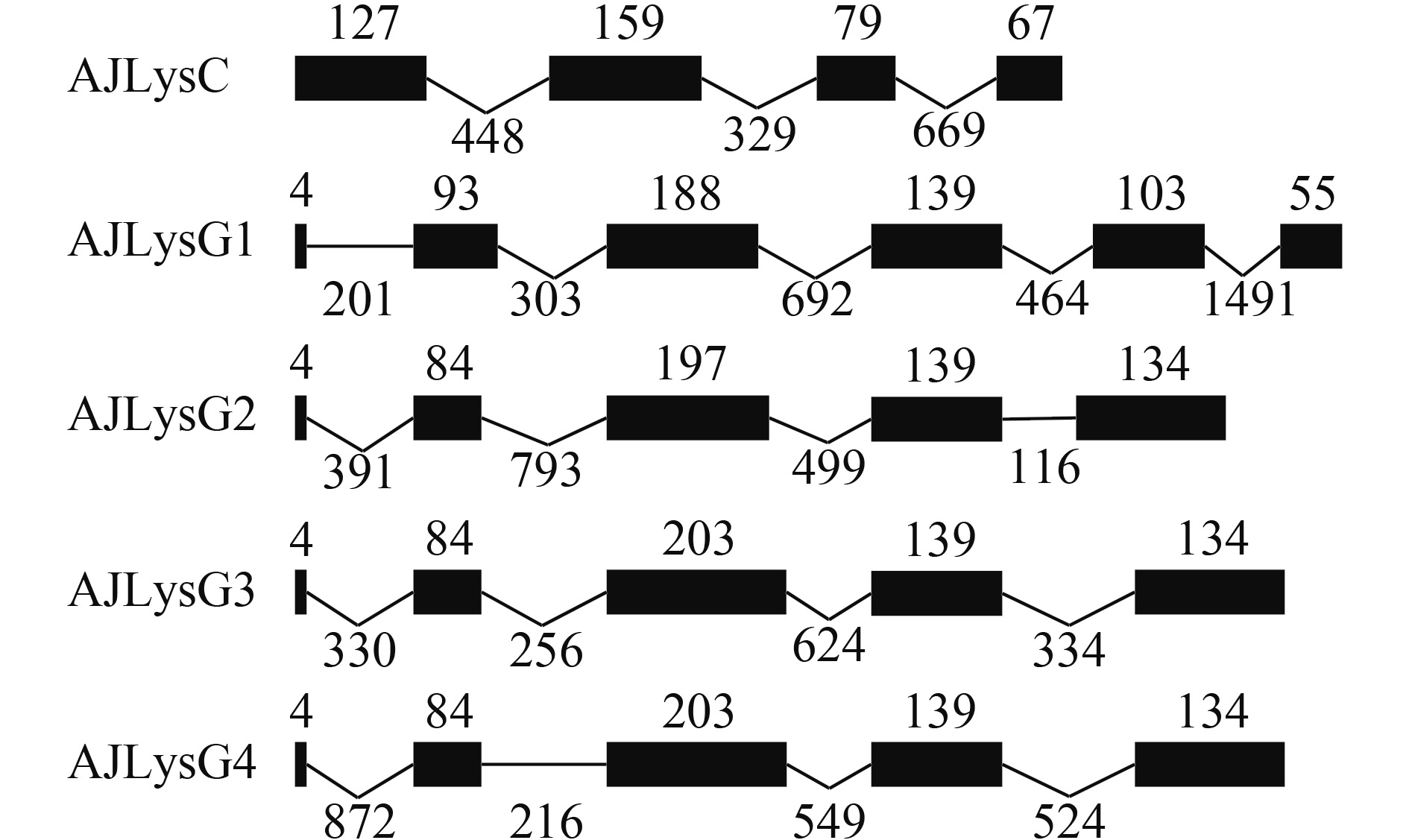
日本鳗鲡的5个溶菌酶基因的基因组结构信息见图 4。这5个溶菌酶基因的基因组结构均符合经典的剪切模式(GT/内含子/AG)。AJLysC基因组为1878 bp, 含有4个外显子和3个内含子。AJLysG1基因组长3733 bp, 含有6个外显子, 5个内含子。AJLys-G2、AJLysG3和AJLysG4的基因组长度分别为2357、2108和2725 bp, 这3个G-型溶菌酶基因和其他鱼类同类溶菌酶基因一样, 均含5个外显子和4个内含子, 并且对应的外显子碱基数目接近, 特别是AJLysG3和AJLysG4, 其对应的外显子的数目完全相同。
![]() 图 1 日本鳗鲡C-型溶菌酶AJLysC和其他鱼类C-型溶菌酶的氨基酸序列比较黑色框中为信号肽, 8个半胱氨酸用阴影表示, 黑色框与阴影共同标注的是酶活性位点。“*”表示相同的氨基酸, “:”和“.”相似的氨基酸。比对的序列分别为: SRLysC, 代表菱鲆的C-型溶菌酶, GenBank登录号为BAF75844; SSLyC, 塞内加尔鳎, ABY84356; TRLysC, 红鳍东方鲀, NP_001027914; POLysC, 牙鲆, BAB17215; CILysC, 草鱼, ACF41166; DRLysC, 斑马鱼, NP_631919Figure 1. Comparison of deduced amino acid sequence of C-type lysozyme, AJLysC of the Japanese eel with homologues of other species of fishSignal peptides are boxed. The 8 cysteine residues are shaded, and the catalytic residues are shaded in black boxes. (*) indicates identical amino acids, and (:) or (.) the similar amino acids. SRLysC represents C-type lysozyme in brill with the GenBank accession number being BAF75844; SSLyC, Senegalese sole, ABY84356; TRLysC, Fugu rubripes, NP_001027914; POLysC, Japanese flounder, BAB17215; CILysC, grass carp, ACF41166; DRLysC, zebrafish, NP_631919
图 1 日本鳗鲡C-型溶菌酶AJLysC和其他鱼类C-型溶菌酶的氨基酸序列比较黑色框中为信号肽, 8个半胱氨酸用阴影表示, 黑色框与阴影共同标注的是酶活性位点。“*”表示相同的氨基酸, “:”和“.”相似的氨基酸。比对的序列分别为: SRLysC, 代表菱鲆的C-型溶菌酶, GenBank登录号为BAF75844; SSLyC, 塞内加尔鳎, ABY84356; TRLysC, 红鳍东方鲀, NP_001027914; POLysC, 牙鲆, BAB17215; CILysC, 草鱼, ACF41166; DRLysC, 斑马鱼, NP_631919Figure 1. Comparison of deduced amino acid sequence of C-type lysozyme, AJLysC of the Japanese eel with homologues of other species of fishSignal peptides are boxed. The 8 cysteine residues are shaded, and the catalytic residues are shaded in black boxes. (*) indicates identical amino acids, and (:) or (.) the similar amino acids. SRLysC represents C-type lysozyme in brill with the GenBank accession number being BAF75844; SSLyC, Senegalese sole, ABY84356; TRLysC, Fugu rubripes, NP_001027914; POLysC, Japanese flounder, BAB17215; CILysC, grass carp, ACF41166; DRLysC, zebrafish, NP_631919![]() 图 2 日本鳗鲡G-型溶菌酶和其他鱼类G-型溶菌酶氨基酸序列的比较阴影标注的是酶活性位点。“*”表示相同的氨基酸, “:”和“.”表示相似的氨基酸。日本鳗鲡的4个G-型溶菌酶AJLysG1、AJLysG2、AJLysG3、AJLysG4的GenBank登录号见表 1。比对的其他鱼类的G-型溶菌酶分别为: ECLysG, 石斑鱼的G-型溶菌酶, GenBank登录号为AAL08021; SCLysG, 鳜, AAU86896; SRLysG, 菱鲆, BAF75845; LClysG, 尖吻鲈, ABV66069; POLysG, 牙鲆, BAB17215; TRLysG, 红鳍东方鲀, NP_001027764; CCLysG, 鲤, BAA95698; DRLysG, 斑马鱼, AAI34160; CILysG, 草鱼, ACF41166Figure 2. Comparison of deduced amino acid sequences of G-type lysozymes of the Japanese eel with homologues of other species of fishCatalytic residues are shaded. (*) indicates identical amino acids, and (:) or (.) indicate similar amino acids. AJLysG1, AJLysG2, AJLysG3, AJLysG4 represent G-type lysozymes of Japanese eel with GenBank accession numbers indicated in Tab. 1. ECLysG represents G-type lysozyme from orange-spotted grouper with the GenBank accession number being AAL08021; SCLysG, mandarin fish, AAU86896; SRLysG, brill, BAF75845; LClysG, barramundi perch, ABV66069; POLysG, Japanese flounder, BAB17215; TRLysG, fugu rubripes, NP_001027764; CCLysG, common carp, BAA95698; DRLysG, zebrafish, AAI34160; CILysG, grass carp, ACF41166
图 2 日本鳗鲡G-型溶菌酶和其他鱼类G-型溶菌酶氨基酸序列的比较阴影标注的是酶活性位点。“*”表示相同的氨基酸, “:”和“.”表示相似的氨基酸。日本鳗鲡的4个G-型溶菌酶AJLysG1、AJLysG2、AJLysG3、AJLysG4的GenBank登录号见表 1。比对的其他鱼类的G-型溶菌酶分别为: ECLysG, 石斑鱼的G-型溶菌酶, GenBank登录号为AAL08021; SCLysG, 鳜, AAU86896; SRLysG, 菱鲆, BAF75845; LClysG, 尖吻鲈, ABV66069; POLysG, 牙鲆, BAB17215; TRLysG, 红鳍东方鲀, NP_001027764; CCLysG, 鲤, BAA95698; DRLysG, 斑马鱼, AAI34160; CILysG, 草鱼, ACF41166Figure 2. Comparison of deduced amino acid sequences of G-type lysozymes of the Japanese eel with homologues of other species of fishCatalytic residues are shaded. (*) indicates identical amino acids, and (:) or (.) indicate similar amino acids. AJLysG1, AJLysG2, AJLysG3, AJLysG4 represent G-type lysozymes of Japanese eel with GenBank accession numbers indicated in Tab. 1. ECLysG represents G-type lysozyme from orange-spotted grouper with the GenBank accession number being AAL08021; SCLysG, mandarin fish, AAU86896; SRLysG, brill, BAF75845; LClysG, barramundi perch, ABV66069; POLysG, Japanese flounder, BAB17215; TRLysG, fugu rubripes, NP_001027764; CCLysG, common carp, BAA95698; DRLysG, zebrafish, AAI34160; CILysG, grass carp, ACF41166![]() 图 4 日本鳗鲡的1个C-型(AJLysC)和4个G-型(AJLysG1-4)溶菌酶基因结构图黑色框表示外显子, 黑色线条表示内含子。其对应的数字表示外显子或内含子的大小。黑色框和直线与数字成比例, 而折线则与数字不成比例Figure 4. Gene organization of the C-type (AJLysC) and four G-type (AJLysG1-4) lysozymes of the Japanese eelThe black boxes represent exons, and the lines represent introns. The numbers indicate the size of exons or introns. The black boxes and the straight lines are proportional to the size of exons and introns respectively, while the concave lines are not proportional
图 4 日本鳗鲡的1个C-型(AJLysC)和4个G-型(AJLysG1-4)溶菌酶基因结构图黑色框表示外显子, 黑色线条表示内含子。其对应的数字表示外显子或内含子的大小。黑色框和直线与数字成比例, 而折线则与数字不成比例Figure 4. Gene organization of the C-type (AJLysC) and four G-type (AJLysG1-4) lysozymes of the Japanese eelThe black boxes represent exons, and the lines represent introns. The numbers indicate the size of exons or introns. The black boxes and the straight lines are proportional to the size of exons and introns respectively, while the concave lines are not proportional2.3 C-型和G-型溶菌酶基因在健康鱼体组织/器官的表达
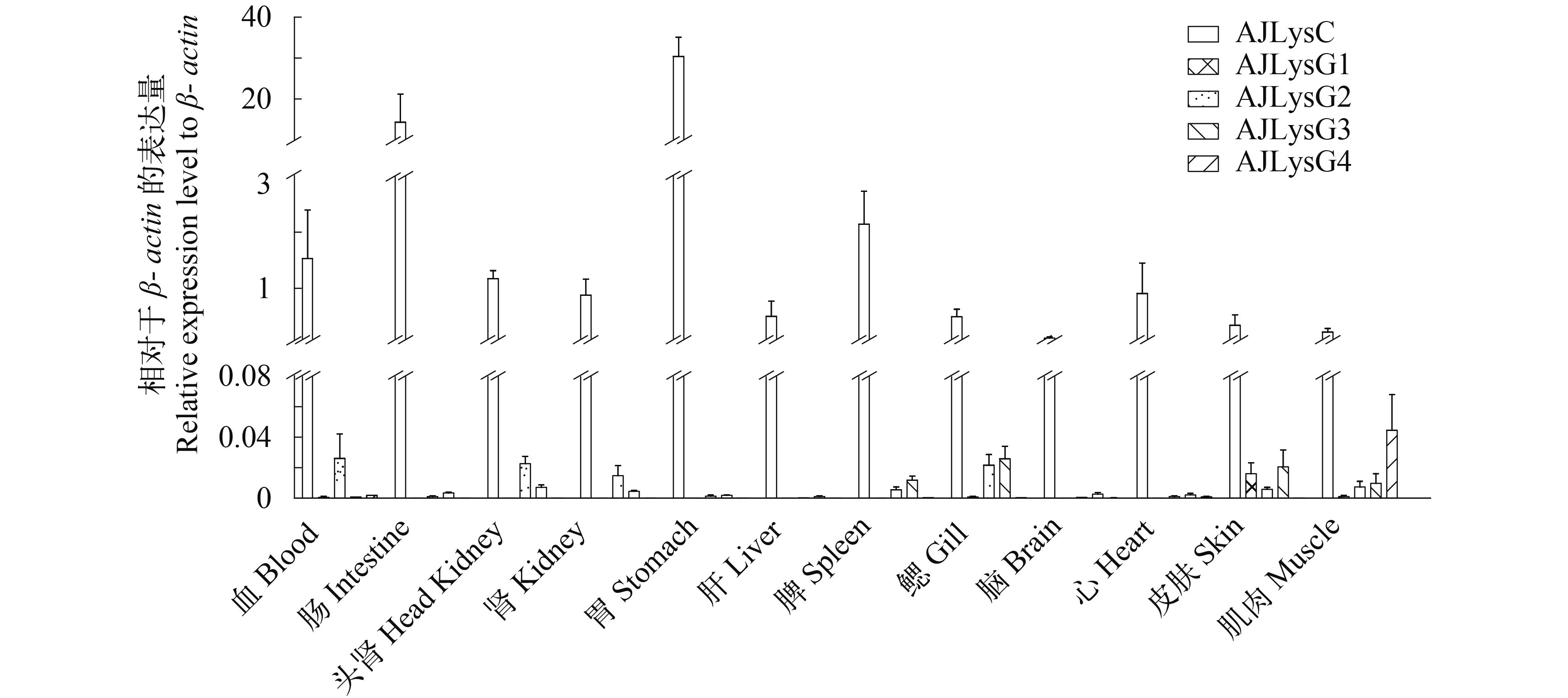
荧光定量PCR分析显示, 克隆到的1个C-型溶菌酶和4个G-型溶菌酶基因在健康鱼体的组织/器官都有表达(图 5、图6)。然而, 这5个溶菌酶基因的表达水平及其在不同组织/器官的表达似乎都不一样。C-型溶菌酶在所有组织/器官中的表达量都比相对应的组织/器官中G-型溶菌酶的表达量高, C-型溶菌酶在胃中的表达量最高, 是β-actin表达量的30倍; 在肠和脾中的表达量也很高, 分别是β-actin表达量的14和2倍(图 5)。G-型溶菌酶基因在所有组织/器官中的表达都较低。AJLysG1在各组织/器官中的表达差异很大, 在皮肤的表达量最高, 却只有β-actin表达量的0.011倍, 在头肾、肝和脾等免疫器官中几乎不表达。AJLysG2基因在各组织/器官中的表达差异较小, 在鳃中表达量最高, 为β-actin表达量的0.023倍; 在血液、头肾、鳃和体肾中的表达量相对较高。AJLysG3基因在鳃中的表达量最高, 为β-actin表达量的0.017倍。AJLysG4基因在在肌肉中的表达水平最高, 在其他组织/器官中的表达普遍较低。
2.4 C-型和G-型溶菌酶基因在迟缓爱德华氏菌感染后的表达变化
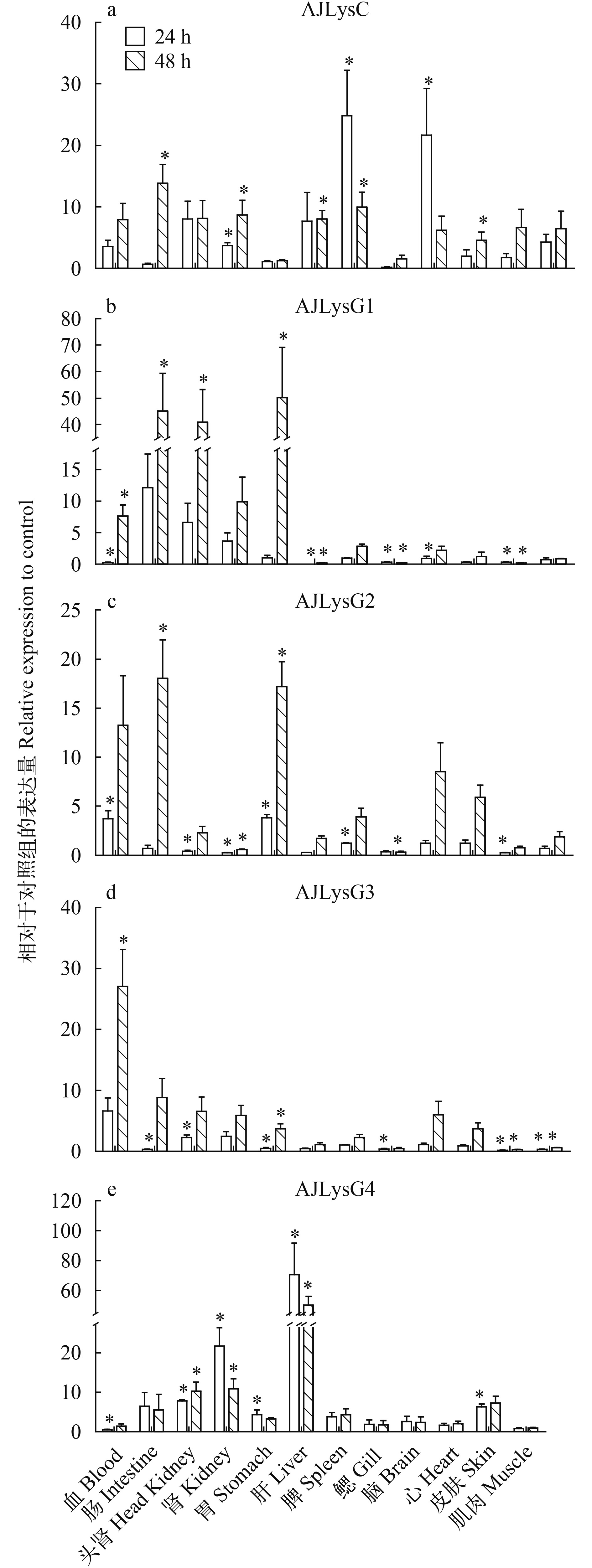
经定量分析显示, 迟缓爱德华氏菌刺激后, 溶菌酶基因在组织/器官中均有明显的上调表达。刺激后, AJLysC在各组织/器官中的表达均上调, 24h后在脾和脑中表达量上调25和22倍(图 6A)。AJLysG1在刺激48h后上调更明显, 其表达量在胃、肠和头肾中上调倍数分别为50、45和41(图 6B)。AJLysG2在刺激48h后表达上调更明显, 在肠道、胃和血液中的上调倍数分别为18、17和13(图 6C)。与AJLysG1和AJLysG1相似, AJLysG3在刺激后48h上调较为明显, 如血液中表达上调27倍(图 6D)。AJLysG4的表达量在刺激后24h和48h均有较明显的上调, 在肝脏中分别上调了67和50倍(图 6E)。刺激后, 目的基因在部分组织中表达量也存在下调的现象, 如AJLysG1在刺激24h后, 其在鳃和心脏中的表达量分别下调了3和3.4倍。
2.5 溶菌酶重组蛋白表达及活性分析
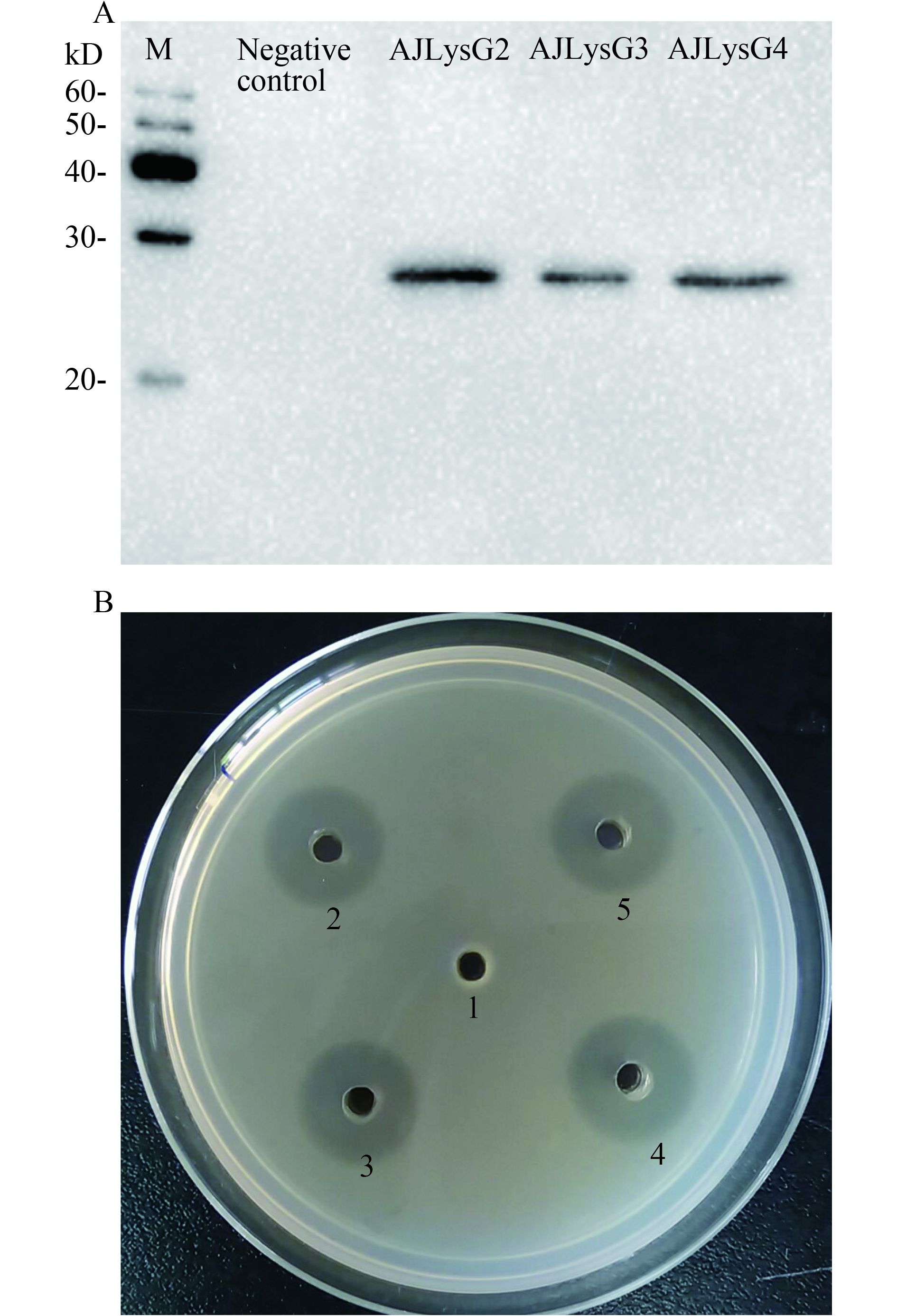
使用生物软件分析显示, AJLysG2、AJLysG3、AJLysG4蛋白分子量分别为20.7、20.4和20.7 kD; 加上真核表达载体上Myc、His标签以及多克隆位点等碱基表达的肽链, 这3个重组蛋白的分子量分别为26.3、26.0和26.3 kD, 目的条带分子量与预测的分子量基本相符(图 7)。
参考Hikima等[2]和Sun等[21]的实验, 本实验使用溶壁微球菌为底物进行溶菌酶重组蛋白对细菌生长抑制作用的检测。实验结果表明, 这3个重组蛋白对溶壁微球菌的生长均有明显的抑制作用, 抑制作用与鸡蛋白溶菌酶的作用基本一致(图 7B)。
![]() 图 7 日本鳗鲡三个G-型溶菌酶基因(AJLysG2—4)的重组表达(a)与抗菌活性检测(b)a. 重组表达溶菌酶的Western blotting检测。M是MagicMarkTM XP Western Protein Standard, Negative control为从转染pcDNATM 3.1/myc-His(-) A的EPC中提取的上清蛋白, AJLysG2、AJLysG3和AJLysG4表示从这3个溶菌酶基因重组表达细胞的上清蛋白; b. 重组表达溶菌酶对溶壁微球菌的抗菌活性。1为阴性对照, 加入了20 μL从转染pcDNATM 3.1/myc-His(-) A的EPC中提取的上清蛋白; 2为阳性对照, 加入的量为10 μg鸡蛋蛋白溶菌酶; 3—5依次为重组表达AJLysG2、AJLysG3和AJLysG4的上清蛋白, 加入量均为10 μgFigure 7. Expression (a) and bacterial inhibition (b) of recombinant proteins of three G-type lysozymes from the Japanese eela. Western blotting of recombinant proteins. M represents MagicMarkTM XP Western Protein Standard. Negative control is the supernatant extracted from EPC cells transfected with pcDNATM 3.1/myc-His (-) A, and the later three columns are recombinant proteins of AJLysG2, AJLysG3 and AJLysG4; b. Lytic activity of recombinant proteins against Micrococcus lysodeikticus. 1 negative control that was added with 20 μL supernatant extracted from EPC cells transfected with pcDNATM 3.1/myc-His(-) A; 2 positive control with the quantity of 10 μg hen egg-white lysozyme (HEWL); 3, 4, 5, recombinant proteins of AJLysG2, AJLysG3 and AJLysG4, respectively
图 7 日本鳗鲡三个G-型溶菌酶基因(AJLysG2—4)的重组表达(a)与抗菌活性检测(b)a. 重组表达溶菌酶的Western blotting检测。M是MagicMarkTM XP Western Protein Standard, Negative control为从转染pcDNATM 3.1/myc-His(-) A的EPC中提取的上清蛋白, AJLysG2、AJLysG3和AJLysG4表示从这3个溶菌酶基因重组表达细胞的上清蛋白; b. 重组表达溶菌酶对溶壁微球菌的抗菌活性。1为阴性对照, 加入了20 μL从转染pcDNATM 3.1/myc-His(-) A的EPC中提取的上清蛋白; 2为阳性对照, 加入的量为10 μg鸡蛋蛋白溶菌酶; 3—5依次为重组表达AJLysG2、AJLysG3和AJLysG4的上清蛋白, 加入量均为10 μgFigure 7. Expression (a) and bacterial inhibition (b) of recombinant proteins of three G-type lysozymes from the Japanese eela. Western blotting of recombinant proteins. M represents MagicMarkTM XP Western Protein Standard. Negative control is the supernatant extracted from EPC cells transfected with pcDNATM 3.1/myc-His (-) A, and the later three columns are recombinant proteins of AJLysG2, AJLysG3 and AJLysG4; b. Lytic activity of recombinant proteins against Micrococcus lysodeikticus. 1 negative control that was added with 20 μL supernatant extracted from EPC cells transfected with pcDNATM 3.1/myc-His(-) A; 2 positive control with the quantity of 10 μg hen egg-white lysozyme (HEWL); 3, 4, 5, recombinant proteins of AJLysG2, AJLysG3 and AJLysG4, respectively3. 讨论
多种溶菌酶基因在同一物种中同时存在的现象极其普遍, 如鸡, 除了是最早发现具有C-型溶菌酶的物种外, 还具有2个G-型溶菌酶基因[19]; 在一些哺乳类也具有类似的现象, 如人类、鼠等物种同时具有C-型和G-型溶菌酶基因[5, 35, 23]。目前研究过的大部分鱼类均同时具有C-型和G-型溶菌酶基因, 如牙鲆、草鱼、菱鲆等鱼类[2, 10, 12, 26]。经过将完整的cDNA序列在NCBI上进行Blast分析和构建进化树, 本研究成功地从日本鳗鲡克隆到了C-型和G-型两类溶菌酶基因, 其中C-型溶菌酶基因1个, G-型溶菌酶基因4个。这4个G-型溶菌酶基因的结构极为相似, 这应该是基因倍化理论一个例证。
在同一物种中也有多个C-型溶菌酶基因的报道。反刍动物中有检测到约10个C-型溶菌酶基因, 并且这些基因在胃中大量表达[36—38]; 鱼类中也有多个C-型溶菌酶基因的报道, 如罗非鱼有3个C-型溶菌酶基因[8]。然而, 本研究只在日本鳗鲡的基因组数据库中发现了一个C-型溶菌酶基因, 对其他鳗鲡种类的研究将有助于揭示鳗鲡这一类群中C-型溶菌酶基因的组成。
鸡蛋蛋白溶菌酶具有2个酶活性位点, 即Glu35和Asp52[37]。目前, 所发现的C-型溶菌酶均具有这2个酶活性位点。鹅蛋蛋白溶菌酶(Goose egg-white lysozyme, GEWL)的酶活性位点为Glu73、Asp86和Asp97[1]。Weaver等[39]通过蛋白三维结构证实GEWL的Glu73和HEWL的Glu35对应; Kawamura等[40]以鸵鸟G-型溶菌酶为材料通过点突变证明Glu73是酶活性位点。Asp86和Asp97的作用尚未得到证明, 仅有部分实验证明HEWL的Asp97可能与HEWL Asp52对应[11, 41]。目前, 鲤、鼠[11, 24]等物种和本研究所获得的AJLysG1均出现第2个酶活性位点缺失或突变的现象。若能证实这些G-型溶菌酶具有酶活性, 这对于溶菌酶的研究将有重要的促进作用。
进化分析显示, AJLysC与其他脊椎动物的C-型溶菌酶聚为一支, 所有AJLysG均和其他物种的G-型溶菌聚为一支。尽管本研究所发现的AJLysG1和另外3种AJLlysG以及其他鱼类G-型溶菌酶聚在一起, 但自成一支。在基因结构上, AJLysG3和AJLysG4的外显子数目完全相同, 但是进化树的分析显示AJLysG2和AJLysG3同源性较高, AJLysG4则与前两者的同源性较低; AJLysG1则在基因组成和蛋白序列上有差异, 这也反映在它们之间的进化关系上。
目前所发现在脊椎动物C-型溶菌酶均为4个外显子, 3个内含子结构, 基因结构很保守[14]。G-型溶菌酶的基因结构有所不同, 哺乳类和鸟类动物G-型溶菌酶有6个及更多外显子[24, 25]; 已报道的鱼类G-型溶菌酶则只有5个外显子。鸟类和哺乳类的G-型溶菌酶N-端均含有信号肽结构, 鱼类则没有, 故Irwin等[24]推测外显子的数目与G-型溶菌酶是否分泌直接相关。而在大西洋鲑[22]和大西洋鳕[23]中的发现表明, G-型溶菌酶能否分泌似乎和外显子的数目没有直接的关系。本研究所获得的AJLysG1含有6个外显子, 经预测含有信号肽。这一发现部分证实了Irwin等[24]的推测。
C-型溶菌酶的作用主要是通过裂解细胞壁的组成成分肽聚糖, 而很多动物的C-型溶菌酶在一定程度上表现出适应性进化, 还具有消化等其他的功能。在反刍动物中胃中大量表达的C-型溶菌酶基因被认为具有消化的功能[36, 37]; 鱼类, 包括虹鳟、菱鲆等物种也在胃中检测到C-型溶菌酶基因的表达。由此可见鱼类的C-型溶菌酶也可能同时具有免疫和消化的功能。这些发现可以解释日本鳗鲡AJLysC在胃中表达量最高, 同时在免疫组织中表达量也较高这一现象。G-型溶菌酶的功能主要表现在免疫方面。根据表达规律, 推测含有信号肽的AJLysG1可能是通过分泌到皮肤表面而发挥抵御外界病原入侵的作用; AJLysG2、AJLysG3和AJLysG4不具有信号肽, 应该属于胞内蛋白, 它们在不同的组织/器官表达丰度不同, 表明它们可能在不同的组织/器官具有不同的作用, 这些都有待实验的进一步证实。经病原菌刺激后, 鱼类溶菌酶基因几乎均呈上调表达。注射嗜水气单胞菌后, 草鱼C-和G-型溶菌酶基因表达量均有上调[26]; 人工感染溶藻弧菌72h后, 斜带石斑鱼胃、脾、肾等组织G-型溶菌酶基因的表达量均上调[20]。本实验也得到了类似的结果, 人工感染迟缓爱德华氏菌后, 日本鳗鲡的溶菌酶基因都呈上调表达, 然而它们的不同的上调表达的差异可能表明它们在不同的组织/器官具有不同的作用, 这有待进一步的研究。
质粒pcDNATM 3.1是一个非常完善的真核表达系统, 其人类的CMV启动子使目的蛋白在细胞中能大量表达。而在鱼类中, pcDNATM 3.1表达系统也得到广泛的应用。如刘颖等[42]曾用pcDNA3.1载体转染至鱼类细胞用于斑马鱼IFIT家族基因启动子的分析, Zou等[43]曾将重组pcDNA3.1/myc-His(-)A转染至EPC, 同样取得了良好的实验结果。本实验采用pcDNATM3.1/myc-His(-) A为表达载体同样也获得了良好的结果, 3个溶菌酶基因均能重组表达, 而且重组蛋白对溶壁微球菌的生长具有抑制作用。然而, 本研究尝试表达AJLysC和AJLysG1努力没有成功(结果未显示), 含有信号肽的AJLysC基因表达量非常低, AJLysG1的重组表达则没有检测到, 从而限制了这5种溶菌酶溶解细菌功能的比较研究, 这也有待今后的研究探讨。
-
图 1 日本鳗鲡C-型溶菌酶AJLysC和其他鱼类C-型溶菌酶的氨基酸序列比较
黑色框中为信号肽, 8个半胱氨酸用阴影表示, 黑色框与阴影共同标注的是酶活性位点。“*”表示相同的氨基酸, “:”和“.”相似的氨基酸。比对的序列分别为: SRLysC, 代表菱鲆的C-型溶菌酶, GenBank登录号为BAF75844; SSLyC, 塞内加尔鳎, ABY84356; TRLysC, 红鳍东方鲀, NP_001027914; POLysC, 牙鲆, BAB17215; CILysC, 草鱼, ACF41166; DRLysC, 斑马鱼, NP_631919
Figure 1. Comparison of deduced amino acid sequence of C-type lysozyme, AJLysC of the Japanese eel with homologues of other species of fish
Signal peptides are boxed. The 8 cysteine residues are shaded, and the catalytic residues are shaded in black boxes. (*) indicates identical amino acids, and (:) or (.) the similar amino acids. SRLysC represents C-type lysozyme in brill with the GenBank accession number being BAF75844; SSLyC, Senegalese sole, ABY84356; TRLysC, Fugu rubripes, NP_001027914; POLysC, Japanese flounder, BAB17215; CILysC, grass carp, ACF41166; DRLysC, zebrafish, NP_631919
图 2 日本鳗鲡G-型溶菌酶和其他鱼类G-型溶菌酶氨基酸序列的比较
阴影标注的是酶活性位点。“*”表示相同的氨基酸, “:”和“.”表示相似的氨基酸。日本鳗鲡的4个G-型溶菌酶AJLysG1、AJLysG2、AJLysG3、AJLysG4的GenBank登录号见表 1。比对的其他鱼类的G-型溶菌酶分别为: ECLysG, 石斑鱼的G-型溶菌酶, GenBank登录号为AAL08021; SCLysG, 鳜, AAU86896; SRLysG, 菱鲆, BAF75845; LClysG, 尖吻鲈, ABV66069; POLysG, 牙鲆, BAB17215; TRLysG, 红鳍东方鲀, NP_001027764; CCLysG, 鲤, BAA95698; DRLysG, 斑马鱼, AAI34160; CILysG, 草鱼, ACF41166
Figure 2. Comparison of deduced amino acid sequences of G-type lysozymes of the Japanese eel with homologues of other species of fish
Catalytic residues are shaded. (*) indicates identical amino acids, and (:) or (.) indicate similar amino acids. AJLysG1, AJLysG2, AJLysG3, AJLysG4 represent G-type lysozymes of Japanese eel with GenBank accession numbers indicated in Tab. 1. ECLysG represents G-type lysozyme from orange-spotted grouper with the GenBank accession number being AAL08021; SCLysG, mandarin fish, AAU86896; SRLysG, brill, BAF75845; LClysG, barramundi perch, ABV66069; POLysG, Japanese flounder, BAB17215; TRLysG, fugu rubripes, NP_001027764; CCLysG, common carp, BAA95698; DRLysG, zebrafish, AAI34160; CILysG, grass carp, ACF41166
图 4 日本鳗鲡的1个C-型(AJLysC)和4个G-型(AJLysG1-4)溶菌酶基因结构图
黑色框表示外显子, 黑色线条表示内含子。其对应的数字表示外显子或内含子的大小。黑色框和直线与数字成比例, 而折线则与数字不成比例
Figure 4. Gene organization of the C-type (AJLysC) and four G-type (AJLysG1-4) lysozymes of the Japanese eel
The black boxes represent exons, and the lines represent introns. The numbers indicate the size of exons or introns. The black boxes and the straight lines are proportional to the size of exons and introns respectively, while the concave lines are not proportional
图 7 日本鳗鲡三个G-型溶菌酶基因(AJLysG2—4)的重组表达(a)与抗菌活性检测(b)
a. 重组表达溶菌酶的Western blotting检测。M是MagicMarkTM XP Western Protein Standard, Negative control为从转染pcDNATM 3.1/myc-His(-) A的EPC中提取的上清蛋白, AJLysG2、AJLysG3和AJLysG4表示从这3个溶菌酶基因重组表达细胞的上清蛋白; b. 重组表达溶菌酶对溶壁微球菌的抗菌活性。1为阴性对照, 加入了20 μL从转染pcDNATM 3.1/myc-His(-) A的EPC中提取的上清蛋白; 2为阳性对照, 加入的量为10 μg鸡蛋蛋白溶菌酶; 3—5依次为重组表达AJLysG2、AJLysG3和AJLysG4的上清蛋白, 加入量均为10 μg
Figure 7. Expression (a) and bacterial inhibition (b) of recombinant proteins of three G-type lysozymes from the Japanese eel
a. Western blotting of recombinant proteins. M represents MagicMarkTM XP Western Protein Standard. Negative control is the supernatant extracted from EPC cells transfected with pcDNATM 3.1/myc-His (-) A, and the later three columns are recombinant proteins of AJLysG2, AJLysG3 and AJLysG4; b. Lytic activity of recombinant proteins against Micrococcus lysodeikticus. 1 negative control that was added with 20 μL supernatant extracted from EPC cells transfected with pcDNATM 3.1/myc-His(-) A; 2 positive control with the quantity of 10 μg hen egg-white lysozyme (HEWL); 3, 4, 5, recombinant proteins of AJLysG2, AJLysG3 and AJLysG4, respectively
表 1 本研究使用的引物
Table 1 Primers used in the current study
引物Primer 序列Sequence (5′-3′) 用途Usage AJLysCF-out ACCGACGGATCGACGGACTA 扩增开放阅读框 AJLysCF-in AGTCGCTGGTGGTGTAACGA AJLysCR-out AGTGGTTCCGCCATGCCAC AJLysCR-in TGGGATCTCTGACCACACGC AJLysG1F-out CACAAAGACCGAATCAACAGGG AJLysG1F-in GTTCCATTTCCCGTGAGTCCAG AJLysG1R-out CAGAAGTTCAGTGCCTTGACAGTT AJLysG1R-in TGCTGCTCGCTATTCCACTGTCC AJLysG2F-out GTGGGACGGGCTCAGCAAAT AJLysG2F-in CAAGCGATACCACACGCCAAG AJLysG2R-in TGGCGTGTGGTATCGCTTGTC AJLysG3F-out GCATCCCACAAGTTGGCACAG AJLysG3F-in CGGAGTTCTTACAAACGGCTGG AJLysG3R-out TTCTTGTTGAACCACTGGGCTC AJLysG3R-in TGTCCATTCGCTCATAAGTCCG AJLysG4F-out TCACAACAAACCGCTAATCAGGA AJLysG4F-in CTGTGGTCGCCGCTATCATTTC AJLysG4R-out CAACATCATTGGAGTAGTCACGCC AJLysG4R-in ATTGTAGGCTGCTATTCCACCCAT CF AATCATCTGTCACTTCAGAATG 扩增全基因 CR GAAAAGGTGCTTCTCTCTGG G1F TTGCTAGAAAATTACTAAAAGTAGAA G1R GATTGCATTTCACCCTGTGT G2F CCACAGGGAAGAATAACAAC G2R TCCAAGGCTTATTTTTCTCT G3F AGCCAGAAGACAGAACATTAC G3R TTACAAGGTTTTCATCATATTTT G4F CTGAAGAGTTTGTGGAAGTTC G4R AAGATTTCATTCACTCCTTTTA ActinF CCCAAGACATCAGGGCGTGA 定量分析 ActinR CTCAAACATGATCTGCGTCATC CQF CCTGGGAGACTGGGTCTG CQR CCACCCATGCTCTGATCC G1QF TGGAATAGCGAGCAGCACAACT G1QR CCACGGTCAGAAGCCAGATTGA G2QF GGACTCATGCAGATTGACAAGC G2QR TATCCCACCTTTAAATTGGTGCT G3QF GGACTCATGCAGGTTGACAA G3QR GATATTCCTCCTTTTAATTGTTGC G4QF GGCTTATGCAGGTTGACAAG G4QR CTGCTATTCCACCTTTAAGTTGT EUAJLysG2F CGGAATTCGCCACCATGGCAATGACCTGCATTTATGGGGA 构建表达载体 EUAJLysG2R GCGGATCC AAAGCCATTCCGCTTGAACC EUAJLysG3F GCGGATCCGCCACCATGGACGGCATTTATGGCGA EUAJLysG3R CCAAGCTTAAAACCATTCTTGTTGAACCACTGG EUAJLysG4F GCGGATCCGCCACCATGGCCTGCATTTACGGCGA EUAJLysG4R CCAAGCTTAAAACCATTCCTTTTGTAGAACTGG 表 2 日本鳗鲡溶菌酶基因cDNA序列及其氨基酸序列
Table 2 The cDNA sequences and deduced amino acids of lysozyme genes in the Japanese eel
基因名称Gene name 登录号GenBank Acc. No. 总长度Length (bp) 5′端非编码区5′ UTR* 3′端非编码区3′ URT* 氨基酸数目No. of aa 成熟肽氨基酸数目No. of aa in mature peptide 等电点pI C-type AJLysC KM454476 811 83 296 143 128 8.48 G-type AJLysG1 KM454477 749 90 77 193 174 9.42 AJLysG2 KM454478 1352 35 759 185 185 8.89 AJLysG3 KM454479 1175 82 529 185 185 9.00 AJLysG4 KM454480 733 56 113 187 187 7.05 注: * UTR表示untranslated region 表 3 日本鳗鲡G-型溶菌酶之间以及与其他鱼类G-型溶菌酶的同一性分析
Table 3 Comparison among G-type lysozymes of the Japanese eel and other species of fish
鱼类的G-型溶菌酶G-type lysozymes in fish 日本鳗鲡G-型溶菌酶G-type lysozymes in Japanese eel AJLysG1 AJLysG2 AJLysG3 AJLysG4 日本鳗鲡AJLysG1 38.9 41.8 34.5 日本鳗鲡AJLysG2 38.9 68.4 58.8 日本鳗鲡AJLysG3 41.8 68.4 63.6 日本鳗鲡AJLysG4 34.5 58.8 63.6 草鱼Grass carp 36.9 76.2 65.8 59.4 鲤Common carp 34.5 65.4 64.7 54.5 斑马鱼Zebrafish 37.9 73 68.4 57.2 红鳍东方鲀Fugu rubripes 31.1 54.7 49.5 49.5 尖吻鲈Barramundi perch 31.5 57.1 59.7 59.2 牙鲆Japanese flounder 34.1 52.8 53.3 51.8 菱鲆Brill 30.2 56.4 55.3 53.8 斜带石斑鱼Orange-spotted grouper 30 55.1 52.5 52 鳜Mandarin fish 29.7 55.1 54.5 53.5 注: AJLysG1、AJLysG2、AJLysG3、AJLysG4为日本鳗鲡的4个G-型溶菌酶, 其GenBank序列号见表 2。草鱼grass carp Ctenopharyngodon idella: 代表草鱼G-型溶菌酶, 其GenBank登录号为ACF41166; 鲤common carp Cyprinus carpio: BAA95698; 斑马鱼zebrafish Danio rerio: AAI34160; 红鳍东方鲀Fugu rubripes Takifugu rubripes: NP_001027764; 尖吻鲈barramundi perch Lates calcarifer: ABV66069; 牙鲆Japanese flounder Paralichthys olivaceus: BAB17215; 菱鲆brill Scophthalmus rhombus: BAF75845; 斜带石斑鱼orange-spotted grouper Epinephelus coioides: AAL08021; 鳜mandarin fish Siniperca chuatsi: AAU86896 Note: AJLysG1, AJLysG2, AJLysG3 and AJLysG4 are four G-type lysozymes in the Japanese eel with their GenBank accession numbers indicated in Tab. 2 -
[1] Jollés P, Jollés J. What’s new in lysozyme research? Always a model system, today as yesterday [J]. Molecular and Cellular Biochemistry, 1984, 63(2): 165—189
[2] Hikima Jun-ichi, Minagawa S, Hirono I, et al. Molecular cloning, expression and evolution of the Japanese flounder goose-type lysozyme gene, and the lytic activity of its recombinant protein [J]. Biochimica et Biophysica Acta - Gene Structure and Expression, 2001, 1520(1): 35—44 doi: 10.1016/S0167-4781(01)00248-2
[3] Thammasirirak S, Torikata T, Takami K, et al. The primary structure of cassowary (Casuarius casuarius) goose type lysozyme [J]. Bioscience Biotechnology and Biochemistry, 2002, 66(1): 147—156 doi: 10.1271/bbb.66.147
[4] Canfield R E. Amino acid sequence of egg white lysozyme [J]. The Journal of Biological Chemistry, 1963, 238(8): 2698—2707
[5] Peters C W, Kruse U, Pollwein R, et al. The human lysozyme gene. Sequence organization and chromosomal localization [J]. European Journal of Biochemistry, 1989, 182(3): 507—516 doi: 10.1111/ejb.1989.182.issue-3
[6] Callewaert L, Michiels C W. Lysozymes in the animal kingdom [J]. Journal of Bioscience and Bioengineering, 2010, 35(1): 127—160
[7] Liu F, Wen Z. Cloning and expression pattern of the lysozyme C gene in zebrafish [J]. Mechanisms of Development, 2002, 113(1): 69—72 doi: 10.1016/S0925-4773(01)00658-X
[8] 禹绍国, 叶星, 张莉莉, 等. 奥利亚罗非鱼3种C型溶菌酶基因的克隆和序列分析. 农业生物技术学报, 2010, 18(1): 66—74 http://www.cnki.com.cn/Article/CJFDTOTAL-NYSB201001013.htm Yu S G, Ye X, Zhang L L, et al. Molecular cloning and sequencing of three C-type lysozyme genes from Oreochromis aureus [J]. Journal of Agricultural Biotechnology, 2010, 18(1): 66—74
禹绍国, 叶星, 张莉莉, 等. 奥利亚罗非鱼3种C型溶菌酶基因的克隆和序列分析. 农业生物技术学报, 2010, 18(1): 66—74 http://www.cnki.com.cn/Article/CJFDTOTAL-NYSB201001013.htm[9] Dautigny A, Prager E M, Phamdinh D, et al. CDNA and amino-acid-sequences of rainbow trout (Oncorhynchus mykiss) lysozymes and their implications for the evolution of lysozyme and lactalbumin [J]. Journal of Molecular Evolution, 1991, 32(2): 187—198 doi: 10.1007/BF02515392
[10] Hikima J, Hirono I, Aoki T. Molecular cloning and novel repeated sequences of a C-type lysozyme gene in Japanese flounder (Paralichthys olivaceus) [J]. Marine biotechnology, 2000, 2(3): 241—247 doi: 10.1007/s101269900028
[11] Fujiki K, Shin D H, Nakao M, et al. Molecular cloning of carp (Cyprinus carpio) leucocyte cell-derived chemotaxin 2, glia maturation factor β, CD45 and lysozyme C by use of suppression subtractive hybridization [J]. Fish and Shellfish Immunology, 2000, 10(7): 643—650 doi: 10.1006/fsim.2000.0294
[12] Jiménez-Cantizano R M, Infante C, Martin-Antonio B, et al. Molecular characterization, phylogeny, and expression of c-type and g-type lysozymes in brill (Scophthalmus rhombus) [J]. Fish and Shellfish Immunology, 2008, 25(1—2): 57—65 doi: 10.1016/j.fsi.2007.12.009
[13] Ye X, Gao F Y, Zheng Q M. Cloning and characterization of the tiger shrimp lysozyme [J]. Molecular biology reports, 2008, 36(6): 1239—1246
[14] Liu M, Zhang S, Liu Z, Li H, et al. Characterization, organization and expression of AmphiLysC, an acidic c-type lysozyme gene in amphioxus Branchiostoma belcheri tsingtauense [J]. Gene, 2006, 367(15): 110—117
[15] Canfield R E, McMurry S. Purification and characterization of a lysozyme from goose egg white [J]. Biochemical and Biophysical Research Communications, 1967, 26(1): 38—42 doi: 10.1016/0006-291X(67)90249-5
[16] Schoentgen F, Jolles J, Jolles P. Complete amino acid sequence of ostrich (Struthio camelus) egg-white lysozyme, a goose-type lysozyme [J]. European Journal of Biochemistry, 1982, 123(3): 489—497
[17] Thammasirirak S, Torikata T, Takami K, et al. The primary structure of cassowary (Casuarius casuarius) goose type lysozyme [J]. Bioscience, Biotechnology, and Biochemistry, 2002, 66(1): 147—156 doi: 10.1271/bbb.66.147
[18] Pooart J, Torikata T, Araki T. The primary structure of a novel goose-type lysozyme from rhea egg white [J]. Bioscience, Biotechnology, and Biochemistry, 2004, 68(1): 159—169 doi: 10.1271/bbb.68.159
[19] Nile C J, Townes C L, Michailidis G, et al. Identification of chicken lysozyme g2 and its expression in the intestine [J]. Cellular and Molecular Life Sciences, 2004, 61(21): 2760—2766 doi: 10.1007/s00018-004-4345-z
[20] Yin Z X, He J G, Deng W X, et al. Molecular cloning, expression of orange spotted grouper goose-type lysozyme cDNA, and lytic activity of its recombinant protein [J]. Diseases of Aquatic Organisms, 2003, 55(2): 117—123
[21] Sun B J, Wang G L, Xie H X, et al. Gene structure of goose-type lysozyme in the mandarin fish Siniperca chuatsi with analysis on the lytic activity of its recombinant in Escherichia coli [J]. Aquaculture, 2006, 252(2—4): 106—113 doi: 10.1016/j.aquaculture.2005.07.046
[22] Kyomuhendo P, Myrnes B, Nilsen I W. A cold-active salmon goose-type lysozyme with high heat tolerance [J]. Cellular and Molecular Life Sciences, 2007, 64(21): 2841—2847 doi: 10.1007/s00018-007-7372-8
[23] Larsen A N, Solstad T, Svineng G, et al. Molecular characterisation of a goose-type lysozyme gene in Atlantic cod (Gadus morhua L.) [J]. Fish and Shellfish Immunology, 2009, 26(1): 122—132 doi: 10.1016/j.fsi.2008.03.021
[24] Irwin D M, Gong Z. Molecular evolution of vertebrate goose-type lysozyme genes [J]. Journal of Molecular Evolution, 2003, 56(2): 234—242 doi: 10.1007/s00239-002-2396-z
[25] Nakano T, Graf T. Goose-type lysozyme gene of the chicken: sequence, genomic organization and expression reveals major differences to chicken-type lysozyme gene [J]. Biochimica et Biophysica Acta - Gene Structure and Expression, 1991, 1090(2): 273—276 doi: 10.1016/0167-4781(91)90118-6
[26] Ye X, Zhang L L, Tian Y Y, et al. Identification and expression analysis of the g-type and c-type lysozymes in grass carp Ctenopharyngodon idellus [J]. Developmental and Comparative Immunology, 2010, 34(5): 501—509 doi: 10.1016/j.dci.2009.12.009
[27] 胡园, 周朝生, 胡利华, 等. 海、淡水养殖日本鳗鲡肌肉和鱼皮营养分析比较. 水生生物学报, 2015, 39(4): 730-739 doi: 10.7541/2015.96 Hu Y, Zhou C S, Hu L H, et al. Comparative analysis of the nutritional composition in the muscles and skins of Anguilla japonica cultured in the seawater and freshwater [J]. Acta Hydrobiologica Sinica, 2015, 39(4): 730-739
胡园, 周朝生, 胡利华, 等. 海、淡水养殖日本鳗鲡肌肉和鱼皮营养分析比较. 水生生物学报, 2015, 39(4): 730-739 doi: 10.7541/2015.96[28] 于海振, 李秋云, 张坤. 鳗鲡的营养成分·养殖现状与对策. 安徽农业科学, 2014, 42(20): 6632—6633, 6664 doi: 10.3969/j.issn.0517-6611.2014.20.041 Yu H Z, Li Q Y, Zhang K. Nutrition of eels, breeding status and the countermeasures [J]. Journal of Anhui Agriculture Science, 2014, 42(20): 6632—6633, 6664
于海振, 李秋云, 张坤. 鳗鲡的营养成分·养殖现状与对策. 安徽农业科学, 2014, 42(20): 6632—6633, 6664 doi: 10.3969/j.issn.0517-6611.2014.20.041[29] The IUCN Red List of Threatened Species [DB/OL]. Version 2014.3. Available at: www.iucnredlist.org/details/ 166184/0
[30] The IUCN Red List of Threatened Species [DB/OL]. Version 2014.3. Available at: www.iucnredlist.org/details/ 60344/0
[31] Campanella J J, Bitincka L, Smalley J. MatGAT: an application that generates similarity/identity matrices using protein or DNA sequences [J]. BMC Bioinformatics, 2003, 4: 29 doi: 10.1186/1471-2105-4-29
[32] Qi Z T, Nie P. Comparative study and expression analysis of the interferon gamma gene locus cytokines in Xenopus tropicalis [J]. Immunogenetics, 2008, 60(11): 699—710 doi: 10.1007/s00251-008-0326-y
[33] Schmittgen D T, Livak J K. Analyzing real-time PCR data by the comparative CT method [J]. Nature Protocols, 2008, 3(6): 1101—1108 doi: 10.1038/nprot.2008.73
[34] Bustin S A, Benes V, Garson A J, et al. The MIQE Guidelines: Minimum information for publication of quantitative real-time PCR experiments [J]. Clinical Chemistry, 2009, 55(4): 1—12
[35] Yeh T C, Wilson A C, Irwin D M. Evolution of rodent lysozymes: isolation and sequence of the rat lysozyme genes [J]. Molecular Phylogenetics and Evolution, 1993, 2(1): 65—75 doi: 10.1006/mpev.1993.1007
[36] Irwin M D. Evolution of the bovine lysozyme gene gamily: Changes in gene expression and reversion of function [J]. Journal of Molecular Evolution, 1995, 41(3): 299—312 doi: 10.1007/BF01215177
[37] Irwin M D. Evolution of cow nonstomach lysozyme genes [J]. Genome, 2004, 47(6): 1082—1090 doi: 10.1139/g04-075
[38] Cheetham J C, Artymiuk P J, Phillips D C. Refinement of an enzyme complex with inhibitor bound at partial occupancy [J]. Journal of Molecular Biology, 1992, 224(3): 613—628 doi: 10.1016/0022-2836(92)90548-X
[39] Kawamura S, Ohno K, Ohkuma M, et al. Experimental verification of the crucial roles of Glu73 in the catalytic activity and structural stability of goose type lysozyme [J]. Journal of Biochemistry, 2006, 140(1): 75—85 doi: 10.1093/jb/mvj125
[40] Hirakawa H, Ochi A, Kawahara Y, et al. Catalytic reaction mechanism of goose egg white lysozyme by molecular modelling of enzyme-substrate complex [J]. Journal of Biochemistry, 2008, 144(6): 753—761 doi: 10.1093/jb/mvn133
[41] Helland R, Larsen R L, Finstad S, et al. Crystal structures of g-type lysozyme from Atlantic cod shed new light on substrate binding and the catalytic mechanism [J]. Cellular and Molecular Life Sciences, 2009, 66(15): 2585—2598 doi: 10.1007/s00018-009-0063-x
[42] 刘颖, 张义兵, 刘庭凯, 等. 斑马鱼一个IFIT 家族基因的鉴定及启动子分析. 水生生物学报, 2012, 36(1): 1—8 http://ssswxb.ihb.ac.cn/CN/abstract/abstract2040.shtml Liu Y, Zhang Y B, Liu K T, et al. Identification and promoter analysis of an IFIT family gene from zebrafish Danio rerio [J]. Acta Hydrobiologica Sinica, 2012, 36(1): 1—8
刘颖, 张义兵, 刘庭凯, 等. 斑马鱼一个IFIT 家族基因的鉴定及启动子分析. 水生生物学报, 2012, 36(1): 1—8 http://ssswxb.ihb.ac.cn/CN/abstract/abstract2040.shtml[43] Zou P F, Chang M X, Xue N N, et al. Melanoma differentiation-associated gene 5 in zebrafish provoking higher interferon-promoter activity through signalling enhancing of its shorter splicing variant [J]. Immunology, 2013, 141(2): 192—202
-
期刊类型引用(3)
1. 王运娇,吕梦,蔡莎莎. 扎那纹胸鮡g型溶菌酶的cDNA克隆及组织表达分析. 安徽医科大学学报. 2023(05): 711-717 .  百度学术
百度学术
2. 陈华,林晨韬,陈曦,葛均青. 斑马鱼g型溶菌酶基因序列分析及其原核表达. 南方农业学报. 2022(01): 229-237 .  百度学术
百度学术
3. 贾慧霞,张华,张成龙,何毛贤,刘文广. 法螺G型溶菌酶重组蛋白的原核表达及抑菌活性探究. 海洋科学. 2022(10): 32-42 .  百度学术
百度学术
其他类型引用(1)



 下载:
下载: