MOLECULAR CLONING AND EXPRESSION ANALYSIS OF THE α AND γ SUBUNIT GENES OF FcγR IN RAINBOW TROUT (Oncorhynchus mykiss)
-
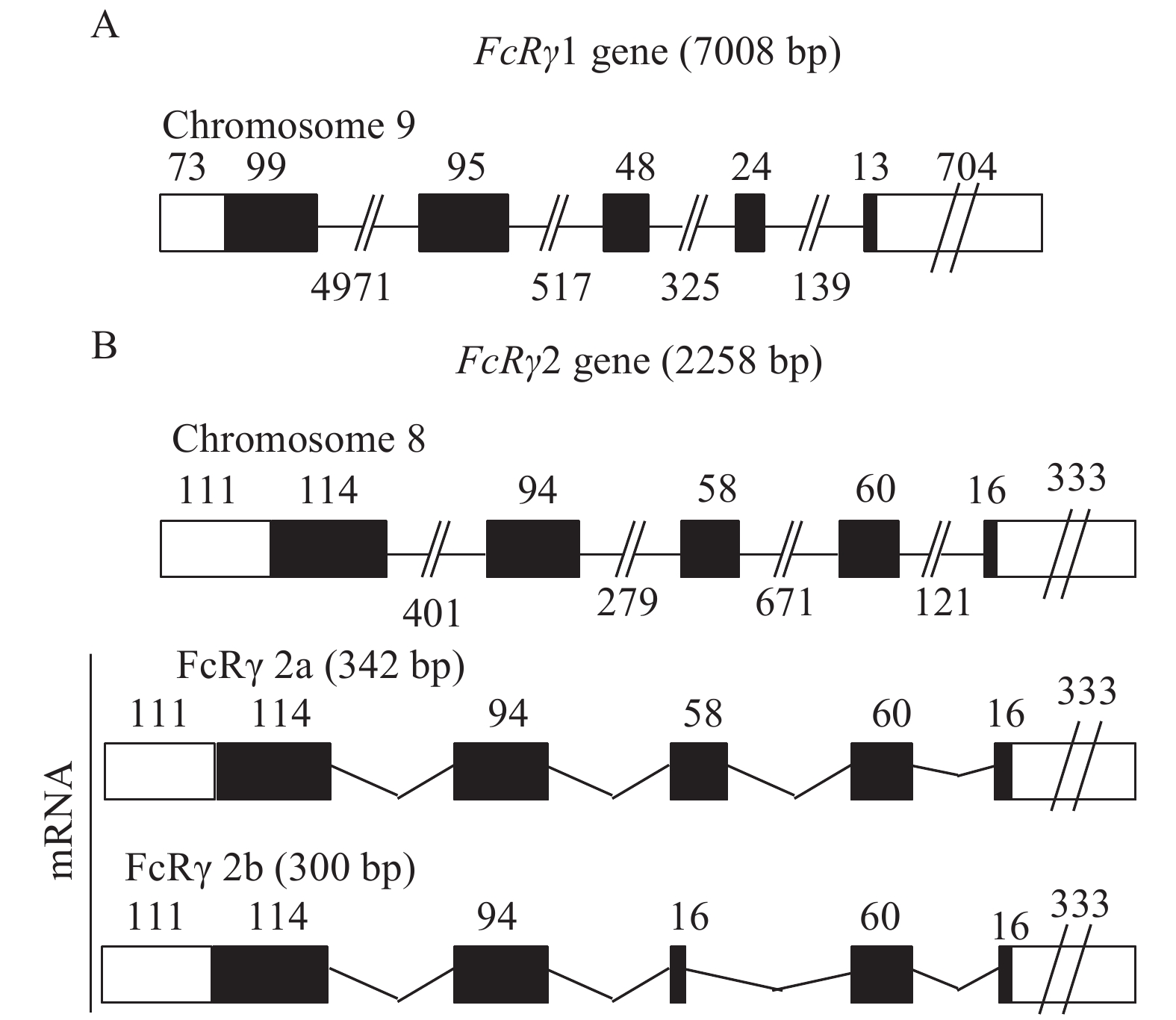
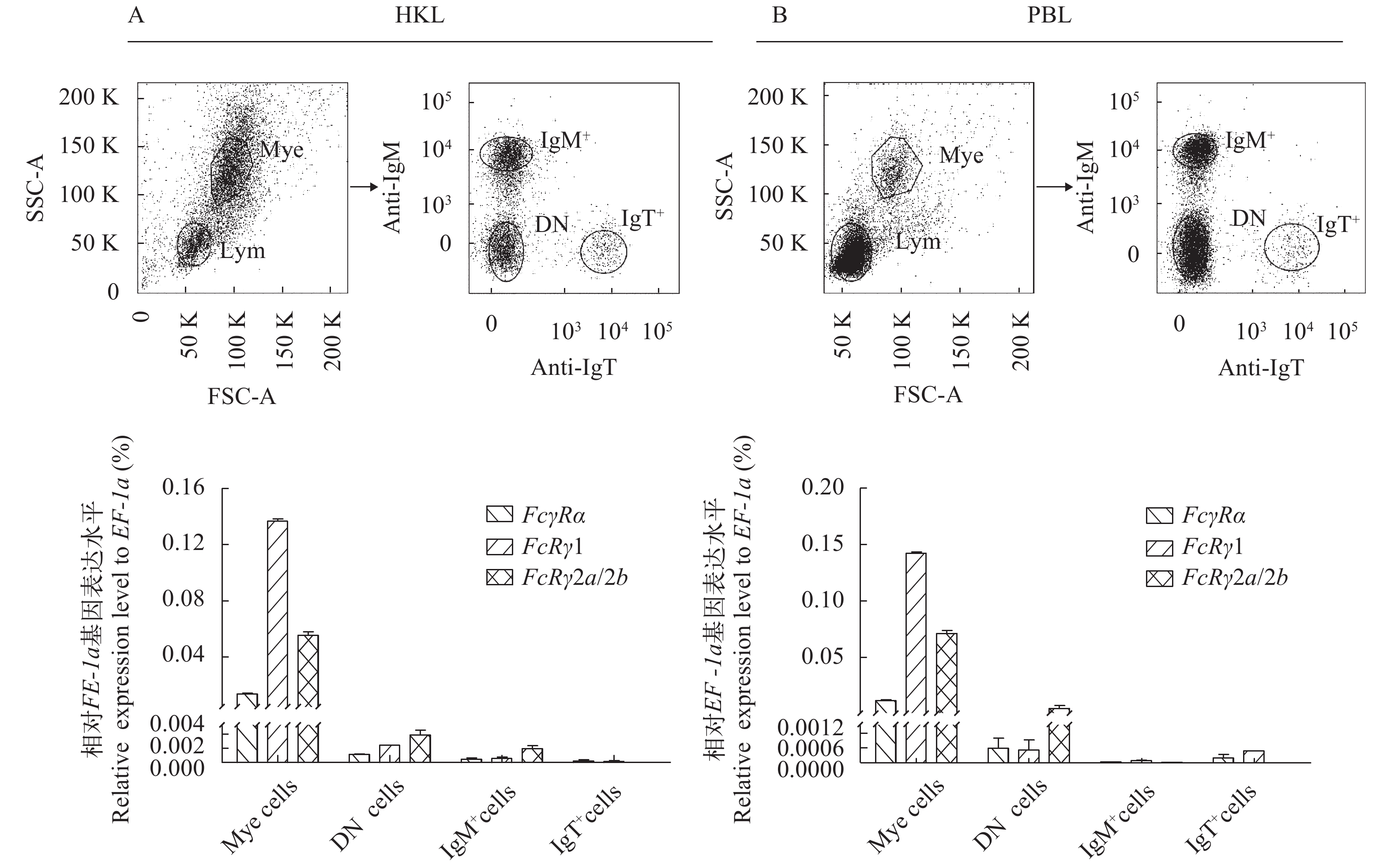
摘要: Fc受体(FcR)是一种表达在免疫细胞表面的受体分子, 由多亚基构成, 通过与免疫球蛋白(Ig)的Fc段结合引起包括炎症因子释放和吞噬作用等体液和细胞免疫反应。研究采用RACE技术首次克隆得到了虹鳟FcγR的α亚基基因(FcγRα)和γ亚基基因(FcRγ)的cDNA序列, 采用生物信息学软件对FcγRα和FcRγ的序列进行了特征分析, 实时荧光定量PCR检测了其在不同组织和细胞亚群中以及在Poly (I鲶C)和LPS刺激后头肾中的表达。结果显示:FcγRα的cDNA全长1677 bp, 开放阅读框为954 bp, 编码317个氨基酸; FcγRα由信号肽和2个Ig样结构域构成, 但没有跨膜区和胞内区。FcRγ亚基存在2种形式, 分别命名为FcRγ1和FcRγ2(包含FcRγ2a和FcRγ2b两个剪接异构体), 它们均由信号肽、跨膜区和胞内的免疫受体酪氨酸活化基序(ITAM)构成。氨基酸序列相似性分析表明虹鳟FcγRα与斑点叉尾鮰FcRI相同率最高(30%), 虹鳟FcRγ1和FcRγ2a/2b与哺乳动物FcRγ相同率最高可达40%。组织表达显示FcγRα、FcRγ1和FcRγ2a/2b在头肾、脾脏和血液中表达较高; 细胞亚群表达显示FcγRα、FcRγ1和FcRγ2a/2b在髓样细胞群中表达最高; LPS和Poly (I鲶C)刺激后,FcγRα、FcRγ1和FcRγ2a/2b在头肾中的表达显著上调, 这表明FcγR在机体抗细菌和抗病毒免疫中可能发挥重要作用。Abstract: In this study, the cDNA sequences of FcγRα and FcRγ subunits of FcγR rainbow trout were cloned for the first time by RACE technique. The sequences of FcγRα and FcRγ were analyzed by using bioinformatics software. The expressions of the genes in different cell subpopulations and tissues, as well as in the head kidney after Poly (I鲶C) and lipopolysaccharide (LPS) stimulations were analyzed by real-time fluorescent quantitative polymerase chain reaction (PCR). The results showed that the full-length cDNA ofFcγRα is 1677 bp with an open reading frame (ORF) of 954 bp encoding 317 amino acids. The FcγRα is composed of a signal peptide and two Ig-like domains, however, without including transmembrane and intracellular regions. There are three kinds of FcγR subunit, involving FcRγ1, FcRγ2a, and FcRγ2b. FcRγ1 and FcRγ2 genes are located on different chromosomes, while FcRγ2a and FcRγ2b are two splicing isoforms of FcRγ2 gene. These subunits are composed of signal peptide, transmembrane domain, and immunoreceptor tyrosine-based activation motif (ITAM). Amino acid sequence similarity analysis showed that the highest identity (30%) of FcγRα is between rainbow trout and channel catfish (Ictalurus punctatus), and FcRγ1 and FcRγ2a/2b in rainbow trout have the maximum sequence identity (40%) with mammalian FcRγ. Tissue distribution analysis showed that the expression of FcγRα, FcRγ1, and FcRγ2a/2b was higher in head kidney, spleen, and blood cells, respectively. Analysis of cell subpopulations showed that the expression of FcγRα, FcRγ1, and FcRγ2a/2b was the highest in the myeloid cell population. In addition, the expression of FcγRα, FcRγ1, and FcRγ2a/2b in the head kidney was significantly up-regulated after LPS and Poly (I鲶C) stimulations, indicating that FcγR plays an important role in the antibacterial and antiviral immunity.
-
Keywords:
- FcR /
- FcγRα /
- FcRγ /
- Rainbow trout /
- Molecular cloning /
- Gene expression
-
微藻在水产养殖中的作用越来越重要, 不但可以作为饵料生物, 提高水产动物幼体的成活率[1]; 而且可以调节水质, 消除氮磷, 抑制病害发生[2]。温度、盐度、光照是影响海洋微藻生长和代谢的重要生态因子, 直接影响细胞内的光合电子传递、代谢途径调控、特异性酶反应和细胞的通透性等, 是影响藻细胞生长、油脂积累和脂肪酸组成的重要因素。大量研究表明改变微藻的培养条件和培养周期, 不但会影响微藻生长速率, 还会影响海洋微藻的油脂含量以及脂肪酸组成, 特别是PUFA、EPA和DHA的含量, 而且两者往往不同步, 所以很多学者提出“分级培养”或“二次培养”[3—6]。
STR01是一种扭鞘藻(Streptotheca sp.), 是本实验室从象山港海区新分离的一株生长快、不易老化和污染、耐高温的浮游硅藻。该藻隶属于硅藻门、中心纲、盒形硅藻目、真弯藻科、扭鞘藻属。该藻壳面呈椭圆形, 壳环面长方形, 壳长22—38 μm, 壳宽12—19 μm, 色素体黄褐色片状。初步试验表明是一种理想的贝类育苗饵料。但有关该藻的生态习性及培养方法迄今为止未见报道。为此, 本文通过单因子和正交试验进行了温度、盐度、光照强度对该藻的生长、总脂和脂肪酸组成影响的研究, 以期为扭鞘藻的进一步开发利用提供理论依据。
1. 材料与方法
1.1 藻种及培养
STR01藻种保藏于宁波大学饵料生物培养室, 实验药品均为分析纯; 培养用水为象山港海域天然海水, 经沙滤、暗沉淀、脱脂棉过滤、烧开, 凉冷后使用; 培养液采用宁波大学#3母液配方[4]。藻种置于GXZ智能型光照培养箱(宁波江南仪器厂)中活化和扩大, 培养静置不充气, 每天定时摇藻 2 次, 早晚各1次。培养条件: 温度 (25±1)℃, 光照 60 μmol/(m2·s), 光暗周期(L﹕D) 12﹕12。
1.2 实验方法
温度试验 设置温度梯度10、15、20、25、30和35℃, 培养容器为消毒的1000 mL三角烧瓶, 各6平行。每瓶加培养液900 mL(宁波大学3#培养液配方), 接种100 mL, 其他培养条件: 光照强度60 μmol/(m2·s), 光周期(L﹕D) 12﹕12, 盐度25, pH 8.10, 所有试验瓶均放在GXZ智能型光照培养箱, 培养时间7d。接种后计数N0, 7d后计数Nt, 采用离心机(Centrifuge 5430 R)藻液离心(6000 r/min)收集藻, 存放并冷冻干燥(–80℃, FD5-3T, Gold-SIM), 测定总脂和脂肪酸。
光照强度试验 设置光照强度梯度40、60、80、100和120 μmol/(m2·s), 各6平行。培养条件: 温度25℃, 光周期(L﹕D) 12﹕12, 盐度25, pH 8.1, 其他条件同上。
盐度试验 盐度梯度设置为10、15、20、25、30和35, 各6平行。培养条件: 光照强度60 μmol/(m2·s), 光周期(L﹕D) 12﹕12, 温度25℃, pH 8.1。其他条件同上。
温、光、盐正交试验 本试验采用温度(A)、光照强度(B)和盐度(C)三因素三水平正交试验(表 1), 其他培养条件: 光周期(L﹕D) 12﹕12, pH 8.1, 其他条件同上。
表 1 正交试验参数因数水平表Table 1. The orthogonal test parameter-factor level水平
Level因素
FactorA B C 1 20 40 20 2 25 60 25 3 30 80 30 1.3 总脂含量及脂肪酸的测定
总脂的抽取及测定 采用改良Bligh-Dyer方法[7, 8]。取一定体积藻液, 6000 r/min离心10min, 再用消毒淡水清洗2—3次, 冷冻干燥保存(FD5-3T, Gold-SIM)。将干燥的藻泥用研钵研碎后, 采用氯仿﹕甲醇提取法, 得出总脂含量。
脂肪酸组成分析 采用KOH甲醇水法[9, 10]。总脂用5%—6%KOH-甲醇﹕水(体积比为4﹕1)皂化2h(60℃水浴), 2 mL氯仿: 正己烷(体积比1﹕4)提取; 后甲酯化, 旋转蒸发蒸用正己烷(色谱纯)到离心管中离心, 取上清液, 再进行GL-MS分析。用6890N/5973气相色谱-质谱联用仪按照面积归一化法计算各组分的相对百分含量。
1.4 统计与数据处理
平均相对生长速率计算公式为: K=(ln Nt-ln N0) /t(式中, N0为初始细胞数目、Nt为培养7d后的细胞数、t为培养时间)。得出的实验数据用平均值(Mean)±标准差(SD)表示, 单因素方差分析(ANOVA)和多元方差分析(MANOVA)以及Duncan多重比较分析(α=0.05)均用SPSS 17.0 统计软件, 用2007版Excel进行图表制作。
2. 结果
2.1 温度对STR01的生长、总脂含量及脂肪酸的影响
温度对STR01生长和总脂含量的影响 不同温度对STR01的平均相对生长速率影响显著(P<0.05)。该藻在15—35℃内, 随着温度升高平均相对生长速率呈先上升后降低趋势, 在30℃时该藻的平均相对生长速率最大(K值为0.682), 但25℃组与30℃组平均相对生长速率差异不显著(P>0.05), 温度在10℃条件下, 培养7d大部分藻死亡分解, 藻液颜色变白。所以该藻适合的温度是15—35℃, 最适温度为25—30℃(图 1)。
在温度10—35℃内, 温度10℃时, 由于藻体死亡分解, 无法收集, 量不足无法完成总脂含量的测定。在15—35℃的温度下, 温度对该藻的总脂含量有影响显著(P<0.05), 随着温度升高, 该藻总脂含量先增加后减少, 其中25℃时总脂含量最高(17.23%), 且显著高于其他组(P<0.05)(表 2)。
表 2 温度对STR01的脂肪酸组成和含量的影响(%)Table 2. Effects of temperature on fatty acid composition and content in STR01 (%)脂肪酸
Fatty acid温度
Light intensity (℃)15 20 25 30 35 C14﹕1 2.51±0.02a 1.29±0.01c 1.11±0.03d 1.43±0.02b 2.40±0.01a C14﹕0 11.70±0.11c 11.80±0.19c 14.40±0.23a 12.85±0.09b 8.94±0.19d C15﹕0 1.20±0.25b 1.51±0.17a 0.53±0.05c 1.01±0.07b 0.23±0.06d C16﹕1 8.66±0.22c 9.51±0.25b 10.60±0.26a 10.20±0.16a 5.44±0.18d C16﹕0 31.20±0.20b 25.00±0.17c 25.60±0.13bc 24.90±0.18d 32.60±0.28a C18﹕2 (n–6) 1.12±0.21b 0.80±0.25c 1.10±0.17b 1.10±0.12b 1.35±0.13a C18﹕3 (n–3) 3.75±0.32e 5.03±0.14c 7.25±0.24a 6.23±0.16b 4.10±0.05d C18﹕0 24.70±0.10b 16.80±0.31d 15.30±0.23e 20.40±0.13c 31.20±0.42a C20﹕5 (n–3) 0.11±0.02d 0.41±0.20c 0.44±0.09c 0.52±0.09b 1.11±0.29a C20﹕4 (n–6) 10.47±0.12d 24.80±0.34a 20.10±0.26b 16.32±0.28c 9.59±0.17e C20﹕2 1.17±0.08b 0.21±0.06d 0.66±0.14c 2.40±0.36a 1.32±0.26b C22﹕6 (n–3) 1.87±0.09b 2.16±0.17a 2.12±0.23a 1.76±0.23b 0.07±0.02c C22﹕2 1.54±0.41a 0.82±0.22c 0.83±0.16c 0.92±0.06b 1.47±0.21a SFA 68.80±0.31b 55.11±0.20d 55.83±0.33d 59.16±0.07c 72.97±0.17a MUFA 11.17±0.16b 10.80±0.12c 11.71±0.30a 11.63±0.32a 7.84±0.24d PUFA 20.03±0.11d 34.23±0.23a 32.50±0.35b 29.52±0.16c 19.01±0.08e n–3 PUFA 5.73±0.23d 7.60±0.04c 9.81±0.17a 8.51±0.26b 5.28±0.05e n–6 PUFA 11.59±0.29d 25.60±0.09a 21.20±0.34b 17.42±0.30c 10.94±0.13e 温度对STR01脂肪酸组成和含量的影响 本试验共测定出该藻含13种主要脂肪酸, 其中饱和脂肪酸(SFA) 4种, 单不饱和脂肪酸(MUFA) 2种, 多不饱和脂肪酸(PUFA) 7种。主要脂肪酸为C14﹕0、C16﹕1、 C16﹕0、 C18﹕0和C20﹕4 (n–6)。试验结果表明, 温度对该藻PUFA、EPA含量影响显著(P<0.05), 在温度15—30℃内, 该藻PUFA的含量随着温度的升高呈现先上升后下降的趋势, PUFA的含量为19.01%—34.23%, 30℃时PUFA的含量最高(34.23%)。该藻的EPA和DHA含量极少, 在15—35℃条件下EPA含量为0.11%—1.11%, DHA含量为0.07%—2.16%。
2.2 光照强度对STR01的生长、总脂含量及脂肪酸的影响
光照强度对STR01生长和总脂含量的影响 不同的光照强度对STR01的平均相对生长速率影响显著(P<0.05)。该藻在40—120 μmol/(m2·s), 随着光照强度的升高生长速率呈先上升后降低趋势, 60 μmol/(m2·s)时平均相对生长速率最大(K值0.630), 且显著高于其他组。该藻生长的适合的光照强度是40—120 μmol/(m2·s), 最适光强为60 μmol/(m2·s)。
光照强度在60 μmol/(m2·s)时该藻的总脂含量显著高于较其他组(P<0.05), 此时含量最高(16.73%)。总脂含量按照从高到低的排序60 μmol/(m2·s)(16.73%)>40 μmol/(m2·s)(13.07%)≥100 μmol/(m2·s)(9.37%)>80 μmol/(m2·s)(6.87%)>120 μmol/(m2·s)(6.67%)(图 2)。
光照强度对STR01脂肪酸组成和含量的影响 由表 3所示, 不同光照强度对该藻PUFA影响显著(P< 0.05), 低光照[40 μmol/(m2·s)]时PUFA含量最高(34.29%)。PUFA中主要是C20﹕4 (n–6)的含量, 光照[80 μmol/(m2·s)]时达18.20%。该藻的EPA和DHA含量不高, 各组差异显著(P< 0.05)。光照强度为20 μmol/(m2·s)该藻EPA最高(2.88%), 光照强度为80 μmol/(m2·s)该藻DHA最高(2.23%)。
表 3 光照强度对STR01的脂肪酸组成和含量的影响Table 3. Effects of light intensity on fatty acid composition and content in STR01 (%)脂肪酸
Fatty acid光照强度
Light intensity [μmol/(m2·s)]40 60 80 100 120 C14﹕1 2.23±0.21a 1.51±0.25b 0.82±0.43c 1.67±0.16b 2.50±0.20a C14﹕0 9.64±0.11d 12.00±0.19c 17.30±0.26a 8.00±0.20e 13.20±0.21b C15﹕0 1.02±0.13c 1.13±0.10b 1.89±0.08a 1.08±0.07c 1.32±0.12b C16﹕1 7.38±0.11c 8.25±0.18b 10.40±0.23a 7.20±0.07c 10.40±0.27a C16﹕0 31.20±0.19a 31.90±0.15a 25.10±0.13c 31.80±0.21a 29.90±0.13b C18﹕2 (n–6) 1.68±0.12a 1.61±0.21a 1.47±0.32b 1.26±0.07b 1.19±0.09c C18﹕3 (n–3) 2.58±0.31d 4.16±0.26b 5.99±0.24a 4.18±0.11a 3.68±0.17c C18﹕0 16.70±0.09c 19.00±0.19b 13.10±0.12d 20.10±0.22a 21.40±0.15a C20﹕5 (n–3) 2.88±0.09a 1.08±0.19c 1.71±0.12b 1.53±0.12b 1.10±0.07c C20﹕4 (n–6) 17.70±0.13b 14.21±0.25c 18.20±0.21a 17.90±0.29b 12.00±0.21d C20﹕2 2.26±0.11a 1.84±0.18c 0.09±0.01e 2.03±0.09b 1.05±0.16d C22﹕6 (n–3) 1.09±0.10c 1.20±0.09b 2.23±0.28a 1.32±0.14b 0.91±0.21c C22﹕2 3.09±0.15a 2.03±0.17b 0.79±0.16d 1.94±0.15b 1.25±0.15c SFA 59.76±0.18c 64.03±0.13b 57.39±0.23d 65.98±0.10a 66.82±0.17a MUFA 10.01±0.21c 9.76±0.12d 11.22±0.11b 8.87±0.17e 13.00±0.22a PUFA 34.29±0.45a 27.72±0.05d 33.29±0.15b 28.16±0.10c 23.08±0.31e n–3 PUFA 18.58±0.17c 20.16±0.21b 24.22±0.08a 17.32±0.22d 20.59±0.28b n–6 PUFA 19.48±0.18c 15.61±0.29d 20.57±0.30b 21.90±0.14 a 13.19±0.12e 2.3 盐度对STR01的生长、总脂含量及脂肪酸的影响
盐度对STR01生长的影响 不同的盐度对STR01的平均相对生长速率影响显著(P<0.05)。在盐度10—35, 随着盐度的升高STR01平均相对生长速率呈先上升后降低趋势, 盐度25时平均相对生长速率最高(K为0.634), 盐度为10时扭鞘藻的数量几乎没有增加(图 3)。该藻生长的适合盐度范围是10—35, 最适盐度为25。
盐度对STR01的总脂含量影响显著(P< 0.05)。在盐度10—35, 随着盐度的升高, 该藻总脂含量呈先增加后减少趋势, 盐度30时最高(13.66%), 且显著高于其他组(图 3)。
盐度对STR01脂肪酸组成和含量的影响 盐度对STR01的脂肪酸组成及含量影响显著(P< 0.05)。盐度10—35 时PUFA的含量随着温度的升高呈现先上升后下降的趋势, 盐度25时PUFA的含量最高(43.42%)。PUFA中主要是C20﹕4 (n–6)的含量, 盐度25时达27.20%。在盐度10—35 条件下EPA含量为1.06%—9.80%, DHA含量为1.92%—2.66%。在盐度25、30、35时EPA含量大幅度增加, 分别达9.80%、9.16%和7.25%, 特别是盐度25时, EPA含量高达9.80%(表 4)。
表 4 盐度对STR01的脂肪酸组成和含量的影响Table 4. Effects of salinity on fatty acid composition and content in STR01 (%)脂肪酸
Fatty acid盐度
Salinity10 15 20 25 30 35 C14﹕1 1.44±0.11a 1.45±0.18a 1.47±0.24a 0.53±0.12b 0.57±0.14b 0.43±0.06c C14﹕0 11.80±0.25e 12.40±0.33d 12.60±0.28d 17.80±0.19c 18.50±0.09b 22.20±12a C15﹕0 1.80±0.04a 1.79±0.19a 1.76±0.07a 1.16±0.20b 0.91±0.18c 0.90±0.23c C16﹕1 10.20±0.13d 10.80±0.24d 10.90±0.21d 12.10±0.12c 14.80±0.09b 16.80±0.11a C16﹕0 31.40±0.23a 31.40±0.22a 30.50±0.09b 18.90±0.25d 20.10±0.17c 21.00±0.09c C18﹕2 (n–6) 0 1.46±0.21b 1.60±0.20b 1.89±0.13a 1.46±0.07b 1.06±0.11c C18﹕3 (n–3) 5.37±0.10a 4.67±0.30b 4.75±0.05b 1.44±0.12c 1.46±0.20c 0.95±0.34d C18﹕0 16.80±0.19a 16.20±0.15a 16.20±0.05a 6.13±0.14b 6.72±0.10b 5.97±0.14c C20﹕5 (n–3) 1.24±0.32c 1.07±0.03d 1.06±0.12d 9.80±0.11a 9.16±0.09a 7.25±0.17b C20﹕4 (n–6) 14.50±0.07d 14.00±0.18d 14.50±0.11d 27.20±0.32a 22.10±0.16b 20.10±0.23c C20﹕2 1.18±0.21a 1.20±0.22a 0.97±0.09a 0.35±0.11c 0.75±0.11b 0.69±0.09c C22﹕6 (n–3) 2.42±0.12a 2.04±0.08a 2.35±0.13a 2.23±0.25a 2.66±0.31a 1.92±0.04b C22﹕2 1.87±0.17a 1.59±0.09b 1.33±0.26c 0.51±0.05e 0.76±0.11d 0.62±0.21d SFA 61.80±0.34a 61.79±0.03a 61.06±0.22a 43.99±0.13d 46.23±0.19c 50.07±0.23b MUFA 11.64±0.09d 12.26±0.22c 12.37±0.17c 12.63±0.19c 15.37±0.34b 17.23±0.26a PUFA 26.58±0.40d 26.03±0.32d 26.56±0.12d 43.42±0.36a 38.35±0.11b 32.59±0.15c n–3 PUFA 9.03±0.14c 7.78±0.21d 8.16±0.31d 13.47±0.05a 13.28±0.10a 10.12±0.11b n–6 PUFA 14.50±0.03f 15.46±0.40e 16.10±0.25d 29.09±0.31a 23.56±0.13b 21.16±0.06c 2.4 温光盐正交试验对STR01生长、总脂及脂肪酸的影响
温光盐正交试验对STR01生长和总脂的影响 采用温度(A)、光照强度(B)、盐度(C)进行正交试验, 结果藻细胞生长和总脂含量见表 5。生长最优组合为: A3B2C2, 即温度30℃、光照强度60 μmol/(m2·s)、盐度25, 该组合时平均相对生长速率最大(K值达0.756); 总脂含量积累的最优组合为A3B2C1即温度30℃、光照强度60μmol/(m2·s)、盐度20, 总脂含量可达20.00%。
表 5 STR01生长速率和总脂正交试验设计Table 5. Orthogonal test design of the relative growth ratio and total lipid combination of STR01试验号
Test numberA B C 生长速率
K-value总脂含量
Total fat
content (%)1 1 1 1 0.385 14.8 2 1 1 2 0.419 12.3 3 1 1 3 0.288 16.4 4 1 2 1 0.382 14.2 5 1 2 2 0.328 12.8 6 1 2 3 0.285 13.6 7 1 3 1 0.160 12.5 8 1 3 2 0.318 15.7 9 1 3 3 0.223 12.3 10 2 1 1 0.333 13.8 11 2 1 2 0.524 14.4 12 2 1 3 0.606 17.5 13 2 2 1 0.360 17.0 14 2 2 2 0.723 16.4 15 2 2 3 0.580 13.1 16 2 3 1 0.406 17.1 17 2 3 2 0.619 12.6 18 2 3 3 0.641 14.3 19 3 1 1 0.560 12.2 20 3 1 2 0.506 13.5 21 3 1 3 0.571 14.7 22 3 2 1 0.494 20.0 23 3 2 2 0.756 16.2 24 3 2 3 0.673 16.1 25 3 3 1 0.460 14.1 26 3 3 2 0.521 13.7 27 3 3 3 0.504 14.4 影响STR01生长的正交因素主次为A>C>B, 3因素中温度对STR01平均相对生长速率影响最大(表 6), 在置信区间95%的情况下, 得出3个因素对平均相对生长速率影响的显著性排序为FA>FC>FB, 即温、光、盐对该藻平均相对生长速率的可靠性影响的大小顺序为A>C>B, 与方差分析的结果一致(表 7)。温度和盐度对该藻生长速率的影响有高度显著差异(P=0.000); 光照强度对STR01平均相对生长速率有显著差异(P=0.012); 温度和盐度有交互作用, 且交互作用极度显著(P=0.000); 温度、光照强度和盐度这三者的交互作用显著(P=0.001)。
表 6 生长速率极差分析Table 6. Range analyses of relative growth ratio试验号
Test number温度
Temperature光照强度
Light intensity盐度
Salinity第一水平均值Ki1 0.310 0.466 0.393 第二水平均值Ki2 0.532 0.509 0.524 第三水平均值Ki3 0.561 0.428 0.486 极差R 0.251 0.081 0.131 极差顺序
Extreme order1 3 2 表 7 生长速率方差分析Table 7. ANOVA of the relative growth ratio来源
SourceⅢ型平方和
Type Ⅲ SS自由度
df均方
MSF值
F valuesP值
P values温度
Temperature0.835 2 0.418 59.921 0.000 光照
Light intensity0.066 2 0.033 4.759 0.012 盐度
Salinity0.244 2 0.122 17.490 0.000 温度×光照
Temperature×
Light intensity0.038 4 0.009 1.357 0.261 温度×盐度
Temperature×
Salinity0.212 4 0.053 7.614 0.000 光照×盐度
Light intensity×
Salinity0.002 4 0.001 0.084 0.987 温×光×盐
Temperature×Light
intensity×Salinity0.209 8 0.026 3.753 0.001 同样影响STR01总脂正交因素主次为B>A>C, 即光照强度对总脂含量的影响最大(表 8、表 9)。温度和光照强度对该藻总脂含量影响高度显著(P=0.000), 盐度对该藻总脂含量影响有显著差异(P=0.002)。温度和光照、光照和盐度之间有交互作用(P=0.000); 温、光、盐三者有交互作用(P=0.000)。
表 9 总脂含量方差分析Table 9. ANOVA of total lipid content来源
SourceⅢ型平方和
Type Ⅲ SS自由度
df均方
MSF值
F valuesP值
P values温度
Temperature28.583 2 14.292 17.659 0.000 光照
Light intensity29.858 2 14.929 18.446 0.000 盐度
Salinity11.807 2 5.904 7.294 0.002 温度×光照
Temperature×
Light intensity61.505 4 15.376 18.999 0.000 温度×盐度
Temperature×
Salinity5.218 4 1.305 1.612 0.185 光照×盐度
Light intensity×
Salinity68.540 4 17.135 21.172 0.000 温×光×盐
Temperature×Light
intensity×Salinity82.101 8 10.263 12.681 0.000 表 8 总脂含量极差分析Table 8. Range analyses of total lipid content实验号
Test number温度
Temperature光照强度
Light intensity盐度
Salinity第一水平均值Ki1 13.844 14.400 15.078 第二水平均值Ki2 15.133 15.489 14.178 第三水平均值Ki3 14.989 14.078 15.300 极差R 1.289 1.411 1.122 极差顺序
Extreme order2 1 3 温光盐正交试验对STR01脂肪酸组成和含量的影响 在试验设定的组合下, PUFA含量最高时的组合为A2B2C1(温度25℃、光照强度60 μmol/(m2·s)、盐度20), 此时PUFA含量为35.37%, 该组合与组合为A2B3C1(温度25℃、光照强度80 μmol/(m2·s)、盐度20)无显著性差异。PUFA中主要是C20﹕4 (n–6)的含量, 组合为A2B2C1(温度25℃、光照强度 60 μmol/(m2·s)、盐度20)时C20﹕4 (n–6)的含量最高为23.80%, 组合为A3B2C1(温度30℃、光照强度60 μmol/(m2·s)、盐度20)时含量最高为21.90%。该藻的EPA和DHA含量虽然极少, 但在A3B2C1(温度30℃、光照强度60 μmol/(m2·s)、盐度20)条件下该藻的EPA含量达到最高(2.91%) A2B2C1 (温度25℃、光照强度60 μmol/(m2·s)、盐度20)条件下该藻的DHA含量达到最高(2.56%)(表 10)。
表 10 正交试验对扭鞘藻的脂肪酸组成和含量的影响Table 10. Effect of orthogonal test on fatty acid composition and content in STR01 (%)脂肪酸
Fatty acid三因子
Three-factorA1B1C1 A1B2C1 A1B3C1 A1B1C2 A1B2C2 A1B3C2 A1B1C3 A1B2C3 A1B3C3 C14﹕1 2.10±0.27b 1.72±0.19c 1.85±0.21c 1.84±0.13c 1.98±0.22c 1.72±0.09c 3.29±0.10a 2.36±0.31b 1.96±0.11c C14﹕0 11.70±0.52e 14.80±0.25c 11.00±0.13e 11.60±0.08e 10.80±0.12e 10.30±0.14e 6.69±0.22g 8.45±0.03f 11.40±0.17d C15﹕0 1.35±0.43b 1.67±0.18b 1.41±0.45b 1.17±0.04c 1.02±0.36c 1.22±0.08c 0.65±0.15c 1.09±0.34c 1.78±0.03b C16﹕1 10.60±0.75a 10.10±0.43b 11.20±0.06a 9.80±0.13b 10.10±0.21b 11.40±0.56a 7.48±0.22c 10.30±0.07b 10.20±0.61b C16﹕0 29.70±0.27b 27.80±0.76c 28.40±0.14c 26.60±0.04d 28.74±0.14b 27.80±0.94c 30.57±0.65a 28.10±0.90b 27.70±0.54c C18﹕2 (n–6) 0.66±0.09e 1.03±0.07d 0.58±0.17f 0.63±0.34f 0.99±0.28d 0.75±0.08e 1.11±0.09d 0.62±0.08f 0.93±0.04e C18﹕3 (n–3) 3.16±0.09e 4.00±0.04f 5.83±0.12d 5.66±0.08d 5.59±0.18e 5.89±0.18d 7.91±0.14b 6.68±0.17bc 5.22±0.24e C18﹕0 17.10±0.75d 16.30±0.61e 16.40±0.56e 16.80±0.59e 18.70±0.60d 17.00±0.43d 24.90±0.91a 20.70±0.23c 17.10±0.67d C20﹕5 (n–3) 0.24±0.05d 1.01±0.14c 0.55±0.04c 1.01±0.01c 0.59±0.02c 0.58±0.01c 0 0.36±0.07d 0.91±0.05c C20﹕4 (n–6) 19.90±0.21b 17.60±0.45d 16.80±0.87d 20.70±1.01b 16.50±0.88d 18.60±1.41c 11.40±0.55g 15.90±0.82d 17.50±0.98d C20﹕2 1.21±0.07c 1.44±0.07c 1.26±0.01c 0.99±1.01b 1.38±0.04c 1.27±0.03c 2.68±0.04a 1.53±0.02c 1.32±0.07c C22﹕6 (n–3) 1.23±0.32d 1.48±0.23c 1.45±0.19c 0.98±0.08bc 1.33±0.01d 1.57±0.01c 0.78±0.13e 1.59±0.16c 1.72±0.05c C22﹕2 1.09±0.02d 1.00±0.23c 3.31±0.04a 2.21±0.23d 2.28±0.17d 2.30±0.03d 2.57±0.03c 2.32±0.08bd 2.25±0.01d SFA 59.85±0.99c 60.57±1.32c 57.21±1.45e 56.17±0.85f 59.26±1.02d 56.32±0.90e 62.81±1.17b 58.34±1.81d 57.98±0.83d MUFA 12.70±0.15a 11.82±0.13b 13.05±0.08a 11.64±0.34a 12.08±0.42a 13.12±0.27a 10.77±0.51b 12.66±0.46a 12.16±0.29a PUFA 27.49±0.97e 27.56±1.45e 29.78±0.90c 32.18±1.07b 28.66±0.86d 30.96±1.21c 26.45±0.94e 29.00±0.99d 29.85±1.27c n–3PUFA 4.63±0.02f 6.49±0.04d 7.83±0.03b 7.65±0.07b 7.51±0.25c 8.04±0.24b 7.91±0.42a 8.63±0.99e 7.85±0.21c n–6PUFA 20.56±0.76b 18.63±0.23c 17.38±0.65d 21.33±0.78b 17.49±0.08d 19.35±0.93c 12.51±0.67g 16.52±0.87e 18.43±0.96d 3. 讨论
3.1 温度对微藻的影响
温度是影响微藻生长和代谢的重要环境因子之一[11]。温度对海洋微藻的生长及发育等具有调节作用, 对营养物的吸收利用效率、酶的活性、及细胞分裂的周期等存在不同程度不同方式的影响[12]。正交试验结果表明, 温度对STR01平均相对生长速率影响最大, 单因子试验表明: STR01生长的适温为15—35℃, 最适温度25—30℃ (0.679—0.682)。该藻在15—35℃内, 随着温度升高平均相对生长速率呈先上升后降低趋势, 在30℃时该藻的平均相对生长速率最大(K值为0.682), 但25℃组与30℃组平均相对生长速率差异不显著(P> 0.05), 温度在10℃条件下, 培养7d大部分藻死亡分解, 藻液颜色变白。这一结果与金德祥等[13]研究扭鞘藻对温度的适应范围是在10—30℃, 最适温度是在20—25℃的试验结果不一致, 可能由于其试验环境、条件和藻种不同所致。不同海洋微藻对不同的温度的适应能力不同, 海链藻(Thalassiossira sp.)在温度24—27℃时, 其生长则明显受到抑制, 在30℃时则完全不能生长, 但在18—24℃温度范围内, 海链藻具有较高的生长速率[14], 而牟氏角毛藻(Chaetoceros muelleri)是一种耐高温藻种, 其生长的最佳温度为25—35℃。本试验STR01在10℃以下完全不能生长, 在35℃时平均相对生长速率明显降低。
有研究表明, 在极端低温或高温情况下, 微藻合成油脂的含量减少[15—17], STR01在低温15℃和高温35℃时总脂积累很少, 总脂含量也相差不大, 低温和高温对微藻的危害方式并不相同。Opute[16]认为海洋微藻的油脂合成, 在极端温度下微藻的油脂合成受限, 可能是由于合成脂肪相关的酶发生不可逆损伤。低温对微藻的破坏是机械性, 而高温对微藻的破坏是化学性, 大量的研究表明微藻对低温的抵抗和忍耐性较强。不同藻种总脂含量积累的最适温度不同, 本试验总脂含量积累的最适温度是25℃(总脂含量可达17.23%)。三角褐指藻(Phaeodactylum tricornutum)的诱变株MP-2, 总脂积累的最适温度是20℃[18]。硅藻的脂肪酸主要成分有C14﹕0、C16﹕1 (n–7)、C16﹕0, 有些硅藻的富含EPA。扭鞘藻属于硅藻, 本试验表明其主要脂肪酸成分是C14﹕0、C16﹕1、C16﹕0、C18﹕0和C20﹕4 (n–6), 其EPA含量不高。有研究表明海链藻低温处理可明显提高长链多不饱和脂肪酸含量, 但并不是其积累PUFAs的最佳温度[11]。在本试验的单因子试验中, 温度20℃下, PUFA含量最高达34.23%, 在正交试验中, 温度为25℃时, PUFA积累较多, 而30℃该藻平均相对生长速率最高, 若要获得较多的不饱和脂肪酸可考虑分步培养。
3.2 光照强度对微藻的影响
光照影响微藻的生长速率和光合作用, 对细胞体内生化成分产生影响, 光强和照射时间的长短影响微藻光合作用的效率[14, 19]。光是影响海洋微藻生化成分种类和含量多少及其生长的最重要的因子之一, 有研究实验结果表明超过一定的光照强度范围, 微藻生长会受到抑制。本次试验光照强度为60 μmol/(m2·s)时, STR01平均相对生长速率K值达到最大(0.630), 当光照强度超过100 μmol/(m2·s)时, STR01的平均相对生长速率明显降低, 说明高光强不利于该藻的生长。不同的藻种的生长适应的光强不一样, 如后棘藻(Ellipsoidion sp.)在光强较高下生长速率高, 其适宜光强范围为108.75—244.15 μmol/(m2·s), 光强低于71.12 μmol/(m2·s)时生长速率有所降低[20]。多方面的研究显示, 浮游海洋微藻在变化的环境中生存和生长的, 其生理适应是关键过程, 水生浮游植物为了提高其不同环境中生存能力, 可以通过提高对环境因子的适应能力来实现, 特别是在资源能够循环再生的情况下, 这种调节机制更加的明显[21]。
正交试验结果表明, 光照强度对STR01总脂含量的积累影响最大, 单因子试验表明光照强度为60 μmol/(m2·s)时, STR01的总脂含量最高可达 16.73%。单因子试验结果表明, 随着光照强度的升高, 总脂含量呈下降趋势, 表明高光不利于STR01总脂的积累。国内学者对微绿球藻[22]、后棘藻[19]、小球藻和三角褐指藻[23]的研究中也发现PUFA含量随光强增加而呈下降趋势。低光下两种藻小新月菱形藻和等鞭金藻的脂肪含量多, 而高光下则相反[23]。低光照[20 μmol/(m2·s)]更有利于2 株曼氏骨条藻(Skeletonema munzelii)总脂及PUFA的积累[22], 单因子光照强度试验和正交试验都结果表明光强[60 μmol/(m2·s)]有利于STR01的PUFA的积累, 单因子试验结果表明, 随着光照强度的升高, PUFA呈下降趋势, 表明高光不利于STR01PUFA的积累。
3.3 盐度对微藻的影响
本试验结果表明, 适合STR01生长的盐度为15—35, 最适生长盐度为25(K值0.634)。盐度影响藻类的渗透压和营养盐的吸收。当微藻生活的盐度环境发生改变时, 细胞的渗透压受到影响也随之改变, 过低或过高的盐度均会对微藻造成伤害[24, 25]。盐度在 10 以下, STR01几乎完全不能生长, 在盐度 15—35 时, K值呈现先上升后下降趋势。藻种不同, 对盐度的适应力也不同, 亚历山大藻(Alexandrium sp.) 偏嗜较高盐度环境, 在低盐度下几乎不能生长。蛋白核小球藻适盐范围比较广, 在盐度15—45范围内均可以快速生长。
不同藻种的在不同的盐度下, 总脂含量的积累也不同, 本试验STR01总脂含量积累的适宜盐度范围是10—35, 盐度30时, 总脂含量最高(13.66%)。有研究表明高盐有利于总脂的积累, 虽然高盐下微藻的生长率不高。在高盐度下微藻的生长需要更多能量, 藻细胞内的储存的脂肪的含量相应增加[26, 27]。三角褐指藻在较高盐度30—35条件下, 其总脂含量显著高于其他组[18], 本试验在高盐度 30 时总脂含量最高, 与上述试验结果吻合。这种藻产脂应用中可分两步培养。增大培养基的盐度虽使得微绿球藻的生长速率降低, 但有利于油脂积累[28]。本试验的盐度单因子试验表明, 在盐度25时, PUFA含量最高(43.42%), 在正交试验中, 较低盐度(20)有利于该藻PUFA的积累, 在25℃、光照强度60 μmol/(m2·s)和盐度20的条件下STR01的PUFA含量达到35.37%。有试验研究表明, 低盐有利于微藻PUFA的积累, 如盐度(15)更有利于2 株曼氏骨条藻PUFA的积累[22]; 在对绿色巴夫藻的研究中, 低盐度条件下有利于PUFA (n–3)的合成, 盐度为6时C20﹕5 (n–3)的含量最高, 为总脂的6.02%。在实际的大规模培养中, 可以考虑现在最适微藻生长的盐度下培养藻细胞, 然后再转到低盐下培养收集到更多的PUFA。
-
图 1 虹鳟FcγRIα (A)、FcRγ1 (B)、FcRγ2a (C)和FcRγ2b (D)的cDNA及预测氨基酸序列
信号肽、免疫球蛋白样结构域、跨膜区和免疫受体酪氨酸基序分别被标注, 潜在N-糖基化位点以灰色阴影表示, 终止密码子和多聚腺苷酸化信号(AATAAA)以下划线表示
Figure 1. cDNA and predicted amino acid sequences of trout FcγRIα (A)、FcRγ1 (B)、FcRγ2a (C) and FcRγ2b (D)
Predicted signal peptide, Ig-like domains, transmembrane regions, and ITAM motif are labeled, potential N-linked glycosylation sites are gray shaded, the stop codon and polyadenylation signal (AATAAA) are underlined
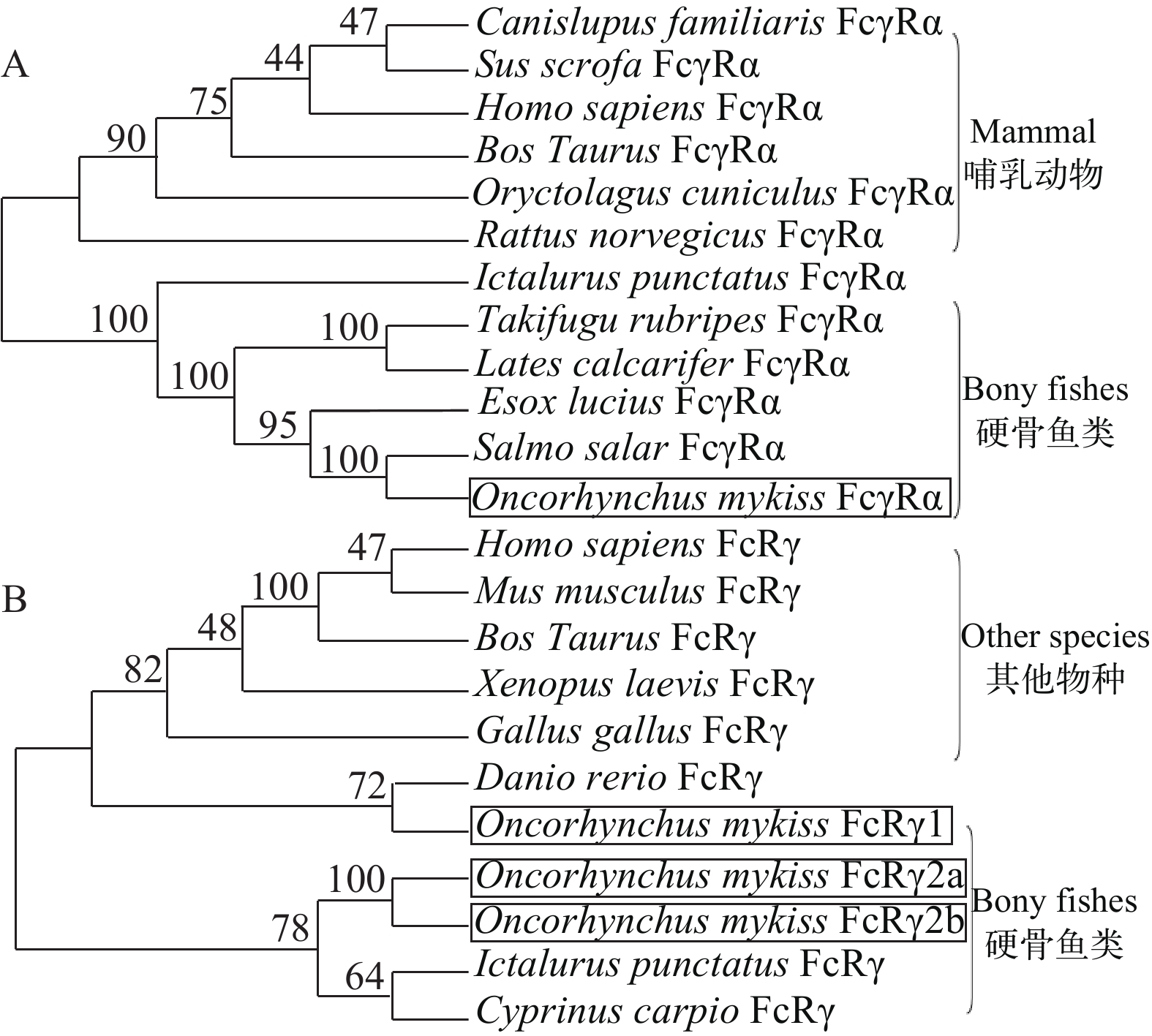
图 3 脊椎动物FcγRα(A)和FcRγ(B)氨基酸序列的系统进化树
数字代表自举置换1000次的各分支置信度(%)
Figure 3. Phylogenetic analysis of amino acid sequences of vertebrate FcγRα (A) and FcRγ (B)
Neighbor-joining trees were reconstructed with 1000 bootstrap replications (%)各物种FcγRα和FcRγ蛋白序列登录号: 虹鳟Oncorhynchus mykiss FcγRα (MG993321)、狗Canislupus familiaris FcγRα (NP_001002976.1)、猪Sus scrofa FcγRα (NP_001028183.1)、人Homo sapiens FcγRα (NP_000557.1)、牛Bos Taurus FcγRα (NP_776963.1)、兔Oryctolagus cuniculus FcγRα (XP_017201412.1)、大鼠Rattus norvegicus FcγRα (NP_ 001094306.1)、斑点叉尾鮰Ictalurus punctatus FcγRα (NP_ 001187150.1)、红鳍东方鲀Takifugu rubripes FcγRα (XP_ 011606648.1)、尖吻鲈Lates calcarifer FcγRα (XP_ 018557658.1)、白斑狗鱼Esox lucius FcγRα (XP_ 010897459.1)、大西洋鲑Salmo salar FcγRα (ACN10664.1); 人H. sapiens FcRγ (AAH33872.1)、小鼠Mus musculus FcRγ (NP_034315.1)、牛B. taurus FcRγ (NP_776962.1)、非洲爪蟾Xenopus laevis FcRγ (NP_001082448.1)、鸡Gallus gallus FcRγ (NP_001092081.1)、斑马鱼Danio rerio FcRγ (ABO61033.1)、虹鳟O. mykiss FcRγ1 (MG993322)、虹鳟O. mykiss FcRγ2a (MG993323)、虹鳟O. mykiss FcRγ2b (MG993324)、斑点叉尾鮰I. punctatus FcRγ (NP_001187098.1)、鲤Cyprinus carpio FcRγ (BAA92270.1)
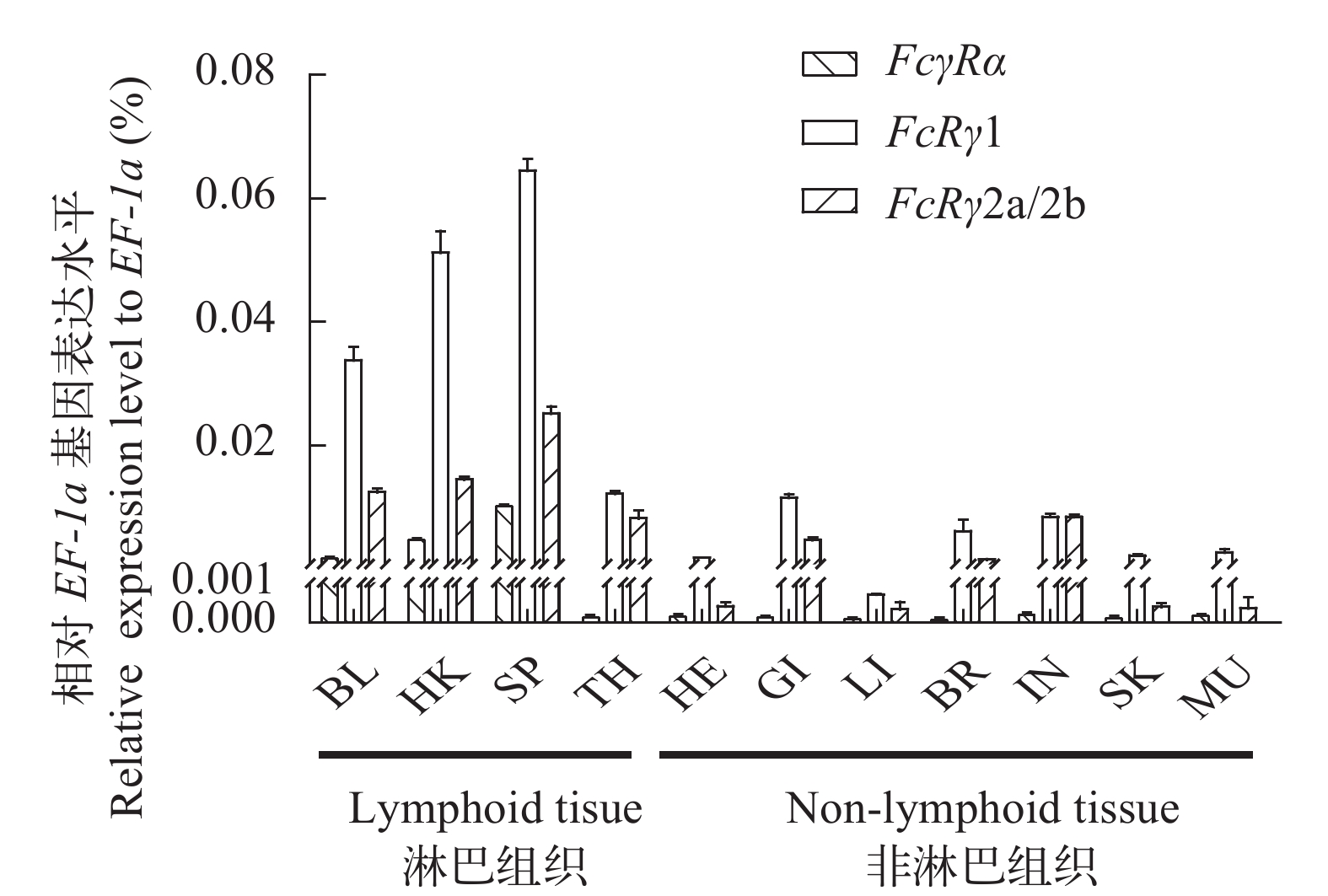
图 5 虹鳟FcγR基因的组织表达分布
BL. 血液, HK. 头肾, SP. 脾脏, TH. 胸腺, HE. 心脏, GI. 鳃, LI. 肝脏, BR. 脑, IN. 肠, SK. 皮肤, MU. 肌肉
Figure 5. Expression patterns of FcγR in different tissues of rainbow trout
BL. blood, HK. head kidney, SP. spleen, TH. thymus, HE. heart, GI. gill, LI. liver, BR. brain, IN. intestine, SK. skin, MU. muscle
图 2 虹鳟FcγRα与其他鱼类相应分子氨基酸序列比对(A)和虹鳟FcRγ与其他物种相应分子氨基酸序列比对(B)
各物种FcγRα和FcRγ蛋白序列登录号: 虹鳟Oncorhynchus mykiss FcγRα (MG993321)、斑点叉尾鮰Ictalurus punctatus FcγRα (NP_001187150.1)、大西洋鲑Salmo salar FcγRα (ACN10664.1)、白斑狗鱼Esox lucius FcγRα (XP_010897459.1)、红鳍东方鲀Takifugu rubripes FcγRα (XP_011606648.1)、尖吻鲈Lates calcarifer FcγRα (XP_018557658.1); 虹鳟O. mykiss FcRγ1 (MG993322)、虹鳟O. mykiss FcRγ2a (MG993323)、虹鳟O. mykiss FcRγ2b (MG993324)、斑马鱼Danio rerio FcRγ (ABO61033.1)、人Homo sapiens FcRγ (AAH33872.1)、小鼠Mus musculus FcRγ (NP_034315.1)、牛Bos Taurus FcRγ (NP_776962.1)、非洲爪蟾Xenopus laevis FcRγ (NP_001082448.1)、斑点叉尾鮰I. punctatus FcRγ (NP_001187098.1)、鲤Cyprinus carpio FcRγ (BAA92270.1)
Figure 2. Amino acid alignment of trout FcγRα with the related sequences in other fish (A); Amino acid alignment of trout FcRγ with the related sequences in other species (B)
表 1 本文中所用的引物序列
Table 1 Primers used in the present study
引物名称
Primer name引物序列
Primer Sequence (5ʹ-3ʹ)退火温度
Tm (℃)用途
ApplicationFcγRα-F ACTCAGCCTCCTTGTTGTTTCA 66 克隆中间
片段FcγRα-R ACTGGAGCAGCCACTGAGATTA 66 FcRγ1-F GCTGTGATGGGAGGCAGATTC 66 FcRγ1-R TCCGCTTTCGGTGGCTTTTCT 64 FcRγ2a-F ACCGTGTCTTTCATTTTGTGCCT 66 FcRγ2a-R TGTTGTTTACCACCACTCACAC 66 FcRγ2b-F ATCTTGGTCGCTCTACTCTTG 66 FcRγ2b-R AGGTGTCCTGACCTTTATGGG 66 FcγRα-R1 GATTACAGAGTTGGTTTCCCTC 64 5ʹRACE FcγRα-R2 CACCCACCGTCCACCTCTAT 64 FcRγ1-R1 TCCGCTTTCGGTGGCTTTTCT 66 FcRγ1-R2 TAGAGCACGGTGAGGATGATAC 64 FcRγ2a-R1 GTACAAGATGGTGAGAAGGACG 66 FcRγ2a-R2 GTACAAGATGGTGAGAAGGACG 66 FcRγ2b-R1 CAAGAGTAGAGCGACCAAGAT 66 FcRγ2b-R2 CTGTGGATACCCGCCAGTGA 66 FcγRα-F1 ATAGAGGTGGACGGTGGGTG 64 3ʹRACE FcγRα-F2 GAGGGAAACCAACTCTGTAATC 64 FcRγ1-F1 GTATCATCCTCACCGTGCTCTA 66 FcRγ1-F2 AGAAAAGCCACCGAAAGCGGA 64 表 2 虹鳟FcRγ各亚型间氨基酸序列相同率
Table 2 Amino acid sequence identity between the subunit of FcRγ in rainbow trout
FcRγ1 (%) FcRγ2a (%) FcRγ2b (%) FcRγ1 100 40.98 45.37 FcRγ2a 100 90.01 FcRγ2b 100 -
[1] 史平玲, 王爱萍, 乔松林, 等. 家畜IgG Fc受体研究进展[J]. 河南农业科学, 2010, (1): 124—128
[2] Akula S, Mohammadamin S, Hellman L. Fc receptors for immunoglobulins and their appearance during vertebrate evolution [J]. PLoS One, 2014, 10(4): e0124530
[3] Nimmerjahn F, Ravetch J V. Fc gamma receptors as regulators of immune responses [J]. Nature Reviews Immunology, 2008, 8(1): 34—47 doi: 10.1038/nri2206
[4] Edberg J C, Qin H, Gibson A W, et al. The CY domain of the Fc gamma RIa alpha-chain alter CD(64) gamma-chain tyrosine-based signaling and phagocytosis [J]. The Journal of Biological Chemistry, 2002, 277(43): 41287—41293 doi: 10.1074/jbc.M207835200
[5] Rivera J, Fierro N A, Olivera A, et al. New insights on mast cell activation via the high affinity receptor for IgE [J]. Advances in Immunology, 2008, 98(8): 85—120
[6] Kubagawa H, Oka S, Kubagawa Y, et al. Identity of the elusive IgM Fc receptor (FcmuR) in humans [J]. The Journal of Experimental Medicine, 2009, 206(12): 2779—2793 doi: 10.1084/jem.20091107
[7] Tamma S M, Amin A R, Finkelman F D, et al. IgD receptors on murine T-helper cells bind to Fd and Fc regions of immunoglobulin D [J]. Proceedings of the National Academy of Sciences of the United States of America, 1991, 88(20): 9233—9237 doi: 10.1073/pnas.88.20.9233
[8] Flesch B K, Neppert J. Functions of the Fc receptors for immunoglobulin G [J]. Journal of Clinical Laboratory Analysis, 2000, 14(4): 141—156 doi: 10.1002/(ISSN)1098-2825
[9] Cassatella M A, Flynn R M, Amezaga M A, et al. Interferon gamma induces in human neutrophils and macrophages expression of the mRNA for the high affinity receptor for monomeric IgG (Fc gamma R-I or CD64) [J]. Biochemical and Biophysical Research Communications, 1990, 170(2): 582—588 doi: 10.1016/0006-291X(90)92131-I
[10] Pan L Y, Mendel D B, Zurlo J, et al. Regulation of the steady state level of Fc gamma RI mRNA by IFN-gamma and dexamethasone in human monocytes, neutrophils, and U-937 cells [J]. Journal of Immunology, 1990, 145(1): 267—275
[11] Parren P W, Warmerdam P A, Boeije LC, et al. On the interaction of IgG subclasses with thelow affinity Fc gamma RIIa (CD32) on human monocytes, neutrophils, and platelets [J]. The Journal of Clinical Investigatio, 1992, 90(4): 1537—1546 doi: 10.1172/JCI116022
[12] Sondermann P, Kaiser J, Jacob U. Molecular basis for immune complex recognition: a comparison of Fc-receptor structures [J]. Journal of Molecular Biology, 2001, 309(3): 737—749 doi: 10.1006/jmbi.2001.4670
[13] Rankin B M, Yocum S A, Mittler R S, et al. Stimulation of tyrosine phosphorylation and calcium mobilization by Fc gamma receptor cross-linking. Regulation by the phosphotyrosine phosphatase CD45 [J]. Journal of Immunology, 1993, 150(2): 605—616
[14] Takai T, Li M, Sylvestre D, et al. FcR gamma chain deletion results in pleiotrophic effector cell defects [J]. Cell, 1994, 76(3): 519—529 doi: 10.1016/0092-8674(94)90115-5
[15] Anderson C L, Shen L, Eicher D M, et al. Phagocytosis mediated by three distinct Fc gamma receptor classes on human leukocytes [J]. The Journal of Experimental Medicine, 1990, 171(4): 1333—1345 doi: 10.1084/jem.171.4.1333
[16] Fanger M W, Shen L, Graziano R F, et al. Cytotoxicity mediated by human Fc receptors for IgG [J]. Immunology Today, 1989, 10(3): 92—99 doi: 10.1016/0167-5699(89)90234-X
[17] Clynes R, Maizes J S, Guinamard R, et al. Modulation of immune complex-induced inflammation in vivo by the coordinate expression of activation and inhibitory Fc receptors [J]. The Journal of Experimental Medicine, 1999, 189(1): 179—185 doi: 10.1084/jem.189.1.179
[18] Simister N E, Story C M. Human placental Fc receptors and the transmission of antibodies from mother to fetus [J]. Journal of Reproductive Immunology, 1997, 37(1): 1—23
[19] Nimmerjahn F, Gordan S, Lux A. Fc gammaR dependent mechanisms of cytotoxic, agonistic, and neutralizing antibody activities [J]. Trends in Immunology, 2015, 36(6): 325—336 doi: 10.1016/j.it.2015.04.005
[20] Osman N, Kozak C A, McKenzie I F, et al. Structure and mapping of the gene encoding mouse high affinity Fc gamma RI and chromosomal location of the human Fc gamma RI gene [J]. Journal of Immunology, 1992, 148(5): 1570—1575
[21] Qiu W Q, de Bruin D, Brownstein B H, et al. Organization of the human and mouse low-affinity Fc gamma R genes: duplication and recombination [J]. Science, 1990, 248(4956): 732—735 doi: 10.1126/science.2139735
[22] Nimmerjahn F, Bruhns P, Horiuchi K, et al. Fc gammaRIV: a novel FcR with distinct IgG subclass specificity [J]. Immunity, 2005, 23(1): 41—51 doi: 10.1016/j.immuni.2005.05.010
[23] Collins R A, Gelder K I, Howard C J. Nucleotide sequence of cattle FcGRIII: its identification in gammadelta T cells [J]. Immunogenetics, 1997, 45(6): 440—443 doi: 10.1007/s002510050228
[24] Qiao S, Liu Y, Zhang J, et al. Genetic characterization and ligand specificity of the ovine Fc gamma receptor Ⅰ (ovFc gamma RI) [J]. Veterinary Immunology and Immunopathology, 2010, 137(2): 317—321
[25] Kumura B H, Sone T, Shimazaki K, et al. Sequence analysis of porcine polymeric immunoglobulin receptor from mammary epithelial cells present in colostrum [J]. The Journal of Dairy Research, 2000, 67(4): 631—636 doi: 10.1017/S0022029900004404
[26] McAleese S M, Halliwell R E, Miller H R. Cloning and sequencing of the horse and sheep high-affinity IgE receptor alpha chain cDNA [J]. Immunogenetics, 2000, 51(10): 878—881 doi: 10.1007/s002510000200
[27] Zhang G, Young J R, Tregaskes C R, et al. Cattle Fc gamma RII: molecular cloning and ligand specificity [J]. Immunogenetics, 1994, 39(6): 423—427
[28] Sweeney S E, Kim Y B. Identification of a novel Fc gamma RIIIa alpha-associated molecule that contains significant homology to porcine cathelin [J]. Journal of Immunology, 2004, 172(2): 1203—1212 doi: 10.4049/jimmunol.172.2.1203
[29] Astier A, Salle H, Salle C, et al. Human epidermal Langerhans cells secrete a soluble receptor for IgG (Fc gamma RII/CD32) that inhibits the binding of immune complexes to Fc gamma R+cell [J]. Journal of Immunology, 1994, 152(1): 201—212
[30] Taylor A I, Gould H J, Sutton B J, et al. The first avian Ig-like Fc receptor family member combines features of mammalian FcR and FCRL [J]. Immunogenetics, 2007, 59(4): 323—328 doi: 10.1007/s00251-007-0195-9
[31] Guselnikov S V, Bell A, Najakshin A M, et al. Signaling FcRgamma and TCRzeta subunit homologs in the amphibian Xenopus laevis [J]. Developmental and Comparative Immunology, 2003, 27(8): 727—733 doi: 10.1016/S0145-305X(03)00055-7
[32] Fujiki K, Shin D H, Nakao M, et al. Molecular cloning and expression analysis of carp (Cyprinus carpio) interleukin-1 beta, high affinity immunoglobulin E Fc receptor gamma subunit and serum amyloid A [J]. Fish & Shellfish Immunology, 2000, 10(3): 229—242
[33] Stafford JL, Wilson M, Nayak D, et al. Identification and characterization of a FcR homolog in an ectothermic vertebrate, the channel catfish (Ictalurus punctatus) [J]. Journal of Immunology, 2006, 177(4): 2505—2517 doi: 10.4049/jimmunol.177.4.2505
[34] Wang W. Culture and Enhancement of Fishes [M]. China Agricultural Press. 2000, 469—480
[35] Zhang X J, Zhang X Y, Zhang N, et al. Distinctive structural hallmarks and biological activities of the multiple cathelicidin antimicrobial peptides in a primitive teleost fish [J]. Journal of Immunology, 2015, 194(10): 4974—4987
[36] Zhang N, Zhang X J, Song Y L, et al. Preferential combination between the light and heavy chain isotypes of fish immunoglobulins [J]. Developmental and Comparative Immunology, 2016, 61: 169—179 doi: 10.1016/j.dci.2016.04.001
[37] Zhang Y A, Hikima J, Li J, et al. Conservation of structural and functional features in a primordial CD80/86 molecule from rainbow trout (Oncorhynchus mykiss), a primitive teleost fish [J]. Journal of Immunology, 2009, 183(1): 83—96
[38] Zhang X J, Wang P, Zhang N, et al. B cell functions can be modulated by antimicrobial peptides in rainbow trout Oncorhynchus mykiss: novel insights into the innate nature of B cells in fish [J]. Frontiers in Immunology, 2017, 8: 388—392
[39] Ravetch J V, Bolland S. IgG Fc receptors [J]. Annual Review of Immunology, 2001, 19(1): 275—290 doi: 10.1146/annurev.immunol.19.1.275
[40] Fridman W H, Teillaud JL, Bouchard C, et al. Soluble Fc gamma receptors [J]. Journal of Leukocyte Biology, 1993, 54(2): 504—512
[41] Galon J, Bouchard C, Fridman W H, et al. Ligands and biological activities of soluble Fc gamma receptors [J]. Immunology Letters, 1995, 44(2): 175—181
[42] Galon J, Paulet P, Galinha A, et al. Soluble Fc gamma receptors: interaction with ligands and biological consequences [J]. International Reviews of Immunology, 1997, 16(1): 87—111
[43] Galon J, Gauchat J F, Mazieres N, et al. Soluble Fcgamma receptor type Ⅲ (FcgammaRIII, CD16) triggers cell activation through interaction with complement receptors [J]. Journal of Immunology, 1996, 157(3): 1184—1192
[44] Hamerman J A, Lanier L L. Inhibition of immune responses by ITAM-bearing receptors [J]. Science Signal, 2006, (32031): 300—321
[45] Kacskovics I. Fc receptors in livestock species [J]. Veterinary Immunology and Immunopathology, 2004, 102(3): 351—362
[46] Chettri J K, Raida M K, Holten-Andersen L, et al. PAMP induced expression of immune relevant genes in head kidney leukocytes of rainbow trout (Oncorhynchus mykiss) [J]. Developmental and Comparative Immunology, 2011, 35(4): 476—482 doi: 10.1016/j.dci.2010.12.001
[47] Nimmerjahn F, Ravetch J V. Fcgamma receptors: old friends and new family members [J]. Immunity, 2006, 24(1): 19—28 doi: 10.1016/j.immuni.2005.11.010
-
期刊类型引用(4)
1. 夏心怡,彭瑞冰,张聪颖,蒋霞敏. 光质对新月菱形藻生长、总脂含量和脂肪酸组成的影响. 广东海洋大学学报. 2023(05): 53-59 .  百度学术
百度学术
2. 杨宇航,边雪强,孙翠竹,姜晓华,单欣,李锋民. 光照强度对丝状藻去除再生水中氮磷的影响. 环境科学研究. 2022(05): 1211-1220 .  百度学术
百度学术
3. 汤坤贤,宋晖,姜德刚,孙元敏,蔡鹭春,陈珊,涂武林. 遮光处理对微藻繁殖及其水质指标的影响. 环境科技. 2022(03): 25-31 .  百度学术
百度学术
4. 刘权迪,宁延昶,温斌,张峰,吴香莹,左然涛. 饲料中脂肪水平对海湾扇贝性腺发育、脂肪酸组成和组织结构的影响. 上海海洋大学学报. 2021(06): 981-991 .  百度学术
百度学术
其他类型引用(5)



 下载:
下载: