MIRNAS SEQUENCING AND ANALYSIS OF INTERMUSCULAR BONE BETWEEN RICE FLOWER CARP AND JIAN CARP
-
摘要: 为了研究禾花鲤(Rice flower carp)肌间骨柔软的分子调控机制, 采集禾花鲤和建鲤(Jian carp)的肌间骨进行micro RNAs (miRNAs)的高通量测序, 并进行了生物信息学分析。从禾花鲤肌间骨的小RNA文库测序获得18—32 nt的高质量miRNA序列25474895条、鉴定出成熟miRNAs 595种, 而从建鲤文库获得miRNA序列24625715条、成熟miRNAs 570种。表达差异分析结果显示, 与建鲤相比, 禾花鲤肌间骨中84个miRNAs呈上调表达、267个呈下调表达。禾花鲤下调表达的miRNAs中有7条为已报道的促进人类成骨的miRNAs, 上调表达的miRNAs中有6条为已报道的抑制人类成骨的miRNAs。因此推测禾花鲤可能是通过下调表达促进成骨的miRNAs及上调表达抑制成骨的miRNAs来抑制成骨过程, 从而维持其肌间骨细小及柔软的特性。研究为国内首次开展禾花鲤肌间骨发育的分子调控机制研究, 为水产动物肌间骨发育的研究奠定了基础。Abstract: In order to study the molecular regulation mechanism of the soft intermuscular bones in Rice flower carp, the intermuscular bones of Rice flower carp and Jian carp were collected for high-throughput sequencing of microRNAs (miRNAs) and bioinformatics analysis. 25474895 and 24625715 high quality sequences of 18—32 nt, as well as 595 and 570 known mature miRNAs were obtained from the small RNA library of Rice flower carp and Jian carp intermuscular bone, respectively. Compared with Jian carp, 84 miRNAs were up-regulated and 267 were down-regulated in the intermuscular bone of Rice flower carp. Seven of the down-regulated miRNAs in Rice flower carp were reported as promoting human osteogenesis. Six of the up-regulated miRNAs were reported as inhibit human osteogenesis. These results indicated that Rice flower carp may inhibit the process of osteogenesis by down-regulating the expression of osteogenic miRNAs and up-regulating the expression of osteogenesis inhibiting miRNAs. Therefore it can maintain its intermuscular bone small and soft characteristics. This study laid the groundwork for the study of molecular regulation mechanism of intermuscular bone development.
-
Keywords:
- Rice flower carp /
- Jian carp /
- Intermuscular bone /
- miRNAs /
- High-throughput sequencing
-
鲤(Cyprinus carpio)是目前世界上养殖历史最悠久, 养殖范围最广的淡水鱼类, 是许多国家的主要养殖品种[1], 在漫长的自然演化和人工驯养背景下, 形成了许多著名的鲤品种, 如黄河鲤、建鲤、黑龙江鲤等。广西全州禾花鲤(又名禾花鱼), 是最具广西地方特色的稻鱼共养品种, 也是鲤的一个独特的地方土著品种。禾花鲤因经稻田长期驯化, 食禾花而得名, 它体型粗短、鳞细皮薄、肉质细嫩、味道鲜美, 特别是肌间骨柔软细小且可食用, 清代乾隆年间曾成为宫廷贡品。建鲤是以荷包红鲤和沅江鲤为亲本选育的良种, 生长速度快、抗病能力强、含肉多。建鲤是我国养殖鲤鱼的代表性品种之一, 在我国广泛养殖[2, 3]。
鱼类的骨骼肌间骨又称为肌间刺, 是位于脊椎骨两侧肌间隔中的硬骨小刺, 是由肌膈间的结缔组织直接骨化而形成的[4]。肌间骨在低等真骨鱼类中普遍存在, 随着鱼类的演化, 其形态逐渐复杂化[5]。众多细小的肌间骨一直以来影响多种鱼类的食用品质, 因此, 鱼类肌间骨的形态和发育机制一直是水产研究的热点。其中, 关于真骨鱼类肌间骨的骨化模式研究主要集中在黄鳝(Monopterus albus)[6]、鲢(Hypophthalmichthys molitrix)[7]、鲫(Carassius auratus)[8]、鲤(Cyprinus carpio)[9]和团头鲂(Megalobrama amblycephala)[10], 这些研究主要集中在肌间骨的形态、数量和分布等规律。而关于肌间骨发育分子调控研究的仅仅有罗非鱼(Tilapia)[11]、鲤(Common carp)[11] 和团头鲂(Megalobrama amblycephala)[12]。
小分子RNA(microRNA, miRNA)是一类进化保守的、内源性的、非编码的小RNA。miRNA通过与靶基因的3′非翻译区(Untranslated regions, UTR)结合从而抑制靶基因的翻译或降解靶基因[13]。1993年在秀丽隐杆线虫(Caenorhabditis elegans)时序性发育调控过程中发现了首个miRNA(lin-4)[14]; 2000年又在线虫中找到了第2个调控时序性发育的miRNA基因let-7[15]。miRNA参与许多重要的生物学过程, 如在个体发育、组织分化、病毒感染等逆境适应过程中都具有重要作用。miRNA在鱼类的研究比较多地集中于免疫[16]方面。近年来, miRNA在鱼类发育过程中的作用也越来越受到重视, 鱼类骨骼要经历软骨化和硬骨化的过程, 受多种转录因子的调控, 如Sox9[17]和Runx2[18]等。而miR-140[19]、miR-204[20]和miR-211[20]则调控上述斑马鱼骨化转录因子的表达。以上结果表明, miRNA是调控鱼类骨骼形成的重要因素。研究发现, miRNA一般是通过多个信号通路来调控骨骼发育[21]。但目前为止, miRNA在水产动物肌间骨发育中的作用研究仅仅在团头鲂中开展过[22, 23]。
作为广西稻田养鱼的主要本地特色品种禾花鲤, 其相关的基础研究十分缺乏。本研究针对禾花鲤肌间骨细小且柔软可食用的特点, 开展其分子调控机制的研究。采集禾花鲤及建鲤的肌间骨进行miRNAs的高通量测序, 进行生物信息学比较分析, 为禾花鲤肌间骨发育的分子调控机制研究奠定基础。
1. 材料与方法
1.1 实验动物
本研究所用的实验动物为来自全州的禾花鲤及来自南宁的建鲤(均为24月龄), 均暂养在温度为25℃的水中。
1.2 组织样本采集
分别采集禾花鲤及建鲤的肌间刺数根, 于液氮中速冻。
1.3 小RNA文库构建和测序
用EASYspin Plus骨组织RNA快速提取试剂盒分别提取禾花鲤及建鲤骨头的总RNA, 随后通过15%变性聚丙烯酰胺凝胶电泳分离纯化16—30 nt的小分子RNA, 进行Solexa高通量测序。小RNA文库的Solexa测序由武汉华大生物公司完成。
1.4 生物信息学分析
一般来说, 小RNA的长度区间为18—30 nt。在测序完成后, 首先进行序列去杂, 筛选一定长度范围内的sRNA, 用软件bowtie (http://computing.bio.cam.ac.uk/local/doc/bowtie2.html#)将长度筛选后的sRNA定位到基因组参考序列上, 分析small RNA在参考序列上的分布情况, 然后将上述mapped到参考序列上的reads, 在Rfam (http://Rfam.sanger.ac.uk/)、miRBase (http://www.mirbase.org/)中进行比对, 得到各样品匹配上的sRNA的详细情况, 包括匹配上的已知miRNAs的二级结构, 各样本中miRNAs的序列、长度、出现的次数等信息。进一步通过miRNAs前体的标志性发夹结构, 使用miRDeep2[24]预测新的miRNAs。对各样本中已知和新miRNAs进行表达量的统计, 并用TPM进行表达量归一化处理。参照Audic等[25]发表在Genome Research上的基于测序的差异基因检测方法开发ExpDiff进行样品间表达差异分析。根据差异miRNAs检测结果, 使用R软件中的pheatmap函数进行层次聚类分析。我们用miRanda[26]来对分析得到的已知、novel miRNAs进行靶基因预测。得到各组比较间的差异表达miRNAs后, 根据miRNAs与其靶基因间的对应关系, 对每组差异表达miRNAs的靶基因的集合分别进行GO (Gene Ontology)和KEGG (Kyoto Encyclopedia of Genes and Genomes)富集分析。
2. 结果
2.1 小RNAs的基本特征
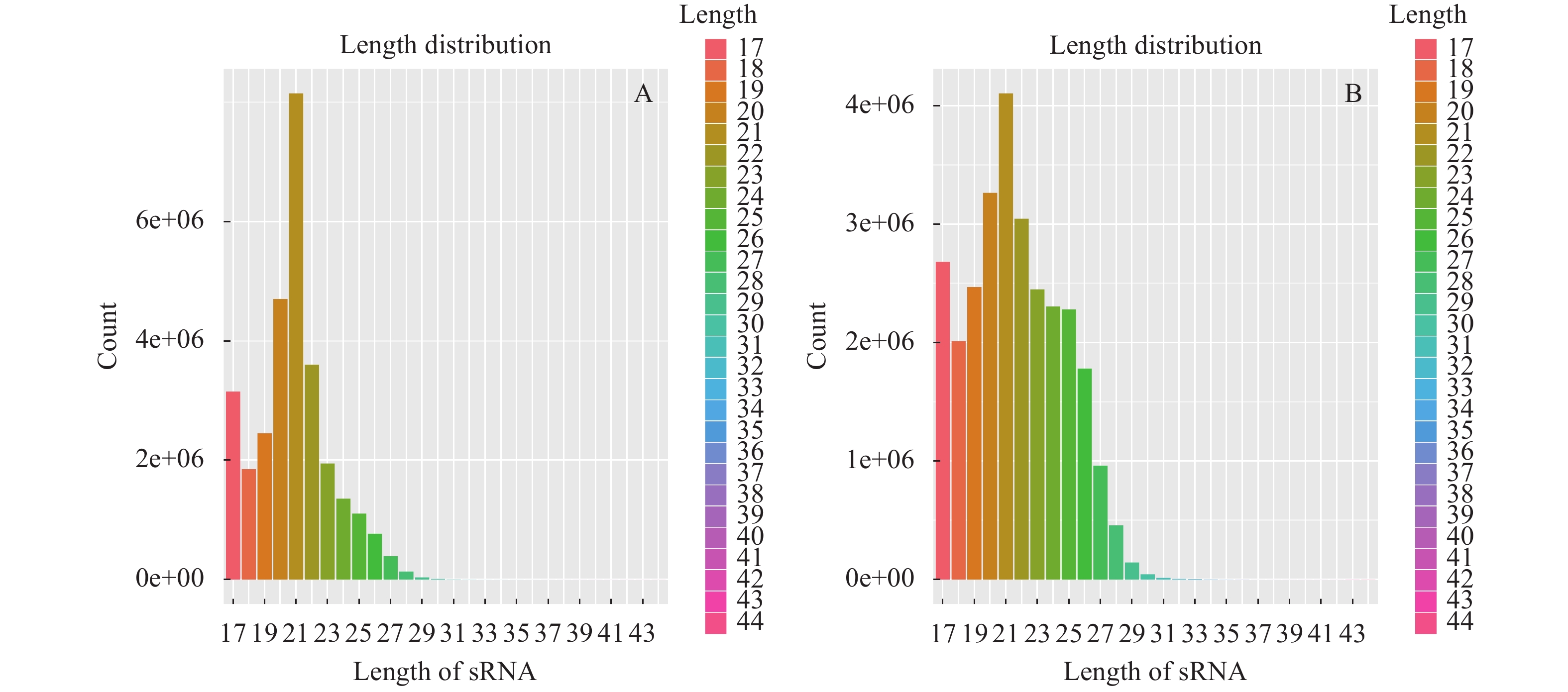
为了比较禾花鲤及建鲤肌间刺miRNAs的表达差异, 本研究分别从禾花鲤及建鲤肌间刺提取小RNA, 构建了2个小RNA文库。对这2个文库进行Solexa测序, 分别产生约30179426和29750080条序列。去除低质量序列、接头序列和短序列后, 分别从禾花鲤和建鲤的肌间刺文库中筛选出25474895和24625715条高质量序列进行进一步的分析。结果表明, 禾花鲤以及建鲤小RNA长度主要分布在17—30个核苷酸, 其中21个核苷酸的小RNA最丰富, 其次是20个核苷酸、22个核苷酸(图 1)。
2.2 MiRNAs的鉴定
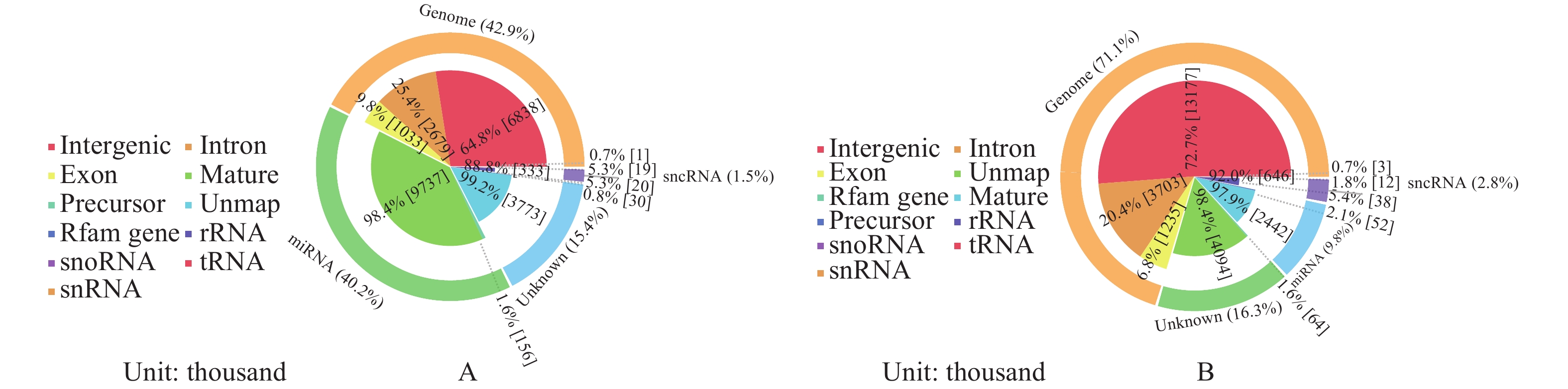
在上述高质量小RNA序列中, 禾花鲤与建鲤分别有20765041 (84.32%)和21326842 (83.72%)条序列与参考基因组比对成功。比对上的小RNA通过与Rfam、miRBase数据库比对, 进行各种小RNA分类, 其中, 禾花鲤miRNAs占其小RNA总量的9.80%, 远低于建鲤(40.2%, 图 2)。从禾花鲤和建鲤中分别鉴定出已知的成熟miRNAs 595和570种。
2.3 MiRNAs的差异表达分析
通过比较样品之间的表达量, 筛选差异表达miRNAs。我们采用ExpDiff进行差异miRNAs筛选。禾花鲤与建鲤相比, 一共有351个差异miRNAs, 其中84个上调表达, 267个下调表达。部分显著差异表达的miRNAs见表 1。
表 1 部分显著差异表达及成骨发育相关的miRNAsTable 1. Partial significantly differential expressed or osteogenesis-related miRNAsmiRNAs的名称Names of miRNAs 差异倍数(禾花鲤/建鲤)
log2 Ratio (Rice_flower_
carp/Jian_carp)成骨发育中的作用Role of human osteogenesis miR-135a-3p 8.13 抑制成骨[35] miR-133a-5p 7.57 抑制成骨[35] miR-138 2.8 抑制成骨[34] miR-34b-5p 1.66 抑制成骨[36] miR-148 2.18 抑制成骨[37] miR-29b –1.95 促进成骨[29] miR-130a –1.93 促进成骨[33] miR-199a –1.89 促进成骨[33] miR-21 –1.61 促进成骨[33] miR-27c-3p –1.5 促进成骨[31] miR-210-3p –1.5 促进成骨[32] 2.4 差异miRNAs的靶基因GO显著性富集分析
我们用WEGO软件对差异miRNA的靶基因做GO功能分类统计, 从宏观上认识差异基因的功能分布特征, 部分结果展示如表 2。GO总共分为三大功能类, 分别描述基因的分子功能(Molecular function)、所处的细胞位置(Cellular component)和参与的生物过程(Biological process)。差异miRNA的靶基因注释最多的为Biological process, 其中以细胞过程(Cellular process)为最多, 其次为单一组织过程(Single-organism process)、新陈代谢过程(Metabolic process)及生物调节(Biological regulation); Molecular function中以结合(Binding)及催化活性(Catalytic activity)为最多; Cellular component中以细胞(Cell)、细胞器(Organelle)为最多。
表 2 部分差异miRNAs靶基因的GO分类Table 2. Partial GO of differentially expressed miRNAsGO分类
GO categoriesGO条目
GO terms基因数量
Number of gene生物学过程
Biological process细胞过程Cellular process 15812 单一组织过程
Single-organism process14435 新陈代谢过程
Metabolic process11062 生物调节
Biological regulation9566 分子功能
Molecular function结合Binding 12682 催化活性
Catalyticactivity7245 细胞成分
Cellular component细胞Cell 13759 细胞器Organelle 13738 2.5 差异miRNAs的靶基因Pathway显著性富集分析
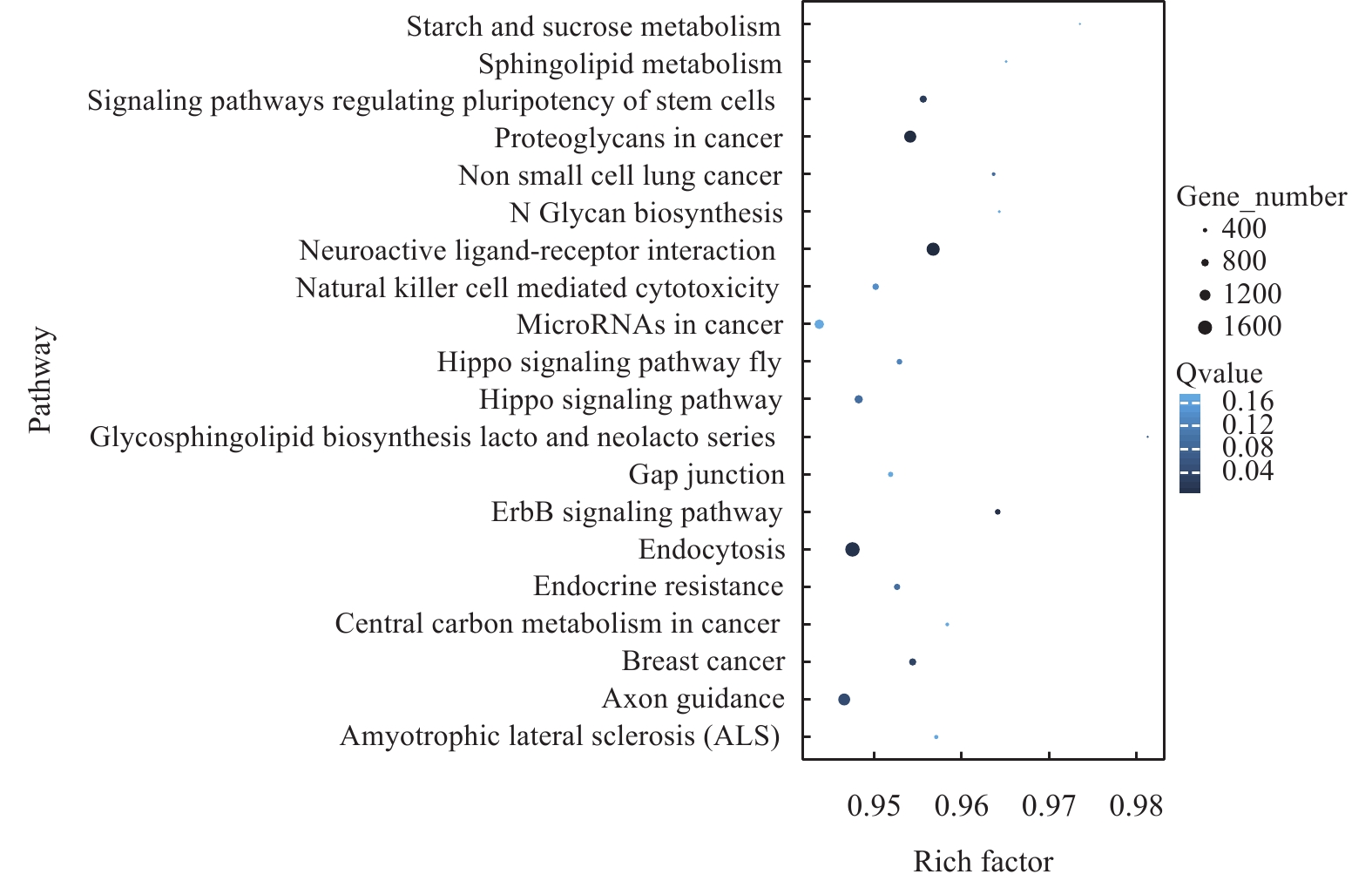
在生物体内, 不同基因相互协调行使其生物学功能, 基于Pathway的分析有助于更进一步了解基因的生物学功能。我们以KEGG公共数据库对差异miRNAs作Pathway富集分析。图 3显示了排名前20的差异miRNAs的靶基因富集的Pathway。其中包括signaling pathways regulating pluriotency of stem cells (调节干细胞多能性的信号通路)、axon guidance(轴突导向)等。
3. 讨论
Li等[27]发现在MC3T3细胞成骨分化过程中, miR-29b可以通过降解抑制成骨的靶基因HDAC4、TGF-β3、ACVR2A的表达促进成骨。团头鲂肌间骨中miR-29的表达量较结缔组织中的低, 推测miR-29在团头鲂中可能起到抑制成骨的作用[23]。miR-29虽然与miR-29b为一个家族的成员, 但功能却不同, 也可能是在不同物种中作用的差异。He等[28]发现miR-20b通过降低PPARγ的表达提高了成骨的核心转录因子Runx2转录, 促进成骨分化。有研究发现miR-27可以抑制靶基因APC, 从而反激活WNT促进成骨分化[29]。miR-210在ST2成骨分化过程中可能通过抑制成骨分化的重要基因AcvR1b来促进成骨[30]。Oskowitz等[31]则从由美国国立卫生研究中心提供的MSCs其诱导分化的成骨细胞中, 利用芯片筛选出在成骨细胞中高表达的miRNAs, 如hsa-miR-130a、has-miR-199a、hsa-miR-346、hsa-miR-21及hsa-miR-10a等。在本研究中禾花鲤肌间骨中miR-29b、20b-5p、miR-27-3p、miR-210-3p、miR-130a、miR-199a及miR-21的表达与建鲤相比均为下调表达。这些均为上述已报道的促进人类成骨的miRNAs, 因此我们推断禾花鲤通过下调表达促进成骨的miRNAs来抑制成骨。
Eskildscn等[32]发现miR-138通过抑制FAK-ERK1/2信号通路降低下游成骨的核心结合因子Runx2和OSX的表达来降低成骨作用。Li等[33]发现在BMP2诱导的C2C12细胞成骨分化中, 对其中显著下调的miR-133和miR-135进行研究后发现, miR-133的靶基因为Runx2, 而miR-135的靶基因为mad5, 两者共同作用来抑制骨的形成。团头鲂肌间骨中miR-133b的表达量随着鱼龄的增长呈增加的趋势, 推测miR-133b在团头鲂肌间骨的形成过程中起到促进的作用[28]。miR-133b虽然与miR-133为一个家族的成员, 但功能有所不同, 也可能是在不同物种中作用的差异。Wei等[34]发现, miR-34b通过两条途径来抑制成骨, 一方面通过阻碍CDK4、CDK6和Cyclin D1蛋白表达来减少成骨细胞的数量; 另一方面通过降低SATB2蛋白的表达来抑制成骨细胞的分化。Gao[35]发现hsa-miR-31、hsa-miR-106a、hsa-miR-148a通过抑制BMSCs的分化来抑制成骨的过程。在本研究中禾花鲤肌间骨中的miR-133、miR-135、miR-34b、hsa-miR-31、hsa-miR-148a以及miR-138的表达与建鲤相比均为上调表达。这些均为上述已报道的抑制人类成骨的miRNAs, 因此我们推断禾花鲤通过上调表达抑制成骨的miRNAs来达到抑制成骨的目的。
总之, 我们推测禾花鲤可能是通过下调表达促进成骨miRNAs及上调表达抑制成骨miRNAs来抑制成骨过程, 从而维持其肌间骨细小及柔软的特性。该研究为禾花鲤肌间骨发育的分子调控机制研究奠定了基础。
-
表 1 部分显著差异表达及成骨发育相关的miRNAs
Table 1 Partial significantly differential expressed or osteogenesis-related miRNAs
miRNAs的名称Names of miRNAs 差异倍数(禾花鲤/建鲤)
log2 Ratio (Rice_flower_
carp/Jian_carp)成骨发育中的作用Role of human osteogenesis miR-135a-3p 8.13 抑制成骨[35] miR-133a-5p 7.57 抑制成骨[35] miR-138 2.8 抑制成骨[34] miR-34b-5p 1.66 抑制成骨[36] miR-148 2.18 抑制成骨[37] miR-29b –1.95 促进成骨[29] miR-130a –1.93 促进成骨[33] miR-199a –1.89 促进成骨[33] miR-21 –1.61 促进成骨[33] miR-27c-3p –1.5 促进成骨[31] miR-210-3p –1.5 促进成骨[32] 表 2 部分差异miRNAs靶基因的GO分类
Table 2 Partial GO of differentially expressed miRNAs
GO分类
GO categoriesGO条目
GO terms基因数量
Number of gene生物学过程
Biological process细胞过程Cellular process 15812 单一组织过程
Single-organism process14435 新陈代谢过程
Metabolic process11062 生物调节
Biological regulation9566 分子功能
Molecular function结合Binding 12682 催化活性
Catalyticactivity7245 细胞成分
Cellular component细胞Cell 13759 细胞器Organelle 13738 -
[1] Kitanishi S, Onikura N, Mukai T. A simple SNP genotyping method reveals extreme invasions of non-native haplotypes in pale chub Opsariichthys platypus, a common cyprinid fish in Japan [J]. PLoS One, 2018, 13(1): e0191731 doi: 10.1371/journal.pone.0191731
[2] 单玲玲, 李小勤, 郑小淼, 等. 不同形式蛋氨酸对建鲤生长性能及血清游离氨基酸含量的影响. 水生生物学报, 2015, 39(2): 259—264 Shan L L, Li X Q, Zheng X M, et al. Effects of different forms of methionine on growth performance and serum free amino acids content of Jian carp [J]. Acta Hydrobiologica Sinica, 2015, 39(2): 259—264
[3] 朱双宁, 朱辰娴, 俞菊华, 等. 建鲤和荷包红鲤肌肉脂肪、脂肪酸组成以及Apo-C-I转录水平的比较. 水生生物学报, 2015, 39(1): 209—215 Zhu S N, Zhu C X, Yu J H, et al. Comparisons of muscle fat content, fatty acids compositions and transcriptional levels of Apo-C-I between Jian carp and Red purse carp [J]. Acta Hydrobiologica Sinica, 2015, 39(1): 209—215
[4] Patterson C, Johnson G D. The intermuscular bones andligaments of teleostean fishes [J]. Smithsonian Contributions to Zoology, 1995, 559: 1—85
[5] Danos N, Ward A B. The homology and origins ofintermuscular bones in fishes: phylogenetic orbiomechanical determinants [J]. Biological Journal of the Linnean Society, 2012, 106(3): 607—622 doi: 10.1111/bij.2012.106.issue-3
[6] 聂春红, 陈祖萱, 戴彩娇, 等. 不同鱼类肌间骨的骨化模式研究. 水生生物学报, 2018, 42(1): 131—137 Nie C H, Chen Z X, Dai C J, et al. Ossification patterns of intermuscular bones in different fishes [J]. Acta Hydrobio-logica Sinica, 2018, 42(1): 131—137
[7] 柯中和, 张炜, 蒋燕, 等. 鲢肌间小骨发育的形态学观察. 动物学杂志, 2008, 43(6): 88—96 doi: 10.3969/j.issn.0250-3263.2008.06.013 Ke Z H, Zhang W, Jiang Y, et al. Morphological observation on the development of intramuscular small bone in silver carp [J]. Chinese Journal of Zoology, 2008, 43(6): 88—96 doi: 10.3969/j.issn.0250-3263.2008.06.013
[8] 王静, 张俏, 赵琼, 等. 合方鲫肌间骨骨化过程及形态学观察. 湖南师范大学自然科学学报, 2017, 40(2): 39—43 Wang J, Zhang Q, Zhao Q, et al. Ossification process and morphology of intermuscular bones in hybrids crucian carp [J]. Journal of Natural Science of Hunan Normal University, 2017, 40(2): 39—43
[9] 陈琳, 田雪, 米佳丽, 等. 黄河鲤肌间骨发育的形态学观察. 上海海洋大学学报, 2017, 26(4): 481—489 Chen L, Tian X, Mi J L, et al. Morphological observation of intermuscular bone development in Cyprinus carpio haematopterus [J]. Journal of Shanghai Ocean University, 2017, 26(4): 481—489
[10] 万世明, 易少奎, 仲嘉, 等. 团头鲂肌间骨发育的形态学观察. 水生生物学报, 2014, 38(6): 1143—1148 Fang S M, Yi S K, Zhong J, et al. Morphological observation of muscular bone development in blunt snout bream [J]. Acta Hydrobiologica Sinica, 2014, 38(6): 1143—1148
[11] Su S G, Dong Z J, et al. Comparative expression analyses of bone morphogenetic protein 4(BMP4) expressions in muscles of tilapia and common carp indicate that BMP4 plays a role in the intermuscular bone distribution in a dose-dependent manner [J]. Gene Expression Patterns, 2018, 27: 106—113 doi: 10.1016/j.gep.2017.11.005
[12] Zhang W Z, Lan T, Nie C H, et al. Characterization and spatiotemporal expression analysis of nine bonemorphogenetic protein family genes during intermuscular bonedevelopment in blunt snout bream [J]. Gene, 2018, (642): 116—124
[13] 李法君, 李明爽, 付春鹏, 等. MicroRNA在水产动物中的研究进展. 水产学报, 2016, 40(6): 976—977 Li F J, Li M S, Fu C P, et al. Research progress of microRNA in aquatic animals [J]. Journal of Fisheries of China, 2016, 40(6): 976—977
[14] Lee R C, Feinbaum R L, Ambros V. The C. elegansheterochronic gene lin-4 encodes small RNAs withantisense complementarity to lin-14 [J]. Cell, 1993, 75(5): 843—854 doi: 10.1016/0092-8674(93)90529-Y
[15] Reinhart B J, Slack F J, Basson M, et al. The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans [J]. Nature, 2000, 403(6772): 901—906 doi: 10.1038/35002607
[16] 沙珍霞, 陈学杰, 陈亚东, 等. 半滑舌鳎miR-200a和miR-200b前体克隆及其免疫应答分析. 水生生物学报, 2017, 41(2): 314—319 Sha Z X, Chen X J, Chen Y D, et al. Cloning and immune response analysis of miR-200a and miR-200b precursors of Cynoglossus semilaevis [J]. Acta Hydrobiologica Sinica, 2017, 41(2): 314—319
[17] Yan Y L, Miller C T, Nissen R, et al. A zebrafish sox9 gene required for cartilage morphogenesis [J]. Development, 2002, 129(21): 5065—5079
[18] Flores M V, Lam E Y N, Crosier P, et al. A hierarchy of Runx transcription factors modulate the onset of chondrogenesis in craniofacial endochondral bones in zebrafish [J]. Developmental Dynamics, 2006, 235(11): 3166—3176 doi: 10.1002/(ISSN)1097-0177
[19] Nakamura Y, He X, Kato H, et al. Sox9 is upstream of micro RNA-140 in cartilage [J]. Applied Biochemistry and Biotechnology, 2012, 166(1): 64—71 doi: 10.1007/s12010-011-9404-y
[20] Huang J, Zhao L, Xing L, et al. MicroRNA-204 regulates Runx2 protein expression and mesenchymal progenitor cell differentiation [J]. Stem Cells, 2010, 28(2): 357—364
[21] Gradus B, Hornstein E. Role of microRNA in skeletondevelopment [J]. Bone and Development, 2010, (6): 81—91
[22] Wan S M, Yi S K, Zhong J, et al. Dynamic mRNA and miRNA expression analysis in response to intermuscular bone development of blunt snout bream (Megalobrama amblycephala) [J]. Scientific Reports, 2016, (6): 31050
[23] Wan S M, Yi S K, Zhong J, et al. Identification of MicroRNA for intermuscular bone development in blunt snout bream (Megalobrama amblycephala) [J]. International Journal of Molecular Sciences, 2015, 16(5): 10686—10703
[24] Friedlander M R, Chen W, et al. Discovering microRNAs from deep sequencing data using miRDeep [J]. Nature Biotechnology, 2008, (26): 407—415
[25] Audic S, Claverie J M. The significance of digital gene expression profiles [J]. Genome Research, 1997, (10): 986—995
[26] John B, Enright A J, Aravin A, et al. Correction: Human MicroRNA targets [J]. PLoS Biology, 2005, 3(7): e264 doi: 10.1371/journal.pbio.0030264
[27] Li Z, Hassan M Q, Jafferji M, et al. Biological functions of miR-29b contribute to positive regulation of osteoblast differentiation [J]. Journal of Biological Chemistry, 2009, 284(23): 15676—15684 doi: 10.1074/jbc.M809787200
[28] He J, Zhang J F, Yi C, et al. MiRNA-mediated functional changes through co-regulating function related genes [J]. PLoS One, 2010, 5(10): e13558 doi: 10.1371/journal.pone.0013558
[29] Wang T, Xu Z. miR -27 promotes osteoblast differentia-tion by modulating Wnt signaling [J]. Biochemical and Biophysical Research Communication, 2010, 402(2): 186—189 doi: 10.1016/j.bbrc.2010.08.031
[30] Mizuno Y, Tokuzawa Y, Ninomiya Y, et al. MiR-210 promotes osteoblastic differentiation through inhibition of Acv R1b [J]. Febs Letters, 2009, 583(13): 2263—2268 doi: 10.1016/j.febslet.2009.06.006
[31] Oskowitz A Z, Lu J, Penfornis P, et al. Human multipotent stromalcells from bone marrow and micrRNA: regulation of differentiationand leukemia inhibitory factor expression [J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(47): 18372—18377 doi: 10.1073/pnas.0809807105
[32] Eskildscn T, Taipalccnmaki H, Stcnvang J, et al. MicroRNA-138 regulates osteogenic differentiation of human stromal (mesenchymal) stem cells invivo [J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(15): 6139—6144 doi: 10.1073/pnas.1016758108
[33] Li Z, Hassan M Q, Volinia S, et al. A microRNA signature for a BMP2-induced osteoblast lineage commitment program [J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(37): 13906—13911 doi: 10.1073/pnas.0804438105
[34] Wei J, Shi Y, Zheng L, et al. MiR-34s inhibit osteoblast proliferation and differentiation in the mouse by targeting SATB2 [J]. Journal of Cell Biology, 2012, 197(4): 509—521 doi: 10.1083/jcb.201201057
[35] Gao J, Yang T, Han J, et al. MicroRNA expression during osteogenic differentiation of human multipotent mesenchymal stromal cells from bonemarrow [J]. Journal of Cell Biochemistry, 2011, 112(7): 1844—1856 doi: 10.1002/jcb.23106
-
期刊类型引用(7)
1. 何昌熙,郑建波,马建波,贾永义,刘士力,蒋文枰,迟美丽,程顺,李飞. 翘嘴鲌Runx2b基因的克隆与表达特征分析. 浙江农业学报. 2024(05): 1024-1031 .  百度学术
百度学术
2. 董传举,贾颖颖,申亚伟,李学军. 黄河鲤肌间骨转录组分析及斑马鱼bmp1基因敲除模型构建. 河南师范大学学报(自然科学版). 2024(04): 71-80 .  百度学术
百度学术
3. 纪达,杨玉梅,姚俊杰,王益海,张书海. 贵州7个鲤养殖群体遗传多样性与遗传结构分析. 海洋渔业. 2023(03): 257-266 .  百度学术
百度学术
4. 陈洁,吕耀平,戴庆敏,张乐,徐传保,陆君. 唇?bmp2a基因克隆及其与肌间刺骨化相关性分析. 水生生物学报. 2021(01): 8-13 .  本站查看
本站查看
5. 龚葭薇,何梅,闫学春,孔德麟,梁洋. 斑马鱼和鲤miR-1-2/133a-1基因间序列活性和调控研究. 水生生物学报. 2021(03): 507-513 .  本站查看
本站查看
6. 王旭东,聂春红,高泽霞. 鱼类肌间骨发育分子调控机制及遗传选育研究进展. 水生生物学报. 2021(03): 680-691 .  本站查看
本站查看
7. 李福贵,程林慧,韩雨晴,张士林,何坤,孙艺文,吴华东,刘凯月,段晓迪,董传举. 江西3个地理群体野生鲫肌间骨的比较分析. 江西农业大学学报. 2020(04): 755-765 .  百度学术
百度学术
其他类型引用(6)



 下载:
下载: