MOLECULAR CHARACTERISTICS, GENOMIC STRUCTURE AND EXPRESSION PATTERNS OF DIVERSE BMP15 ALLELES IN POLYPLOID GIBEL CARP CLONE F
-
摘要: 为探究骨形态发生蛋白15(Bone morphogenetic protein 15, bmp15)在银鲫(Carassius gibelio Bloch) F系卵巢发育过程中的表达特征, 研究采用cDNA末端快速扩增(RACE)方法首先从银鲫F系卵巢cDNA中克隆了2个bmp15部分同源基因(Homeologs), 将其命名为Cgbmp15a和Cgbmp15b, 它们各自具有3个不同等位基因。不同脊椎动物Bmp15蛋白序列多重比对和系统进化树均表明, 银鲫为异源六倍体, 在其进化历程中发生了两轮多倍化, 其中早期的异源多倍化整合了来自2个原始祖先的染色体组, 导致银鲫和鲫的四倍体共同原始祖先基因组中包含bmp15a和bmp15b; 随后第二轮同源多倍化最终导致银鲫存在6个bmp15等位基因。bmp15基因及其邻近基因同线性分析表明, 银鲫在形成异源多倍体后, 其基因组发生了复杂的变化, 包括bmp15邻近基因的丢失。Cgbmp15a和Cgbmp15b均主要在卵巢中表达, 在皮质泡期卵母细胞中表达量最高; 无论是在银鲫成体组织还是不同发育阶段的卵子中, Cgbmp15a的表达水平显著高于Cgbmp15b的表达水平。在孕酮激素DHP体外诱导银鲫F系GV1期卵母细胞6h后, Cgbmp15a开始上调表达, 诱导8h后达到最高表达水平, 随后下降; 而Cgbmp15b在诱导8h后表达水平有微弱上调, 随后下降。上述的结果表明, Cgbmp15a和Cgbmp15b在银鲫F系成体组织和不同发育和成熟阶段的卵子中, 均表现出偏性表达的特征, 暗示Cgbmp15a在银鲫的卵母细胞发育和成熟中起着主要作用。Abstract: Bone morphogenetic protein 15 (bmp15) plays key roles in regulating development and function maintenance of reproductive system in vertebrates. In this study, we first identified two divergent bmp15 homeologs from gibel carp (Carassius gibelio Bloch) clone F. Both Cgbmp15a and Cgbmp15b possess 3 different alleles, which were obtained from ovary cDNA by using rapid-amplification of cDNA ends (RACE). Multiple sequence alignment of Bmp15 protein in vertebrates and phylogenetic analysis of gibel carp, crucian carp, and zebrafish bmp15 genomic sequences indicated that two rounds of polyploidization had been occurred during allo-hexaploid gibel carp evolution. The early polyploidy produced the two divergent bmp15a and bmp15b, and the late polyploidy generated the 6 Cgbmp15 alleles of gibel carp. Synteny analysis of genomic regions around bmp15 gene in vertebrates suggested that the complex genomic changes might occur after the polyploidization in gibel carp, such as the loss of bmp15 adjacent genes. Cgbmp15a and Cgbmp15b were predominantly expressed in ovary with the highest level in cortical alveolus oocytes. The Cgbmp15a level is remarkably higher than Cgbmp15b in adult tissues and oocytes at different developmental stages (P<0.05). Moreover, Cgbmp15a increased at 6 hours and reached peak level at 8 hours after DHP induction in vitro, while Cgbmp15b slightly increased at 8 hours after DHP induction. These results indicate that Cgbmp15a play a major role in gibel carp oocyte development and maturation.
-
Keywords:
- Gibel carp /
- Allo-hexaploid /
- Bmp15 /
- Oogenesis and oocyte maturation /
- Biased expression
-
骨形态发生蛋白信号通路(Bone morphogenetic protein signaling, BMP signaling)在动物生殖系统的发育和功能维持的多个生命过程中发挥着重要的调控作用, 包括原始生殖细胞特化、增殖和迁移, 配子发生、滤泡形成等[1]。其中, BMP15主要由卵母细胞分泌, 首先在人和小鼠中被克隆[2], 后被证实为脊椎动物所特有[3], 在哺乳动物的滤泡发育和闭锁、产卵、繁殖力维持以及授精等过程中起着关键作用[4—10]。
关于鱼类bmp15基因研究得较少, 近年来仅在斑马鱼(Danio rerio)[11]、欧洲鲈鱼(Dicentrarchus labrax)[12, 13]、银鲫(Carassius gibelio)[14]、稀有鲫(Gobiocypris rarus)[15]、黄尾(Seriola lalandi)[16]、黑鲷(Acanthopagrus schlegelii)[17]、大西洋鲑(Salmo salar L.)[18]和胡鲇(Clarias batrachus)[19]中报道了bmp15的克隆和表达分析。对这些鱼类bmp15基因的表达分析表明, bmp15基因不仅在成体组织的分布, 而且在不同发育阶段性腺和胚胎中的表达谱式以及在卵巢中的定位都存在差异, 表现出物种的特异性。譬如, 在斑马鱼、欧洲鲈鱼、银鲫和稀有鲫中, 除了在卵巢中高表达和精巢中中等丰度的表达外, 在肝、肾、脑、垂体等多个组织中也有低丰度的表达[11, 12, 14, 15]; 而大西洋鲑bmp15-like仅在性腺中表达, 在其他组织中检测不到[18]。不同于哺乳动物和其他鱼类bmp15的报道[2, 12, 14, 15, 20—22], 斑马鱼bmp15不仅在卵子中表达, 还在颗粒细胞和鞘细胞中检测到了mRNA和蛋白的存在[11]。
多倍化(Polyploidization)是指基因组中加入了一套或多套完整的染色体组。除了在脊椎动物进化历程中均发生的两轮全基因组复制外, 辐鳍鱼类还发生了第3轮鱼类特异的全基因组复制(Fish-specific genome duplication, Ts3R)[23—25]。在突破不稳定的瓶颈后, 多倍体会通过随后的二倍化进化成一个新的二倍体。在“多倍化-二倍化”的进化历程中, 多倍体通过重复基因的新功能化或亚功能化、基因组的重排、基因的表达变异、等位基因的特异沉默、表观遗传的重塑等创造了大量的进化机会, 增加了等位基因的多样性, 改变了基因组的复杂性, 产生了新的性状并驱动改变生态位和扩大地理分布[26—31]。多倍体银鲫通常被视为鲫(Carassius auratus L.)的一个亚种(C. auratus gibelio Bloch)[32], 但随着对其核型、生殖方式、性别决定方式等研究的深入, 已逐渐被视为一个独立的物种[33-40]。银鲫拥有156条染色体或162条染色体[41], 由于其具有100条染色体的近缘种金鱼已被证明发生了四倍化事件[42], 因此银鲫应为超三倍体或进化的六倍体[43]。通过对银鲫2个歧化的Dmrt1基因的序列和染色体定位分析揭示了银鲫进化历程中发生了两轮多倍化事件[44]。尽管已在银鲫中鉴定了一个bmp15基因[14], 但对银鲫Dmrt1基因[44]、头肾组织的转录组[45]以及干扰素系统基因的分析[46]均表明, 银鲫是一个异源多倍体, 一个基因在其基因组中至少存在2个高度分化的等位基因。除了已鉴定的bmp15基因外, 银鲫是否还存在其他bmp15等位基因, 它们在表达上是否发生了明显歧化, 目前尚不清楚。因此, 本研究克隆鉴定了2个歧化的bmp15基因Cgbmp15a和Cgbmp15b的6个等位基因, 分析了它们的基因组结构和与其邻近基因的共线性关系, 以及Cgbmp15a和Cgbmp15b在成体组织、不同发育阶段卵子和孕酮激素DHP诱导卵母细胞成熟过程中的表达差异, 为研究多倍体鱼类重复基因的进化和bmp15在鱼类生殖调控中的作用提供基础数据。
1. 材料与方法
1.1 实验材料
本研究所用的银鲫F系选自于中国科学院水生生物研究所官桥实验基地。繁殖季节随机抽取3条性成熟的银鲫F系, 取肝、脾、肾、脑、下丘脑、垂体和卵巢组织部分样品; 并将卵巢置于盛有生理盐水的培养皿中, 在显微镜下利用机械分离法获得不同发育时期的卵细胞, 根据直径大小进行分类, 分为初级卵母细胞(0.06—0.13 mm, Ⅰ期)、皮质泡期卵母细胞(0.21—0.23 mm, Ⅱ期)、卵黄形成期卵母细胞(0.30—0.33 mm, Ⅲ1期和0.45—0.48 mm, Ⅲ2期)、成熟卵母细胞(1.04—1.22 mm, Ⅳ期)。将各样品放入无RNA酶的EP管中, 迅速经液氮速冻后置于–80℃备用。
1.2 总RNA提取和SMARTer cDNA模板合成
所用离心管、枪头、溶液均无RNA酶, 玻璃器皿及金属器械经240℃高温烘烤4h后使用。按照Trizol和SV Total RNA Isolation System试剂盒(Promega)说明书进行各样品总RNA的提取, 并分别用Nanodrop 2000C(Thermo Fisher Scientific, 美国)分光光度计和1.2%(w/v)琼脂糖凝胶电泳检测RNA的浓度和完整性。用SMARTer RACE 5′/3′ Kit(Clontech, 美国)合成了银鲫F系卵巢组织3′ cDNA和5′ cDNA, 具体参考Xie等[47]描述的方法。合成的cDNA置于–20℃保存。
1.3 银鲫F系Cgbmp15a和Cgbmp15b cDNA全长克隆
以斑马鱼bmp15的基因组序列为搜索序列, 在银鲫F系基因组中搜寻到了2个同源性较高的序列ENSDARP00000054590-C1和ENSDARP 00000054590-C2, 分别命名为Cgbmp15a和Cgbmp15b。参照这2个序列, 利用Primer Premier 5和Oligo7.0软件设计RACE内外引物(表 1)。以卵巢3′ cDNA和5′ cDNA为模板, 使用RACE外引物Cgbmp15a-5′-Outer、Cgbmp15b-5′-Outer、Cgbmp15a-3′-Outer、Cgbmp15b-3′-Outer与UPM分别进行5′或3′第一次PCR, 电泳检测首次PCR产物若无目的条带, 将首次PCR产物稀释100倍后为模板, 用相应的RACE内引物Cgbmp15a-5′-Inner、Cgbmp15b-5′-Inner、Cgbmp15a-3′-Inner、Cgbmp15b-3′-Inner和NUP进行巢式PCR, 2次PCR的反应体系和反应程序均参见文献[48]。目的条带经1.1%琼脂糖凝胶电泳采用Gel Extraction Kit(Omega Bio-Tek, 美国)回收后, 产物连接pMD18-T载体(TaKaRa), 各选取20个阳性克隆由铂尚生物技术(上海)有限公司进行双向测序, 序列经拼接后, 得到Cgbmp15a和Cgbmp15b的cDNA全长序列。
表 1 本文中所用引物Table 1. Primers used in this study引物名称Primer 序列Sequence (5′-3′) 应用Usage Cgbmp15aF GGTCACGACTGTCTGAGA BAC克隆筛选 Cgbmp15aR GAGTCTGCAATCATGTCTTC Cgbmp15bF CTACCAGCAGTCACACCT Cgbmp15bR GAGTCTGCAATCATGTCTTC Cgbmp15a-5′-Outer TATACAGGATAGCGGTGGAT RACE Cgbmp15a-5′-Inner ACTGTAAGGCTGCTTCAAG Cgbmp15b-5′-Outer GGCGTCACCAATACAGAA Cgbmp15b-5′-Inner ACAAGTTATTCGGCTCCAA Cgbmp15a-3′-Outer GAGTCTGCAATCATGTCTTC Cgbmp15a-3′-Inner GCAGGATACGAGGACAATC Cgbmp15b-3′-Outer GAGTCTGCAATCATGTCTTC Cgbmp15b-3′-Inner CTCCATCATCAGCACACT UPM CTAATACGACTCACTATAGGGCAAGCAGTGGTATCAACGCAGAGT NUP CTAATACGACTCACTATAGGGC Cgbmp15a QF GGGTCCAACACTGTAAGGC RT-qPCR Cgbmp15a QR CACCAATTTGTCCAACGAG Cgbmp15b QF TTATTCGGCTCCAACACTG Cgbmp15b QR TGTCCAACGAGAGGTTTTCC β-actin-QF AGCACGGTATTGTGACTAACTG β-actin-QR TCGAACATGATCTGTGTCATC 1.4 银鲫F系Cgbmp15a和Cgbmp15b基因序列分析与进化分析
根据已获得的Cgbmp15a和Cgbmp15b全长cDNA序列, 应用DNAMAN预测开放阅读框(ORF), 推导其编码的氨基酸序列, 并在Ensembl genome browser 92(http://asia.ensembl.org/index.html)和NCBI(https://www.ncbi.nlm.nih.gov/)上下载多个物种的Bmp15氨基酸序列, 应用BioEdit软件进行Bmp15多重序列比对分析。不同物种Bmp15氨基酸的登录号如下: 银鲫Carassius gibelio Bmp15(ADW20149.1)、鲫Carassius auratus Bmp15a-1(MH297584)、鲫Carassius auratus Bmp15a-2(MH297583)、鲫Carassius auratus Bmp15b(MH297582)、斑马鱼Danio rerio Bmp15(AAI24107.1)、稀有鲫Gobiocypris rarus Bmp15 (AHK22790.1)、河鲀Takifugu rubripes Bmp15(ENSTRUP00000017355)、罗非鱼Oreochromis niloticus Bmp15(XP_003457772.1)、青鳉Oryzias latipes Bmp15(XP_004079992.1)、雀鳝Lepisosteus oculatus Bmp15(XP_006632858.2)、爪蟾Xenopus tropicalis Bmp15(XP_002936834.2)、鸡Gallus gallus BMP15(ENSGALP00000007547)、小鼠Mus musculus BMP15(AAH55363.1)和人Homo sapiens BMP15(EAW89914.1)。
以Cgbmp15a和Cgbmp15b cDNA序列为基础, 在其ORF区域设计引物来进行银鲫F系BAC文库筛选。筛选到的BAC序列目的片段经测序验证后, 利用同源法注释对BAC上的基因进行预测, 进行bmp15的基因组结构和与其邻近基因的同线性关系分析。用MEGA 6.06软件, 采用最大似然法(ML法)对银鲫bmp15、鲫bmp15和斑马鱼bmp15的基因组序列进行系统进化树构建。
1.5 Cgbmp15a和 Cgbmp15b在银鲫F系不同组织以及不同时期卵母细胞中的表达
选取样品总RNA 0.1 μg, 利用GoldScript cDNA合成试剂盒(invitrongen, 上海)反转录得到cDNA, 具体步骤参见文献[49]。使用Primer Premier 5.0设计Cgbmp15a和Cgbmp15b基因RT-qPCR特异性引物(表 1)。选取异育银鲫β-actin作为内参基因, 用CFX96TM Optics Module(美国Bio-Rad公司)进行荧光定量PCR, 反应条件如下: 95℃预变性30s; 95℃变性5s, 60℃, 30s, 40cycles; 65℃, 5s; 95℃, 5s。进行3次生物学重复, 并每个样品做3次技术性重复, 以β-actin作为内参引物, 序列参见文献[48], 用2–ΔΔCt方法计算基因的相对表达量, 采用平均值±标准误(Mean±SEM)来表示。数据分别经过正态性检验和方差齐性检验后, 采用SPSS 21.0软件进行方差分析, P<0.05表示存在显著性差异, 柱形图用GraphPad Prism 6进行绘制。
1.6 Cgbmp15a和 Cgbmp15b在DHP体外诱导银鲫F系卵母细胞成熟过程中的表达分析
在繁殖季节取性成熟的银鲫F系雌性个体, 用镊子将卵母细胞从卵巢中剥离出, 挑取处于生发泡1期(Germinal vesicle 1 stage, GV1期)卵母细胞, 转移至预热(21—23℃)的卵母细胞培养液格氏平衡盐溶液(Gey’s balanced salt solution, GBSS, 7.25 g NaCl, 0.41 g NaH2PO4, 0.38 g KCl, 0.95 g HEPES, 1.0 g葡萄糖, pH8.5, 0.147 g CaCl2, 0.23 g MgSO4, 0.1 g 1000 U青霉素, 0.1 g硫酸链霉素, 配制1 L)中培养。再向培养液中加入孕酮激素(17α, 20β-dihydroxy-4-pregnen-3-one, DHP, 终浓度为1 μg/mL), 每毫升培养液培养10颗卵母细胞, 23℃培养诱导卵母细胞体外成熟[50]。每隔2h取样1次, 每次取30颗卵, 利用Trizol和SV Total RNA Isolation System试剂盒说明书进行各样品总RNA的提取、反转录cDNA以及qRT-PCR检测Cgbmp15a和Cgbmp15b基因的表达。扩增条件和数据分析同上。
2. 结果
2.1 银鲫F系Cgbmp15a和Cgbmp15b的3个不同等位基因的分子特征
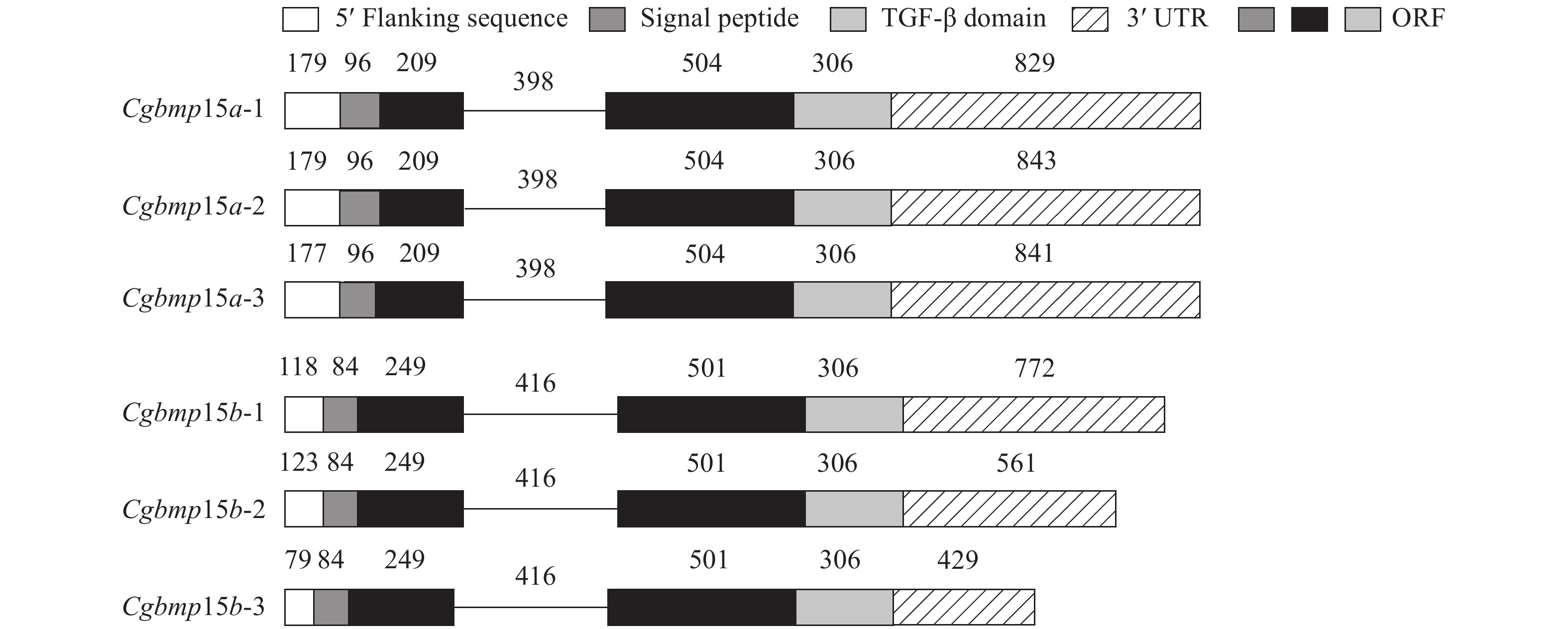
根据银鲫F系基因组中2个与斑马鱼bmp15基因的同源序列设计特异引物, 通过RACE-PCR各获得了Cgbmp15a和Cgbmp15b的3个全长cDNA序列。其中Cgbmp15a的3个全长cDNA大小分别为2133、2147和2143 bp, 它们之间的平均一致性为(98.73±0.35)%; 它们的开放阅读框(ORF)均为1128 bp, 编码375个氨基酸, 平均一致性为(99.29±0.13)%, 它们在第152、第157、第164和第286位氨基酸存在差异(图 1)。而Cgbmp15b的3个全长cDNA存在较大的差异, 大小分别为2030、1824和1648 bp, 它们之间的一致性为(84.88±4.40)%, 差异主要在3′端; 它们的开放阅读框均为1143 bp, 编码380个氨基酸, 平均一致性为(99.3±0.33)%, 在第167、第203、第268和第359位氨基酸存在差异(图 1)。Cgbmp15a和Cgbmp15b之间cDNA的一致性为(73.85±6.25)%, 氨基酸的一致性为(87.36±0.17)%。
不同物种Bmp15蛋白序列比对结果显示, 本研究克隆的CgBmp15a和CgBmp15b与已报道的银鲫Bmp15[14]的一致性分别为(92.13±0.12)%和(94.20±0.12)%, 表明已报道的银鲫Bmp15跟CgBmp15b同源性更高。同时我们在四倍体鲫基因组中搜索到3个bmp15等位基因, 分别命名为Cabmp15a-1、Cabmp15a-2和Cabmp15b; 在斑马鱼、稀有鲫、河鲀、罗非鱼、青鳉和雀鳝等其他鱼类基因组中, 均只搜索到一个bmp15基因。CgBmp15a和CgBmp15b与其他鱼类Bmp15的平均一致性分别在35.50%—80.52%和36.55%—81.82%, 而与人、小鼠、鸡和爪蟾Bmp15的一致性在28.27%—41.52%和29.30%—32.48%。
2.2 银鲫F系Cgbmp15a和Cgbmp15b的不同等位基因的基因组结构和相邻基因共线性分析
我们将所克隆到的6个银鲫bmp15等位基因序列在银鲫F系的BAC文库中进行筛选, 并将筛选到的BAC克隆的全序列与6个cDNA序列进行比对, 分别获得6个含银鲫bmp15 cDNA序列的BAC克隆。Cgbmp15a的3个BAC克隆的大小分别为122.75、145.62和106.83 kb, Cgbmp15b的3个BAC克隆的大小分别为90.06、151.96和35.05 kb。我们对6个BAC克隆上的基因进行了预测, 并分析了银鲫6个bmp15等位基因的基因组结构。研究结果表明, Cgbmp15a和Cgbmp15b的3个不同等位基因具有相同的基因组结构, 均由两个外显子和一个内含子组成(图 1)。Cgbmp15a的3个等位基因的两个外显子长度均分别为315和810 bp, 内含子大小为398 bp; Cgbmp15b的3个等位基因的2个外显子长度均分别为333和807 bp, 内含子为416 bp; 通过SMART(http://smart.embl-heidelberg.de/)软件分析表明这6个Bmp15蛋白都含有一个保守的TGF-β结构域, 且N端都含有一个信号肽。其中CgBmp15a的信号肽处于N端1—32位, TGF-β结构域位于第274—375位氨基酸; 而CgBmp15b的信号肽处于N端1—28位, TGF-β结构域位于第279—380位氨基酸。
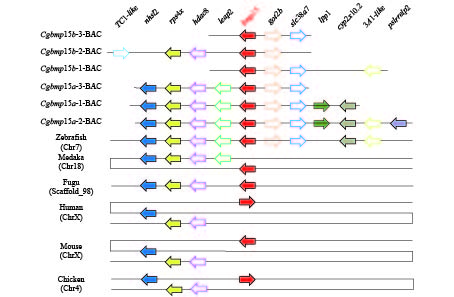
接着, 我们比较了6个BAC克隆上与其他邻近基因的同线性关系(图 2)。Cgbmp15a-1-BAC、Cgbmp15a-2-BAC和Cgbmp15a-3-BAC均含有NHS-like 2(nhsl2)、ribosomal protein S4、X-linked(rps4x)、histone deacetylase 8(hdac8)、liver-expressed antimicrobial peptide 2(leap2)、bmp15、glutamic-oxaloacetic transaminase 2b(got2b)、solute carrier family 38、member 7(slc38a7)基因, 且具有保守的共线性关系。由于Cgbmp15a-2-BAC比Cgbmp15a-1-BAC和Cgbmp15a-3-BAC分别长22.87和38.79 kb, 因此还包含lysosomal protective protein-like(lppl)、cytochrome P450, family 2、subfamily X、polypeptide 10.2(cyp2x10.2)、3A1-like、pollen-specific leucine-rich repeat extensin-like protein 2 (pslr relp2)基因。而Cgbmp15b的3个BAC克隆序列长度和包含的序列差异较大, 除了bmp15基因外, 还均含有got2b基因。与Cgbmp15b-1-BAC和Cgbmp15b-3-BAC相比, Cgbmp15b-2-BAC缺失了slc38a7基因。与Cgbmp15a的3个BAC序列相比, Cgbmp15b-2-BAC的bmp15上游还存在Tc1-like基因, 但缺失了nhsl2、leap2和slc38a7基因; 而Cgbmp15b-1-BAC的bmp15下游还包含3A1-like基因, 但缺失了lppl和cyp2x10.2基因。同时, 我们在Ensembl数据库获得了人、小鼠、鸡、斑马鱼、青鳉和河鲀的bmp15基因所在区域的基因组序列, 与银鲫bmp15基因所在区域进行同线性分析。结果表明, 斑马鱼bmp15基因及其8个邻近基因与银鲫 Cgbmp15a和邻近基因的同线性关系保守性最强, 仅在bmp15下游缺失了lppl和pslrrelp2基因。而其他脊椎动物bmp15基因下游未预测到与银鲫Cgbmp15a的相同下游基因。与银鲫Cgbmp15a-BAC、斑马鱼和青鳉相比, 人、小鼠、鸡和河鲀bmp15基因上游还缺失了leap2基因。
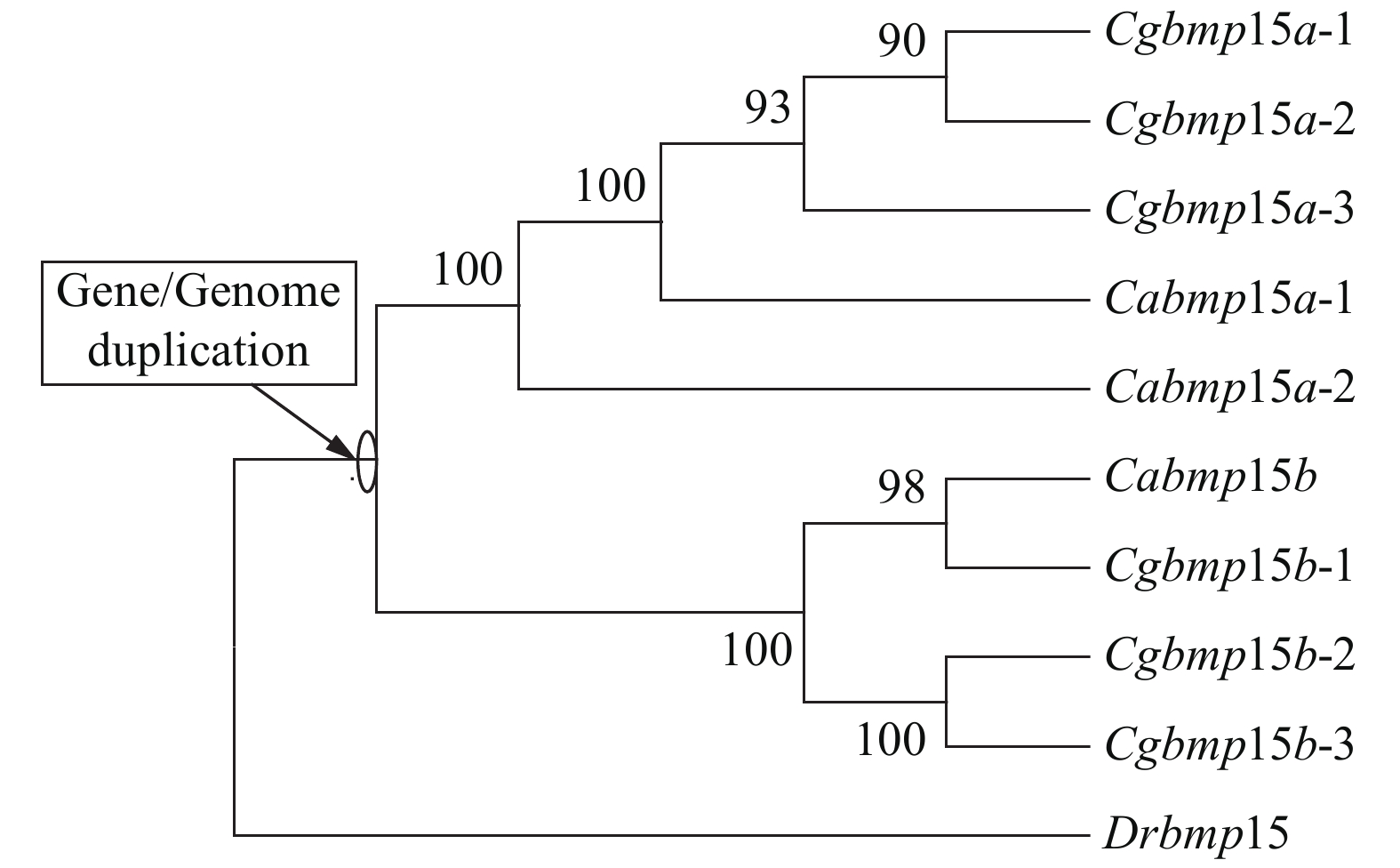
为了揭示银鲫6个bmp15等位基因的进化关系和起源, 我们以斑马鱼bmp15、3个鲫bmp15等位基因、6个银鲫bmp15等位基因的基因组全序列, 采用最大似然法(ML法)构建了系统进化树(图 3)。从图中我们可以看出, 斑马鱼Drbmp15作为外群, 银鲫Cgbmp15a的3个等位基因与鲫的Cabmp15a的2个等位基因聚为一簇, 银鲫Cgbmp15b的3个等位基因与鲫的Cabmp15b聚为另一簇, 而不是同一物种bmp15的不同等位基因聚在一起。由此我们推测银鲫和鲫拥有共同的原始祖先, 并且在图 3中标注出了在祖先家系中基因/基因组复制事件。
2.3 银鲫F系Cgbmp15a和Cgbmp15b在卵巢中高丰度表达
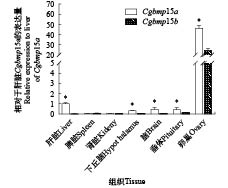
由于Cgbmp15a和Cgbmp15b的3个不同等位基因之间的一致性非常高, 因此我们针对Cgbmp15a和Cgbmp15b之间的序列差异, 设计2对特异引物, 采用RT-qPCR方法检测Cgbmp15a和Cgbmp15b在银鲫F系成体组织中的分布。结果表明, 银鲫F系Cgbmp15a和Cgbmp15b均在卵巢中高表达, 并且Cgbmp15a的表达水平显著高于Cgbmp15b(P<0.05)。Cgbmp15b在垂体中可检测到微量转录本的存在, 而在肝、脾、肾、脑和下丘脑中检测不到转录本的存在。与Cgbmp15b不同的是, 除了在垂体中外, Cgbmp15a还可以在肝、脑和下丘脑中检测到低丰度转录本的存在(图 4)。
![]() 图 4 Cgbmp15a和Cgbmp15b mRNA在银鲫不同成体组织中的表达分析以β-actin为内参; Mean±SD (n=3); *表示Cgbmp15a和Cgbmp15b在同一组织中表达水平具有显著性差异(P<0.05); 下同Figure 4. Relative expression of Cgbmp15a and Cgbmp15b in different adult tissuesβ-actin was used as control. Each bar represents mean ± standard deviation (SD, n=3). Asterisks (*) indicate significant differences (P<0.05) between Cgbmp15a and Cgbmp15b expression in same tissue. The same applies below
图 4 Cgbmp15a和Cgbmp15b mRNA在银鲫不同成体组织中的表达分析以β-actin为内参; Mean±SD (n=3); *表示Cgbmp15a和Cgbmp15b在同一组织中表达水平具有显著性差异(P<0.05); 下同Figure 4. Relative expression of Cgbmp15a and Cgbmp15b in different adult tissuesβ-actin was used as control. Each bar represents mean ± standard deviation (SD, n=3). Asterisks (*) indicate significant differences (P<0.05) between Cgbmp15a and Cgbmp15b expression in same tissue. The same applies below2.4 银鲫F系Cgbmp15a和Cgbmp15b随着DHP体外诱导卵母细胞成熟上调表达水平
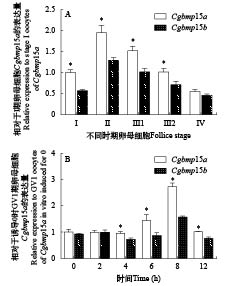
由于Cgbmp15a和Cgbmp15b在银鲫卵巢中高表达, 随后我们分析了它们在不同发育阶段卵母细胞中的表达水平差异。结果表明, 随着卵母细胞的发育和成熟, Cgbmp15a和Cgbmp15b表达水平均呈先升高再降低的趋势; 在皮质泡期卵母细胞中表达量达到最高, 随后降低; 且在初级卵母细胞到卵黄形成期卵母细胞中, Cgbmp15a的表达量始终显著高于同时期Cgbmp15b的表达量(P<0.05); 在成熟卵母细胞中二者无显著性差异(图 5A)。
接着, 我们采用DHP体外诱导银鲫F系GV1期卵母细胞成熟, 并分析了Cgbmp15a和Cgbmp15b在诱导成熟过程中的动态变化。结果表明, Cgbmp15a在诱导6h后开始上调表达, 在诱导8h后达到最高表达水平, 随后下降。而Cgbmp15b在诱导8h后表达水平有微弱上调, 随后下降。在诱导后6—12h, Cgbmp15a表达量均显著高于同时期Cgbmp15b的表达量(P<0.05, 图 5B)。
3. 讨论
在本研究中, 我们首先从银鲫F系中克隆了2个歧化的bmp15基因Cgbmp15a和Cgbmp15b, 它们各自具有一致性较高的3个不同等位基因。在鱼类的进化历程中, 除大部分鱼类共同经历的Ts3R外[23-25], 多倍化是一个重要且频繁发生的现象。多倍化根据染色体组的来源差异分为同源多倍化和异源多倍化, 其中, 异源多倍体通过种内或种间杂交产生。对鲤全基因组序列的解析表明在820万年前发生了一次全基因组复制, 形成了异源四倍体[51]。对银鲫2个歧化的Dmrt1基因的序列分析表明, 银鲫在额外两轮多倍化历程中, 经过大约1849万年前的一次早期多倍化事件, 形成了银鲫、鲫和鲤的四倍体共同祖先; 而第二轮多倍化则发生在大约51万年前, 在鲫这一分支上形成了六倍体银鲫[44]。本研究克隆鉴定的6个银鲫F系bmp15不同等位基因的序列比对表明, 银鲫是一个异源六倍体。接着, 我们搜索了四倍体鲫以及人、小鼠、鸡、爪蟾、雀鳝、斑马鱼、稀有鲫、青鳉、罗非鱼和河鲀的基因组中的bmp15基因, 结果表明, 在鲫基因组中有3个bmp15等位基因, 而在其他物种的基因组中, 均只搜索到一个bmp15基因。系统进化树和bmp15相邻基因的同线性分析均表明(图 2和图 3), 在银鲫的进化历程中发生了两轮多倍化, 其中早期的异源多倍化整合了来自2个原始祖先的染色体组, 导致银鲫和鲫的四倍体共同原始祖先基因组中包含bmp15a和bmp15b; 随后第二轮同源多倍化最终导致银鲫存在6个bmp15等位基因。
由于染色体不平衡和基因组的不稳定, 新形成的异源多倍体会发生复杂的、非孟德尔遗传方式的基因组变化, 包括染色体重排[52, 53]、不同来源的2个部分同源基因(Homeologs)的缺失和沉默[54-59]、偏性或显性表达[59-61]等。相对于Cgbmp15a的3个BAC克隆上的邻近基因的分布, Cgbmp15b的3个BAC克隆基因均缺失了leap2基因, 并且Cgbmp15b-2-BAC还缺失了nhsl2和slc38a7基因, Cgbmp15b-1-BAC还缺失了lppl和cyp2x10.2基因(图 2)。同时, 尽管在成体组织分布、不同发育阶段卵子及孕酮激素DHP诱导卵母细胞成熟过程中, Cgbmp15a和Cgbmp15b具有相似的表达谱式, 但Cgbmp15a的表达水平均显著高于Cgbmp15b的表达水平(图 4和图 5)。这表明银鲫在形成异源多倍体后, 其基因组在随后的二倍化过程中发生了复杂的变化, 包括不同来源的2个部分同源基因的缺失或偏性表达。
与其他物种bmp15在成体组织中的分布相同, 银鲫Cgbmp15a和Cgbmp15b均在卵巢中检测到了高丰度的转录本, 同时Cgbmp15a也在肝脏、脑、下丘脑和垂体等组织中检测到低丰度的mRNA存在。在不同鱼类中, bmp15基因在卵子发生的不同阶段表达水平的变化是不同的。譬如, bmp15在斑马鱼不同发育阶段的卵母细胞中均有表达, 且表达水平无显著性差异[11]。在欧洲海鲈中, bmp15在初级卵母细胞中高表达, 从卵黄生成时期开始显著降低[12]。在黑鲷中, bmp15在早期卵母细胞中高表达, 随着卵母细胞的生长, 表达水平下降, 在卵黄发生的卵母细胞中基本检测不到转录本的存在[17]; bmp15在银鲫Ⅰ期卵母细胞(0.025—0.062 mm)中表达水平最高, 在Ⅱ-Ⅳ期卵母细胞中维持着相对低丰度、稳定的转录本含量[14]。研究结果与已报道的银鲫bmp15不同, Cgbmp15a和Cgbmp15b在卵母细胞发育过程中高表达于皮质泡期卵母细胞中(0.21—0.23 mm, 图 5A)。通过比较卵母细胞分期的直径大小, 我们推测导致这种不同结果的原因可能是由于取材分析的银鲫卵巢处于不同发育阶段, 或者银鲫克隆系的不同。与人绒毛膜促性腺激素(hCG)体外诱导银鲫卵母细胞成熟过程中, 银鲫bmp15上调表达结果相同[14], 当我们采用DHP体外诱导银鲫F系GV1期卵母细胞成熟时, Cgbmp15a在诱导6h后开始上调表达水平, 在诱导8h后达到最高表达水平, 随后下降。而Cgbmp15b变化较小(图 5B)。前期的研究表明, 银鲫GV1卵母细胞在DHP体外诱导4.5h后, 生发泡开始破裂; 诱导8h后, 超过90%的卵母细胞的生发泡均破裂[50]。结合Cgbmp15a和Cgbmp15b在成体组织及不同发育阶段卵子中的表达差异(图 4和图 5A), 我们推测Cgbmp15a在银鲫的卵母细胞发育和成熟中起着主要作用。Cgbmp15a和Cgbmp15b在银鲫生殖中的功能歧化有待进一步的研究。
-
图 4 Cgbmp15a和Cgbmp15b mRNA在银鲫不同成体组织中的表达分析
以β-actin为内参; Mean±SD (n=3); *表示Cgbmp15a和Cgbmp15b在同一组织中表达水平具有显著性差异(P<0.05); 下同
Figure 4. Relative expression of Cgbmp15a and Cgbmp15b in different adult tissues
β-actin was used as control. Each bar represents mean ± standard deviation (SD, n=3). Asterisks (*) indicate significant differences (P<0.05) between Cgbmp15a and Cgbmp15b expression in same tissue. The same applies below
表 1 本文中所用引物
Table 1 Primers used in this study
引物名称Primer 序列Sequence (5′-3′) 应用Usage Cgbmp15aF GGTCACGACTGTCTGAGA BAC克隆筛选 Cgbmp15aR GAGTCTGCAATCATGTCTTC Cgbmp15bF CTACCAGCAGTCACACCT Cgbmp15bR GAGTCTGCAATCATGTCTTC Cgbmp15a-5′-Outer TATACAGGATAGCGGTGGAT RACE Cgbmp15a-5′-Inner ACTGTAAGGCTGCTTCAAG Cgbmp15b-5′-Outer GGCGTCACCAATACAGAA Cgbmp15b-5′-Inner ACAAGTTATTCGGCTCCAA Cgbmp15a-3′-Outer GAGTCTGCAATCATGTCTTC Cgbmp15a-3′-Inner GCAGGATACGAGGACAATC Cgbmp15b-3′-Outer GAGTCTGCAATCATGTCTTC Cgbmp15b-3′-Inner CTCCATCATCAGCACACT UPM CTAATACGACTCACTATAGGGCAAGCAGTGGTATCAACGCAGAGT NUP CTAATACGACTCACTATAGGGC Cgbmp15a QF GGGTCCAACACTGTAAGGC RT-qPCR Cgbmp15a QR CACCAATTTGTCCAACGAG Cgbmp15b QF TTATTCGGCTCCAACACTG Cgbmp15b QR TGTCCAACGAGAGGTTTTCC β-actin-QF AGCACGGTATTGTGACTAACTG β-actin-QR TCGAACATGATCTGTGTCATC -
[1] Lochab A K, Extavour C G. Bone morphogenetic protein (BMP) signaling in animal reproductive system development and function [J]. Developmental Biology, 2017, 427(2): 258-269. doi: 10.1016/j.ydbio.2017.03.002
[2] Dube J L, Wang P, Elvin J, et al. The bone morphogenetic protein 15 gene is X-linked and expressed in oocytes [J]. Molecular Endocrinology, 1998, 12(12): 1809-1817. doi: 10.1210/mend.12.12.0206
[3] Monestier O, Servin B, Auclair S, et al. Evolutionary origin of bone morphogenetic protein 15 and growth and differentiation factor 9 and differential selective pressure between mono- and polyovulating species [J]. Biology of Reproduction, 2014, 91(4): 83.
[4] Caixeta E S, Sutton-McDowall M L, Gilchrist R B, et al. Bone morphogenetic protein 15 and fibroblast growth factor 10 enhance cumulus expansion, glucose uptake, and expression of genes in the ovulatory cascade during in vitro maturation of bovine cumulus-oocyte complexes [J]. Reproduction, 2013, 146(1): 27-35. doi: 10.1530/REP-13-0079
[5] Abir R, Fisch B, Johnson M H. BMP15, fertility and the ovary [J]. Reproductive Biomedicine Online, 2014, 29(5): 525-526. doi: 10.1016/j.rbmo.2014.09.007
[6] De Castro F C, Cruz M H, Leal C L. Role of growth differentiation factor 9 and bone morphogenetic protein 15 in ovarian function and their importance in mammalian female fertility - A review [J]. Asian-Australasian Journal of Animal Sciences, 2016, 29(8): 1065-1074.
[7] Otsuka F, Shimasaki S. A negative feedback system between oocyte bone morphogenetic protein 15 and granulosa cell kit ligand: its role in regulating granulosa cell mitosis [J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(12): 8060-8065. doi: 10.1073/pnas.122066899
[8] Su Y Q, Wu X, O’Brien M J, et al. Synergistic roles of BMP15 and GDF9 in the development and function of the oocyte-cumulus cell complex in mice: genetic evidence for an oocyte-granulosa cell regulatory loop [J]. Developmental Biology, 2004, 276(1): 64-73. doi: 10.1016/j.ydbio.2004.08.020
[9] Yan C, Wang P, DeMayo J, et al. Synergistic roles of bone morphogenetic protein 15 and growth differentiation factor 9 in ovarian function [J]. Molecular Endocrinology, 2001, 15(6): 854-866. doi: 10.1210/mend.15.6.0662
[10] Persani L, Rossetti R, Di Pasquale E, et al. The fundamental role of bone morphogenetic protein 15 in ovarian function and its involvement in female fertility disorders [J]. Human Reproduction Update, 2014, 20(6): 869-883. doi: 10.1093/humupd/dmu036
[11] Clelland E, Kohli G, Campbell R K, et al. Bone morphogenetic protein-15 in the zebrafish ovary: complementary deoxyribonucleic acid cloning, genomic organization, tissue distribution, and role in oocyte maturation [J]. Endocrinology, 2006, 147(1): 201-209. doi: 10.1210/en.2005-1017
[12] Halm S, Ibanez A J, Tyler C R, et al. Molecular characterisation of growth differentiation factor 9 (gdf9) and bone morphogenetic protein 15 (bmp15) and their patterns of gene expression during the ovarian reproductive cycle in the European sea bass [J]. Molecular & Cellular Endocrinology, 2008, 291(1-2): 95-103.
[13] Garcia-Lopez A, Sanchez-Amaya M I, Halm S, et al. Bone morphogenetic protein 15 and growth differentiation factor 9 expression in the ovary of European sea bass (Dicentrarchus labrax): cellular localization, developmental profiles, and response to unilateral ovariectomy [J]. Reproductive Biology & Endocrinology, 2011, 174(3): 326-334.
[14] Chen A Q, Liu Z W, Yang Z G, et al. Characterization of bmp15 and its regulation by human chorionic gonadotropin in the follicle of gibel carp (Carassius auratus gibelio) [J]. Comparative Biochemistry and Physiology B: Biochemistry & Molecular Biology, 2012, 163(1): 121-128.
[15] Zhang Y, Yuan C, Qin F, et al. Molecular characterization of gdf9 and bmp15 genes in rare minnow Gobiocypris rarus and their expression upon bisphenol a exposure in adult females [J]. Gene, 2014, 546(2): 214-221. doi: 10.1016/j.gene.2014.06.013
[16] Palomino J, Herrera G, Dettleff P, et al. Growth differentiation factor 9 and bone morphogenetic protein 15 expression in previtellogenic oocytes and during early embryonic development of Yellow-tail Kingfish Seriola lalandi [J]. Biological Research, 2014, 47(1): 60. doi: 10.1186/0717-6287-47-60
[17] Wu G C, Luo J W, Li H W, et al. Robust gdf9 and bmp15 expression in the oocytes of ovotestes through the Figla-independent pathway in the hermaphroditic black porgy, Acanthopagrus schlegelii [J]. PLoS One, 2017, 12(10): e186991.
[18] Kleppe L, Edvardsen R B, Furmanek T, et al. bmp15l, figla, smc1bl, and larp6l are preferentially expressed in germ cells in Atlantic salmon (Salmo salar L.) [J]. Molecular Reproduction & Development, 2017, 84(1): 76-87.
[19] Yadav H, Lal B. BMP15 in catfish testis: Cellular distribution, seasonal variation, and its role in steroidogenesis [J]. Steroids, 2017, 125: 114-123. doi: 10.1016/j.steroids.2017.07.002
[20] Fernandez T, Palomino J, Parraguez V H, et al. Differential expression of GDF-9 and BMP- 15 during follicular development in canine ovaries evaluated by flow cytometry [J]. Animal Reproduction Science, 2016, 167: 59-67. doi: 10.1016/j.anireprosci.2016.02.005
[21] Pennetier S, Uzbekova S, Perreau C, et al. Spatio-temporal expression of the germ cell marker genes MATER, ZAR1, GDF9, BMP15, and VASA in adult bovine tissues, oocytes, and preimplantation embryos [J]. Biology of Reproduction, 2004, 71(4): 1359-1366. doi: 10.1095/biolreprod.104.030288
[22] Silva J R, Van Den H R, Van Tol H T, et al. Expression of growth differentiation factor 9(GDF9), bone morphogenetic protein 15(BMP15), and BMP receptors in the ovaries of goats [J]. Molecular Reproduction & Development, 2005, 70(1): 11-19.
[23] Kasahara M, Naruse K, Sasaki S, et al. The medaka draft genome and insights into vertebrate genome evolution [J]. Nature, 2007, 447(7145): 714-719. doi: 10.1038/nature05846
[24] Jaillon O, Aury J M, Brunet F, et al. Genome duplication in the teleost fish Tetraodon nigroviridis reveals the early vertebrate proto-karyotype [J]. Nature, 2004, 431(7011): 946-957. doi: 10.1038/nature03025
[25] Braasch I, Gehrke A R, Smith J J, et al. The spotted gar genome illuminates vertebrate evolution and facilitates human-teleost comparisons [J]. Nature Genetics, 2016, 48(4): 427-437. doi: 10.1038/ng.3526
[26] Wapinski I, Pfeffer A, Friedman N, et al. Natural history and evolutionary principles of gene duplication in fungi [J]. Nature, 2007, 449(7158): 54-61. doi: 10.1038/nature06107
[27] Comai L. The advantages and disadvantages of being polyploid [J]. Nature Reviews Genetics, 2005, 6(11): 836-846. doi: 10.1038/nrg1711
[28] Otto S P. The evolutionary consequences of polyploidy [J]. Cell, 2007, 131(3): 452-462. doi: 10.1016/j.cell.2007.10.022
[29] Soltis P S, Liu X, Marchant D B, et al. Polyploidy and novelty: Gottlieb’s legacy [J]. Philosophical Transactions of the Royal Society of London, 2014, 369(1648).
[30] Zhou L, Gui J F. Natural and artificial polyploids in aquaculture [J]. Aquaculture & Fisheries, 2017, 2: 103-111.
[31] Zhou L, Gui J F. Applications of genetic breeding biotechnologies in Chinese aquaculture [A]// Gui J F, Tang Q S, Li Z J, et al (Eds.), Aquaculture in China: Success Stories and Modern Trends [C]. Oxford: John Wiley & Sons, 2017: 463-495
[32] 桂建芳, 周莉. 多倍体银鲫克隆多样性和双重生殖方式的遗传基础和育种应用 [J]. 中国科学: 生命科学, 2010, 40(2): 97-103. Gui J F, Zhou L. Genetic basis and breeding application of clonal diversity and dual reproduction modes in polyploidy Carassius auratus gibelio [J]. Science China Life Science, 2010, 40(2): 97-103.
[33] Liu W, Li S Z, Li Z, et al. Complete depletion of primordial germ cells in an all-female fish leads to sex-biased gene expression alteration and sterile all-male occurrence [J]. BMC Genomics, 2015, 16: 971. doi: 10.1186/s12864-015-2130-z
[34] Rylková K, Kalous L, Šlechtová V, et al. Many branches, one root: First evidence for a monophyly of the morphologically highly diverse goldfish (Carassius auratus) [J]. Aquaculture, 2010, 302(1): 36-41.
[35] Zhang J, Sun M, Zhou L, et al. Meiosis completion and various sperm responses lead to unisexual and sexual reproduction modes in one clone of polyploid Carassius gibelio [J]. Scientific Reports, 2015, 5: 10898. doi: 10.1038/srep10898
[36] Li X Y, Liu X L, Ding M, et al. A novel male-specific SET domain-containing gene setdm identified from extra microchromosomes of gibel carp males [J]. Science Bulletin, 2017, 62(8): 528-536. doi: 10.1016/j.scib.2017.04.002
[37] Li X Y, Liu X L, Zhu Y J, et al. Origin and transition of sex determination mechanisms in a gynogenetic hexaploid fish [J]. Heredity, 2018, doi: 10.1038/s41437-017-0049-7
[38] Liu X L, Jiang F F, Wang Z W, et al. Wider geographic distribution and higher diversity of hexaploids than tetraploids in Carassius species complex reveal recurrent polyploidy effects on adaptive evolution [J]. Scientific Reports, 2017, 7(1): 5395. doi: 10.1038/s41598-017-05731-0
[39] Liu X L, Li X Y, Jiang F F, et al. Numerous mitochondrial DNA haplotypes reveal multiple independent polyploidy origins of hexaploids in Carassius species complex [J]. Ecology & Evolution, 2017, 7(24): 10604-10615.
[40] Zhou L, Wang Z W, Wang Y, et al. Crucian carp and gibel carp culture [A]//Gui J F, Tang Q S., Li Z J, et al (Eds.), Aquaculture in China: Success Stories and Modern Trends [C]. Oxford: John Wiley & Sons, 2017: 149-157
[41] Zhou L, Gui J F. Karyotypic diversity in polyploid gibel carp, Carassius auratus gibelio Bloch [J]. Genetica, 2002, 115(2): 223-232. doi: 10.1023/A:1020102409270
[42] Ohno S, Muramoto J, Christian L, et al. Diploid-tetraploid relationship among old-world members of the fish family Cyprinidae [J]. Chromosoma, 1967, 23(1): 1-9. doi: 10.1007/BF00293307
[43] Zhu H P, Ma D M, Gui J F. Triploid origin of the gibel carp as revealed by 5S rDNA localization and chromosome painting [J]. Chromosome Research, 2006, 14(7): 767-776. doi: 10.1007/s10577-006-1083-0
[44] Li X Y, Zhang X J, Li Z, et al. Evolutionary history of two divergent Dmrt1 genes reveals two rounds of polyploidy origins in gibel carp [J]. Molecular Phylogenetics & Evolution, 2014, 78: 96-104.
[45] Gao F X, Wang Y, Zhang Q Y, et al. Distinct herpesvirus resistances and immune responses of three gynogenetic clones of gibel carp revealed by comprehensive transcriptomes [J]. BMC Genomics, 2017, 18(1): 561. doi: 10.1186/s12864-017-3945-6
[46] Mou C Y, Wang Y, Zhang Q Y, et al. Differential interferon system gene expression profiles in susceptible and resistant gynogenetic clones of gibel carp challenged with herpesvirus CaHV [J]. Developmental & Comparative Immunology, 2018, doi: 10.1016/j.dci.2018.04.024
[47] Xie J, Wen J J, Chen B, et al. Differential gene expression in fully-grown oocytes between gynogenetic and gonochoristic crucian carps [J]. Gene, 2001, 271(1): 109-116. doi: 10.1016/S0378-1119(01)00491-7
[48] Liu Z, Zhang X, Wang W, et al. Molecular characterization and expression of an oocyte-specific histone stem-loop binding protein in Carassius gibelio [J]. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2015, 190: 46-53. doi: 10.1016/j.cbpb.2015.08.011
[49] Xiao Q, Xia J H, Zhang X J, et al. Type-IV antifreeze proteins are essential for epiboly and convergence in gastrulation of zebrafish embryos [J]. International Journal of Biological Sciences, 2014, 10(7): 715-732. doi: 10.7150/ijbs.9126
[50] 李志, 汪洋, 周莉, 等. 银鲫卵母细胞体外诱导成熟技术的建立 [J]. 水生生物学报, 2017, 41(5): 984-991. doi: 10.7541/2017.123 Li Z, Wang Y, Zhou L, et al. Development of in vitro maturation technology for gibel carp oocytes [J]. Acta Hydrobiologica Sinica, 2017, 41(5): 984-991. doi: 10.7541/2017.123
[51] Xu P, Zhang X, Wang X, et al. Genome sequence and genetic diversity of the common carp, Cyprinus carpio [J]. Nature Genetics, 2014, 46(11): 1212-1219. doi: 10.1038/ng.3098
[52] Xiong Z, Gaeta R T, Pires J C. Homoeologous shuffling and chromosome compensation maintain genome balance in resynthesized allopolyploid Brassica napus [J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(19): 7908-7913. doi: 10.1073/pnas.1014138108
[53] He Z, Wang L, Harper A L, et al. Extensive homoeologous genome exchanges in allopolyploid crops revealed by mRNAseq-based visualization [J]. Plant Biotechnology Journal, 2017, 15(5): 594-604. doi: 10.1111/pbi.12657
[54] Salmon A, Flagel L, Ying B, et al. Homoeologous nonreciprocal recombination in polyploid cotton [J]. New Phytologist, 2010, 186(1): 123-134. doi: 10.1111/j.1469-8137.2009.03093.x
[55] Buggs R J, Chamala S, Wu W, et al. Rapid, repeated, and clustered loss of duplicate genes in allopolyploid plant populations of independent origin [J]. Current Biology, 2012, 22(3): 248-252. doi: 10.1016/j.cub.2011.12.027
[56] Doyle J J, Flagel L E, Paterson A H, et al. Evolutionary genetics of genome merger and doubling in plants [J]. Annual Review of Genetics, 2008, 42(1): 443-461. doi: 10.1146/annurev.genet.42.110807.091524
[57] Jackson S, Chen Z J. Genomic and expression plasticity of polyploidy [J]. Current Opinion in Plant Biology, 2010, 13(2): 153-159. doi: 10.1016/j.pbi.2009.11.004
[58] Lashermes P, Hueber Y, Combes M C, et al. Inter-genomic DNA exchanges and homeologous gene silencing shaped the nascent allopolyploid coffee genome (Coffea arabica L.) [J]. G3 Genesgenetics, 2016, 6(9): 2937-2948. doi: 10.1534/g3.116.030858
[59] Wang X, Dong Q, Li X, et al. Cytonuclear variation of rubisco in synthesized rice hybrids and allotetraploids [J]. Plant Genome, 2017, 10(3): 1-11.
[60] Hu G, Koh J, Yoo M J, et al. Gene-expression novelty in allopolyploid cotton: a proteomic perspective [J]. Genetics, 2015, 200(1): 91-104. doi: 10.1534/genetics.115.174367
[61] Yoo M J, Szadkowski E, Wendel J F. Homoeolog expression bias and expression level dominance in allopolyploid cotton [J]. Heredity, 2013, 110(2): 171-180. doi: 10.1038/hdy.2012.94
-
期刊类型引用(1)
1. 张琴琴,周莉,李志,甘瑞海,俞兆曦,桂建芳,汪洋. 多倍体银鲫nanos2等位多态性、共线性和表达模式分析. 水生生物学报. 2020(05): 1087-1096 .  本站查看
本站查看
其他类型引用(0)



 下载:
下载: