OVERVIEW OF CYANOBACTERIA MOTILITY AND ITS REGULATION MECHANISMS
-
摘要: 蓝藻运动是蓝藻积极适应外部环境的体现。根据蓝藻种类及运动方式不同可将蓝藻运动概括为三类: 即蓝藻泳动、蓝藻蹭动、蓝藻滑动。文章综述了蓝藻运动的相关研究进展, 对蓝藻不同运动方式进行描述, 并对蓝藻各种运动方式可能产生机制进行了总结, 列举了未来研究蓝藻运动需要解答的问题。Abstract: The motility of Cyanobacteria is a reflection of Cyanobacteria’s active adaptation to external environment actively. The movement of Cyanobacteria can be divided into three categories according to the types and movement modes: swimming motility, twitching motility, and gliding motility. This paper refers to the related literature on the motility of Cyanobacteria, describes different movement modes of Cyanobacteria, summarizes the possible mechanism of various movement modes of Cyanobacteria, and lists the questions that need to be answered in the future research on Cyanobacteria motility.
-
Keywords:
- Cyanobacteria /
- Swimming motility /
- Twitching motility /
- Gliding motility
-
蓝藻又称蓝细菌, 是地球上最早出现的光合放氧生物[1]。人们通常认为真核生物中光合细胞器(如高等植物叶绿体)的起源可能是吞噬性宿主和蓝藻内共生的结果[2], 深入理解蓝藻生命活动过程可为认识真核生物中光合细胞器的发生、调控及功能发挥具有重要的借鉴意义。蓝藻与某些原核细菌, 如黄色黏球菌(Myxococcus xanthus), 铜绿假单胞菌(Pseudomonas aeruginosa)类似, 具有运动能力, 能够在液体或潮湿固体表面运动[3—6]。蓝藻运动可使蓝藻选择更适应自身生长的环境, 例如更有利的光照或营养环境, 是蓝藻长期进化过程中形成的有效环境适应策略。单细胞蓝藻和丝状蓝藻都可以发生运动。根据蓝藻种类及运动方式的不同可将蓝藻运动分为三类[7]: (1) 蓝藻泳动(Swimming motility), 主要指某些海洋单细胞蓝藻在水中运动的形式, 如聚球藻WH8102(Synechococcus sp. WH8102)在水中的运动; (2) 蓝藻蹭动(Twitching motility), 主要指某些单细胞蓝藻沿固体表面的运动形式, 如集胞藻PCC6803(Synechocystis sp. PCC6803, 下文简称为集胞藻)的趋光运动; (3) 蓝藻滑动(Gliding motility), 主要指某些丝状蓝藻沿固体表面的运动形式, 如某些颤藻目(Oscillatoriales)或念珠藻目(Nostocales)蓝藻的运动。蓝藻运动过程受到精细复杂的调控, 需要大量蛋白协同参与完成, 研究其调控机理可使我们从根本上理解蓝藻运动过程, 丰富我们对原核生物积极主动适应外界环境策略的认识, 为构建人造微型运动设备提供理论模型。下文对蓝藻不同的运动方式及其可能的发生机制分别进行阐述, 概括了蓝藻运动机制研究所需揭示的几个主要问题, 为相关研究者提供参考。
1. 蓝藻泳动
1.1 蓝藻泳动现象描述
蓝藻泳动现象主要发现于生活在热带或亚热带开阔海洋中的某些聚球藻属中[8]。Waterbury等[8]首先描述了聚球藻的泳动状况。发生泳动的聚球藻成球状或棒状, 直径0.7—0.9 μm, 长度1.25—2.5 μm, 其在液体中的泳动速度5—25 μm/s[8]。聚球藻绕其纵轴旋转并移动, 增加液体环境中的黏度可以使其降速或静止, 这些聚球藻可以顺时针旋转也可以逆时针旋转, 但每个细胞只能朝一个方向旋转, 细胞在液体中可以3—5 r/s的速度旋转[8]。与能够在潮湿固体表面发生蹭动或滑动的蓝藻不同, 在液体中泳动的聚球藻不能沿潮湿固体表面运动[8]。
1.2 蓝藻泳动相关蛋白及其运动机制解释模型
各种实验数据表明发生泳动的聚球藻并不像某些原核生物(如大肠杆菌, Escherichia coli)一样具有鞭毛, 敲除聚球藻WH8102中对某些蓝藻(如集胞藻)蹭动具有重要作用的Ⅳ型菌毛合成相关基因同源基因pilT及pilC也不影响其泳动, 说明聚球藻泳动不依靠鞭毛或Ⅳ型菌毛, 其泳动机制与具有鞭毛或Ⅳ型菌毛的原核生物并不相同[7]。在聚球藻WH8102中, 通过转座子突变及表型筛选实验, 发现一种命名为SwmA(Swimming motility protein A), 分子量为130 kD, 构成细胞S-layer的糖蛋白对细胞泳动时推力的产生具有重要作用, 该蛋白含有多个钙离子结合结构域及潜在糖基化位点, 其编码基因的敲除会使细胞丧失在液体中移动的能力, 但不会影响聚球藻细胞自身的转动[9, 10]。该藻中另外一种分子量为1.12 MD, 位于细胞表层并呈点状分布的蛋白SwmB对聚球藻WH8102的泳动过程也是必需的, SwmA和SwmB都位于细胞表层, SwmB编码基因突变并不影响SwmA分布[11]。通过转座子突变实验还发现聚球藻WH8102基因组上有2个基因簇(synw0087-synw0088及synw0192-synw0195)对该藻泳动是必需的, 其中synw0087和synw0195编码糖基转移酶, synw0193编码Ⅰ型分泌系统中的ABC转运子, synw0194编码Ⅰ型分泌系统中的膜融合蛋白, 这些基因编码蛋白通过影响SwmA合成或分布进而影响细胞泳动[12]。该藻基因组上还编码一种细胞骨架类似蛋白, 命名为MreB(Membrane-associated rod shape-determining protein B)类似蛋白, 该蛋白可能对聚球藻WH8102的泳动过程具有重要作用[7]。而在另外一种发生泳动的聚球藻WH8113(Synechococcus sp. WH 8113)中, 培养基中钠离子及钙离子浓度对该藻的泳动过程至关重要[13, 14], 表明这2种离子可能与聚球藻泳动时动力供应或泳动相关蛋白功能发挥有关。
关于聚球藻如何在缺少鞭毛等亚细胞结构情况下泳动有一种解释, 即细胞外膜层收缩和扩张产生波动从而使细胞在液体中泳动[15]。该波动过程产生机理有2种模型解释(图 1): 一种是钙离子去极化过程使细胞表层产生局部膜膨胀并回缩, 进而沿膜表层传播产生波动, 从而推动细胞运动(图 1A)[14]; 另一种是参考黄色黏球菌的单个细胞运动过程[16-18], 该模型认为聚球藻细胞内动力蛋白及其他运动相关蛋白组成的蛋白复合体形成跨质膜结构, 复合体位于胞内的结构沿着胞内骨架蛋白(如MreB类似蛋白)形成的轨道发生连续闭环运动, 并带动复合体位于周质空间部分使细胞肽聚糖层及外膜产生波动, 进而推动细胞运动(图 1B)[16-18]。这2种模型的提出均基于已有的实验结果, 但对这2种模型的验证均需要更多的实验证据。另外聚球藻在泳动时发生自身转动的机制并不清楚。
![]() A. 钙离子去极化过程使细胞外膜局部膨胀并回缩, 波动沿外膜表层扩散并产生推动力; B. 骨架蛋白复合体在胞内形成连续闭环轨道, 动力蛋白复合体形成跨质膜结构连接骨架蛋白和周质空间运动蛋白复合体, 动力蛋白复合体沿轨道运动带动周质空间运动蛋白复合体运动并使肽聚糖层及外膜变形膨胀并波动, 产生推动力推动细胞泳动A. The process of calcium depolarization may serve to generate local swellings and waves on the cell surface, and the waves propagate along the surface to produce driving forces; B. Cytoskeletal protein complexes form a continuous helical track in the cell, and motor protein complexes form transmembrane structure which links cytoskeletal protein complexes and periplasmic space motility related protein complexes. Motor protein complexes travel along the helical track and drive the periplasmic space motility related protein complexes to cause deformations and local swellings of peptidoglycan layer and outer membrane, and subsequently produce surface waves and driving forces to propel cells to swim
A. 钙离子去极化过程使细胞外膜局部膨胀并回缩, 波动沿外膜表层扩散并产生推动力; B. 骨架蛋白复合体在胞内形成连续闭环轨道, 动力蛋白复合体形成跨质膜结构连接骨架蛋白和周质空间运动蛋白复合体, 动力蛋白复合体沿轨道运动带动周质空间运动蛋白复合体运动并使肽聚糖层及外膜变形膨胀并波动, 产生推动力推动细胞泳动A. The process of calcium depolarization may serve to generate local swellings and waves on the cell surface, and the waves propagate along the surface to produce driving forces; B. Cytoskeletal protein complexes form a continuous helical track in the cell, and motor protein complexes form transmembrane structure which links cytoskeletal protein complexes and periplasmic space motility related protein complexes. Motor protein complexes travel along the helical track and drive the periplasmic space motility related protein complexes to cause deformations and local swellings of peptidoglycan layer and outer membrane, and subsequently produce surface waves and driving forces to propel cells to swim1.3 蓝藻泳动意义
发生泳动的聚球藻是从热带或亚热带开阔大洋中分离获得[8, 19], 这些地方光照充足, 但营养盐浓度(尤其生物可利用氮源)含量较低, 营养盐浓度通常是限制自养生物生物量的主要因素[20, 21]。在这些区域动物粪便或大型浮游植物残骸形成富含营养的微斑块或微聚集体对营养元素循环利用具有重要作用[22]。聚球藻泳动可能使细胞对环境中的不同营养物质浓度作出反应, 靠近富含营养的微斑块或微聚集体, 促进自身生长。Willy等[23]在实验室条件下探究过聚球藻对23种不同化合物是否存在因为浓度不同而形成的趋化运动现象, 发现聚球藻细胞对环境中不同浓度的NH4Cl、NaNO3、尿素、甘氨酸和丙氨酸有响应, 细胞会向化合物浓度较高的区域泳动, 暗示聚球藻细胞在自然环境中会通过泳动获得更有利的营养环境。Willy等[24]也探究过聚球藻泳动是否受光照影响, 发现当快速改变环境中光照强度时, 聚球藻细胞泳动速度和方向并未改变, 将泳动细胞置于光强成连续梯度变化的环境中时, 聚球藻细胞并未产生趋光运动或避光运动现象, 说明光照改变对聚球藻细胞泳动过程影响较小。聚球藻泳动能否帮助自己躲避捕食者捕食? Strom等[25]通过对比野生型聚球藻WH8102和swmA、swmB突变株被纤毛虫, 鞭毛虫, 尖尾藻捕食频率发现泳动对细胞降低被捕食率没有显著影响。关于聚球藻泳动对自身的意义需要更多的自然环境下的证据支持。
2. 蓝藻蹭动
2.1 蓝藻蹭动现象描述
蓝藻蹭动现象是法国巴斯德研究中心的几个研究组首次发现的, 其中对淡水单细胞蓝藻集胞藻蹭动研究尤为细致[26, 27]。集胞藻在潮湿固体培养基平板(琼脂浓度0.4%—0.8%)上会展现出较慢的蹭动速度, 通常1—2 μm/min[28]。如果将细胞置于单侧光照条件下, 细胞就会展现出向光源方向移动的能力, 也称为趋光运动[7, 28, 29]。在几种单细胞蓝藻中报道过趋光运动现象, 其中嗜热聚球藻(Thermosynechococcus elongatus)在不同光质及光强条件下趋光运动反应不同, 例如在低光强条件下[3 μmol photons/(m2·s)], 只有红光区展现出趋光运动现象, 而当光强为10 μmol photons/(m2·s)时, 藻细胞在530、570、640和680 nm波段均展现出明显趋光运动[30]。近期Yang等[31]从野外环境中分离到一株聚球藻UTEX3055(Synechococcus elongatus strain UTEX3055), 其基因组序列与聚球藻PCC7942(Synechococcus elongatus PCC7942)相似度达98.5%, 也可发生趋光运动。集胞藻在极微弱光照条件下 [约0.001 μmol photons/(m2·s)] 就能引发趋光运动, 约1 μmol photons/(m2·s) 光照强度即达到趋光运动饱和光强[28, 32]。由于集胞藻存在不同亚种, Fiedler等[33]探究了来自不同实验室的4种集胞藻的运动情况, 发现有一种野生型集胞藻丧失了运动能力, 另外3种野生型藻株在白光和蓝光条件下展现出不同的运动情况。在单侧白光照射条件下, 3种藻株展现出类似的趋光运动性质, 但它们在单侧蓝光照射条件下的趋光运动能力较白光下均受到不同程度的抑制, 其中的一种野生藻株在单侧蓝光照射条件下完全不发生趋光运动[33]。实验室中模拟自然界较复杂光照条件下集胞藻的趋光运动情况发现, 红光和绿光主要影响集胞藻细胞运动方向, 但对细胞运动速度影响不大, 而蓝光显著抑制细胞运动。当集胞藻细胞接受不同方向的单侧红光照时, 细胞会沿着光照方向矢量和运动, 而当同时给予单侧绿光和蓝光照射时, 集胞藻细胞会根据两种光的强度比实时调整运动方向及生长状态, 说明集胞藻细胞在复杂光环境条件下可以整合光照信号并通过运动作出反应[34]。
关于蓝藻蹭动机制的研究主要集中在对集胞藻和聚球藻趋光运动机制的研究, 下面着重介绍。
2.2 集胞藻参与趋光运动相关蛋白及其作用机制
根据集胞藻中参与趋光运动过程的蛋白功能划分, 可将它们分为三类: 第一类是Ⅳ型菌毛蛋白复合体结构及组装相关蛋白, 它们是蹭动现象发生的结构基础; 第二类参与趋光运动光信号感应及光信号传递, 感光蛋白通常含有光响应结构域; 第三类间接参与蹭动过程调节, 这类蛋白并不直接参与到Ⅳ型菌毛蛋白复合体组成及组装, 但会在转录或转录后水平直接或间接调节Ⅳ型菌毛蛋白复合体形成相关蛋白的表达或修饰, 进而影响细胞蹭动, 或者通过影响细胞其他生命活动间接影响细胞运动, 此类蛋白缺失也会使细胞丧失蹭动能力。
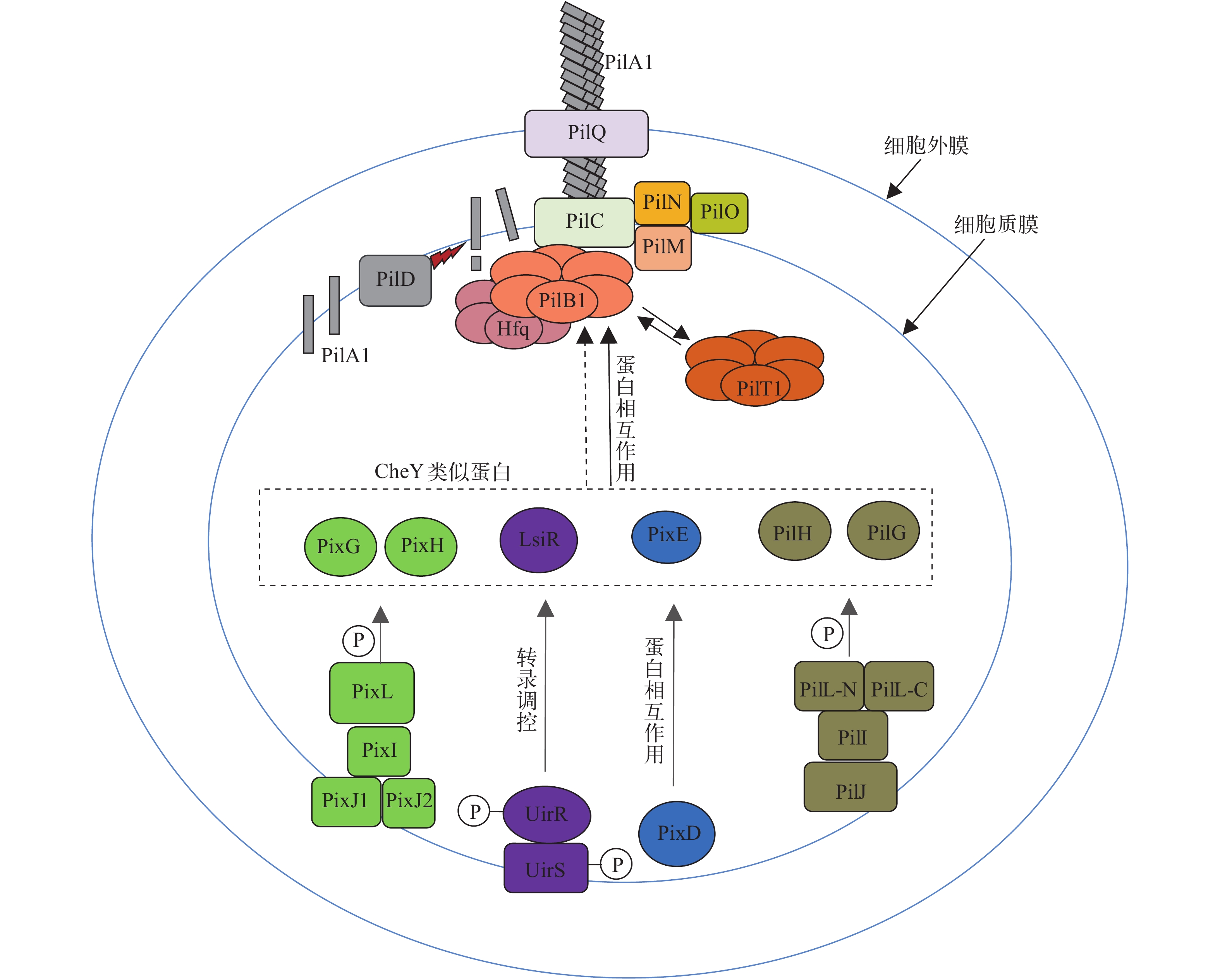
直接参与集胞藻Ⅳ型菌毛蛋白复合体形成蛋白 与高等植物叶绿体响应光照发生运动需要叶绿体微丝蛋白参与不同[35], 集胞藻发生蹭动的结构基础与某些革兰氏阴性菌(如黄色黏球菌)类似, 需要Ⅳ型菌毛蛋白复合体参与[29, 36]。Ⅳ型菌毛伸展、栓系、收缩是实现蹭动的结构基础[37-41]。Bhaya等[29]首先发现集胞藻表层有Ⅳ型菌毛类似结构, 直径为6—8 nm, 长度为4—5 μm, 称为粗菌毛。后续研究表明粗菌毛即为Ⅳ型菌毛[42]。近期Chen等[43]对集胞藻Ⅳ型菌毛具有的多种生物学功能最新研究进展进行了综述, 此处只关注Ⅳ型菌毛与集胞藻运动的关系。根据对黄色黏球菌、铜绿假单胞菌等其他细菌Ⅳ型菌毛合成必需蛋白序列同源性比对, 发现集胞藻中直接参与Ⅳ型菌毛的合成蛋白编码基因有pilA1(sll1694)、pilA9(slr2015)、pilA10(slr2016)、pilA11(slr2017)、pilB1(slr0063)、pilC(slr0162)、pilD(slr1120)、pilT1(slr0161)、pilM(slr1274)、pilN(slr1275)、pilO(slr1276)和pilQ(slr1277)[42, 44]。PilA(Pilus assembly protein A, PilA)是Ⅳ型菌毛构成的基本亚基, 集胞藻中有11个PilA 同源蛋白, 其中PilA1是集胞藻Ⅳ型菌毛的基本构成亚基[42]。参考其他革兰氏阴性菌Ⅳ型菌毛合成模式, PilA合成后在运输至胞外的过程中, 其N端带正电的前导肽被肽酶及甲基化酶PilD切除并甲基化, 然后以螺旋状排列方式装配到菌毛基部[45-48]。pilA1缺失会使集胞藻表层Ⅳ型菌毛完全消失, 同时细胞完全丧失运动能力[42]。PilA2- PilA8氨基酸序列与PilA1有一定同源性, 但PilA2- PilA8编码基因突变并不会影响集胞藻Ⅳ型菌毛合成, 也不影响细胞运动[44]。PilA9、PilA10和PilA11被称为少量菌毛亚基, 这些菌毛亚基N端也有前导肽, 它们合成后可能被分泌到胞外辅助PilA1形成具有正常功能的Ⅳ型菌毛[49, 50]。PilB与PilT为ATP水解酶, 它们均可形成六聚体环装结构, 通过水解ATP改变蛋白的构象从而将PilA亚基添加到菌毛上或从菌毛上解离下来, 进而实现菌毛的伸长或回缩[38, 41, 51-56]。集胞藻中PilB的同源蛋白PilB1编码基因突变会使细胞表层Ⅳ型菌毛消失而丧失运动能力[42], PilT的同源蛋白PilT1编码基因突变会使细胞表层Ⅳ型菌毛数量显著增加, 但细胞也会丧失运动能力[42], 说明在集胞藻中PilB1与PilT1在Ⅳ型菌毛动态合成过程中发挥重要功能。PilC位于质膜上, 有利于菌毛往外延伸或往内收缩并起到稳定菌毛的作用[57-59]。PilM与PilN、PilO在质膜上形成复合体, 在稳定菌毛动态形成的过程中发挥功能[60, 61]。PilM还可与PilC、PilB和PilT发生动态相互作用, 而PilN可通过与PilM相互作用调节PilM的互作对象[62]。PilQ在外膜上形成多聚体孔状结构, 使Ⅳ型菌毛延伸至胞外[63, 64]。另外, 集胞藻中存在一种RNA伴侣蛋白同源蛋白Hfq(Host factor for RNA phage Q beta replication), 该蛋白可以与集胞藻中PilB1的C末端发生相互作用, 共同作用于Ⅳ型菌毛动态合成过程中[65-67]。集胞藻Ⅳ型菌毛合成组装参与蛋白及其功能总结如表 1所示。图 2中集胞藻Ⅳ型菌毛合成参考其他革兰氏阴性菌Ⅳ型菌毛合成过程。
表 1 集胞藻参与Ⅳ型菌毛合成基因及功能Table 1. Genes and its functions involved in the synthesis of type Ⅳ pili in Synechocystis基因编号
Code基因名称
Gene name功能概述
Functionsll1694 pilA1 Ⅳ型菌毛组成主要亚基 slr2015 pilA9 Ⅳ型菌毛组成辅助亚基 slr2016 pilA10 Ⅳ型菌毛组成辅助亚基 slr2017 pilA11 Ⅳ型菌毛组成辅助亚基 slr0063 pilB1 ATP水解酶, 作用于菌毛伸长 slr0161 pilT1 ATP水解酶, 作用于菌毛缩短 slr0162—0163 pilC Ⅳ型菌毛合成质膜平台复合体亚基 slr1120 pilD 菌毛亚基前导肽切除及N端甲基化 slr1274 pilM Ⅳ型菌毛合成质膜平台复合体亚基 slr1275 pilN Ⅳ型菌毛合成质膜平台复合体亚基 slr1276 pilO Ⅳ型菌毛合成质膜平台复合体亚基 slr1277 pilQ Ⅳ型菌毛合成外膜孔状复合体亚基 ssr3341 hfq 辅助菌毛伸长 ![]() 图 2 集胞藻趋光运动感光信号转导及Ⅳ型菌毛合成模式图集胞藻通过多条信号转导途径感应光照信号并调控Ⅳ型菌毛合成。PixJ1、UirS和PixD均可结合发色团感应光照信号并将信号往下游传递。PixJ1响应光照信号后可能通过与其编码基因所在操纵子中其他编码蛋白相互作用, 最终将磷酸基团转移至CheY类似蛋白PixG及PixH、PixG可能通过与PilB1相互作用调控Ⅳ型菌毛合成。UirS感应光照信号后可自磷酸化, 并可将磷酸基团转移至UirR, UirR磷酸化后可以转录激活LsiR编码基因表达, LsiR可能通过与PilB1相互作用调控Ⅳ型菌毛合成。PixD感应光照信号后改变与PixE的相互作用方式, PixE可以与PilB1相互作用调控Ⅳ型菌毛合成。PilG-PilJ及PilL-N、PilL-C所形成的信号转导通路与PixJ1所在通路类似, 但该通路中缺少直接感光蛋白。PixE与PilB1的相互作用已被实验证实, 用实线表示, 其余CheY类似蛋白与PilB1相互作用未被实验证实, 用虚线表示。PilC、PilM、PilN和PilO在质膜上形成Ⅳ型菌毛合成复合体平台, PilA1在PilD的作用下形成成熟菌毛亚基, 在PilC处添加到Ⅳ型菌毛上, 并通过PilQ在外膜上形成多聚体孔状结构使Ⅳ型菌毛延伸至胞外。PilB1和PilT1作为ATP水解酶, 可以形成六聚体结构, 并与质膜Ⅳ型菌毛合成复合体平台相互作用, 为菌毛伸长或缩短提供能量。Hfq可以与PilB1相互作用, 辅助PilB1促使Ⅳ型菌毛伸长Figure 2. Photosensing signal transduction pathways in phototaxis and synthesis model of type Ⅳ pili in SynechocystisSynechocystis cells sense and transduce light signal to regulate the synthesis of type Ⅳ pili through several pathways. PixJ1, UirS, PixD combine chromophore to sense and transduce light signal to downstream pathways. PixJ1 senses light and interacts with proteins encoded by genes in the same operon with pixJ1, and transduces the phosphate group to CheY-like proteins PixG and PixH, then PixG may regulate the synthesis of type Ⅳ pili through interacting with PilB1. UirS senses light and causes self phosphorylation, and transduces the phosphate group to UirR. The phosphorylation of UirR activates transcription of lsiR, and LsiR may regulate the synthesis of type Ⅳ pili through interacting with PilB1. PixD senses light and changes the way of interaction with PixE, and PixE can regulate the synthesis of type Ⅳ pili through interacting with PilB1. The signal transduction pathway composed by PilG-PilJ, PilL-N and PilL-C is similar to the pathway composed by PixJ1 and related proteins, but it lacks light sensing protein. The interaction between PixE and PilB1 had been confirmed, marked with solid line. The interactions between PilB1 and other CheY-like proteins still need to be experimentally verificated, marked with dotted line. PilC, PilM, PilN, PilO form platform complex in plasma membrane for the synthesis of type Ⅳ pili. The mature pili subunit PilA1 was formed through processing by PilD, added to the pili base at the position of PilC, and extended out of the cell through the pore-like structure of PilQ polymer located in the outer membrane. PilB1 and PilT1 are ATP hydrolases, and form hexamer structures, which interact with platform complex, and provide energy for the elongation or retraction of pili. Hfq assists the elongation of type Ⅳ pili by interacting with PilB1
图 2 集胞藻趋光运动感光信号转导及Ⅳ型菌毛合成模式图集胞藻通过多条信号转导途径感应光照信号并调控Ⅳ型菌毛合成。PixJ1、UirS和PixD均可结合发色团感应光照信号并将信号往下游传递。PixJ1响应光照信号后可能通过与其编码基因所在操纵子中其他编码蛋白相互作用, 最终将磷酸基团转移至CheY类似蛋白PixG及PixH、PixG可能通过与PilB1相互作用调控Ⅳ型菌毛合成。UirS感应光照信号后可自磷酸化, 并可将磷酸基团转移至UirR, UirR磷酸化后可以转录激活LsiR编码基因表达, LsiR可能通过与PilB1相互作用调控Ⅳ型菌毛合成。PixD感应光照信号后改变与PixE的相互作用方式, PixE可以与PilB1相互作用调控Ⅳ型菌毛合成。PilG-PilJ及PilL-N、PilL-C所形成的信号转导通路与PixJ1所在通路类似, 但该通路中缺少直接感光蛋白。PixE与PilB1的相互作用已被实验证实, 用实线表示, 其余CheY类似蛋白与PilB1相互作用未被实验证实, 用虚线表示。PilC、PilM、PilN和PilO在质膜上形成Ⅳ型菌毛合成复合体平台, PilA1在PilD的作用下形成成熟菌毛亚基, 在PilC处添加到Ⅳ型菌毛上, 并通过PilQ在外膜上形成多聚体孔状结构使Ⅳ型菌毛延伸至胞外。PilB1和PilT1作为ATP水解酶, 可以形成六聚体结构, 并与质膜Ⅳ型菌毛合成复合体平台相互作用, 为菌毛伸长或缩短提供能量。Hfq可以与PilB1相互作用, 辅助PilB1促使Ⅳ型菌毛伸长Figure 2. Photosensing signal transduction pathways in phototaxis and synthesis model of type Ⅳ pili in SynechocystisSynechocystis cells sense and transduce light signal to regulate the synthesis of type Ⅳ pili through several pathways. PixJ1, UirS, PixD combine chromophore to sense and transduce light signal to downstream pathways. PixJ1 senses light and interacts with proteins encoded by genes in the same operon with pixJ1, and transduces the phosphate group to CheY-like proteins PixG and PixH, then PixG may regulate the synthesis of type Ⅳ pili through interacting with PilB1. UirS senses light and causes self phosphorylation, and transduces the phosphate group to UirR. The phosphorylation of UirR activates transcription of lsiR, and LsiR may regulate the synthesis of type Ⅳ pili through interacting with PilB1. PixD senses light and changes the way of interaction with PixE, and PixE can regulate the synthesis of type Ⅳ pili through interacting with PilB1. The signal transduction pathway composed by PilG-PilJ, PilL-N and PilL-C is similar to the pathway composed by PixJ1 and related proteins, but it lacks light sensing protein. The interaction between PixE and PilB1 had been confirmed, marked with solid line. The interactions between PilB1 and other CheY-like proteins still need to be experimentally verificated, marked with dotted line. PilC, PilM, PilN, PilO form platform complex in plasma membrane for the synthesis of type Ⅳ pili. The mature pili subunit PilA1 was formed through processing by PilD, added to the pili base at the position of PilC, and extended out of the cell through the pore-like structure of PilQ polymer located in the outer membrane. PilB1 and PilT1 are ATP hydrolases, and form hexamer structures, which interact with platform complex, and provide energy for the elongation or retraction of pili. Hfq assists the elongation of type Ⅳ pili by interacting with PilB1Ⅳ型菌毛动态合成模型 关于Ⅳ型菌毛在细菌中具体如何伸长或收缩有两种模型解释。一种是旋转模型, 即PilC、PilM、PilN和PilO在质膜上形成菌毛合成平台复合体, 复合体面向细胞质部分嵌入PilB或PilT形成的六聚体腔内结构, 复合体位于质膜中部分位于动态合成的Ⅳ型菌毛基部。PilB或PilT六聚体结合并水解ATP时, 构象改变产生旋转运动, 带动质膜平台复合体蛋白及菌毛旋转, 并添加或解离下一个菌毛亚基, 使菌毛往外延伸或往内收缩[55, 56, 68]。另一种是压缩模型, 该模型认为PilC、PilM、PilN和PilO形成的菌毛合成质膜平台复合体作为控制菌毛亚基添加到菌毛上或从菌毛上解离下来的开关门。当平台处于开放状态时, 菌毛亚基可以结合到菌毛基部, 菌毛基部亚基也可以从菌毛上解离下来; 当PilB或PilT六聚体结合并水解ATP时, 促使平台处于关闭状态, 产生的挤压力使菌毛往外延伸或往内收缩, 同时平台面向另一角度开放, 并进行下个循环, 但该过程中菌毛本身不会发生旋转[54, 56]。
参与集胞藻光信号感应及传导相关蛋白 蓝藻实现趋光或避光运动的光信号转导途径一直是研究者力求解答的问题。近期研究表明球形单细胞蓝藻(如集胞藻)感光原理类似透镜聚光作用, 当单侧光透过细胞一侧后会汇聚于细胞背光侧表层并形成明亮光斑, 蓝藻中感光蛋白(蓝藻型光敏色素或其他感光蛋白)感应该处光斑, 并可能通过构象改变或与其他信号传递蛋白互作将光照信号往下游传递, 调控Ⅳ型菌毛在细胞表层的合成及分布, 可能使背光一侧Ⅳ型菌毛合成受阻, 向光一侧Ⅳ型菌毛合成增强, 最终实现趋光运动[69]。而当球形单细胞蓝藻表层靠近光源一侧接收光强强度大于细胞背光侧表层光斑时, 蓝藻中感光蛋白感应新的光信号并将信号往下游传递, 使细胞背光侧Ⅳ型菌毛合成增强, 细胞会发生避光运动[69]。集胞藻基因组上存在一些基因及基因簇在细胞感光并调控趋光运动方面发挥重要作用, 下面分别介绍。
集胞藻基因组上存在一个基因簇sll0038-sll0039-sll0040-sll0041-sll0042-sll0043, 其中基因命名为pixG(taxP1)-pixH (taxY1)-pixI (taxW1)-pixJ1 (taxD1)-pixJ2 (taxD’1)-pixL (taxAY1), 除pixI (taxW1)外, 该基因簇中基因突变会使细胞在单侧白光 [10 μmol photons/(m2·s)] 照射条件下产生避光运动(向与光源方向相反一侧运动), 而野生型细胞在相同条件下发生趋光运动[70, 71]。该基因簇编码蛋白与某些具有鞭毛的革兰氏阴性细菌(例如大肠杆菌)中调控细胞趋化运动信号转导相关蛋白有相似之处, 但也有明显不同。以大肠杆菌为例, 在趋化运动过程中具有MCP(Methyl-accepting chemotaxis protein)-signal和TarH结构域的蛋白通过与配体(化合物或结合化合物的蛋白)结合情况改变自身构象感应外界化学物质浓度变化, 通过CheW(Chemotactic W)与具有组氨酸激酶结构域可以自磷酸化的CheA相互作用, 促使CheA自磷酸化, CheA可以将磷酸化基团传递给CheY, 磷酸化的CheY可以与鞭毛动力蛋白相互作用, 调节鞭毛转动方向进而影响细胞运动方向[72]。集胞藻该基因簇中PixJ1(Phototaxis J1)/TaxD1(Chemotaxis-like D1)与PixJ2都含有MCP-signal结构域, 该结构域在细菌趋化运动中通过甲基化或去甲基化状态传递外界环境中化学物质浓度变化信号, 但氨基酸序列比对发现PixJ1和PixJ2所含MCP-signal结构域中缺少甲基化或去甲基化相关的几个关键氨基酸位点, 结合集胞藻突变株表型分析说明含该结构域蛋白在集胞藻趋光运动信号传递中同样发挥重要作用, 但具体信号转导途径与细菌趋化运动存在差异。PixJ1含有2个跨膜结构域, 可能定位于细胞质膜上, 这点与大肠杆菌中感应外界化学物质浓度的含MCP-signal和TarH结构域蛋白(例如Tar, Taxis toward aspartate and related amino acids)类似, 但PixJ1含有GAF(cGMP-specific phosphodiesterases, Adenylyl cyclases and FhlA, GAF)结构域并可结合发色团, 响应蓝光和绿光, 这与Tar含有TarH结构域结合配体感应外界化学物质浓度不同[73, 74]。集胞藻该基因簇中PixI(TaxW1)与CheW同源, pixI (taxW1)突变并不影响集胞藻细胞趋光运动, 说明其在集胞藻趋光运动信号传递过程中并非必需。PixL(TaxAY1)与CheA同源, PixG(TaxP1)、PixH(TaxY1)与CheY同源, 且这3个蛋白编码基因突变细胞会发生避光运动[70, 71], 说明这3个蛋白在集胞藻趋光运动信号传递中是必需的。PixG(TaxP1)在蓝藻中属于PatA家族蛋白, 除含有CheY具有的Response-reg结构域外, 还含有PATAN结构域, 该结构域的具体功能还不是十分清楚, 但近期研究表明含有该结构域的PixE(也属于PatA家族蛋白, 下文详细介绍)可以与集胞藻Ⅳ型菌毛合成动力蛋白PilB1直接相互作用, 调控Ⅳ型菌毛合成及蹭动方向[75], 说明集胞藻中PatA家族蛋白在趋光运动中所行使功能类似CheY在大肠杆菌趋化运动中的作用, 负责环境信号与动力蛋白的直接关联。PixJ1(TaxD1)、PixJ2 (TaxD′1)、PixL(TaxAY1)、PixG(TaxP1)和PixH(TaxY1)可以在集胞藻中形成感光信号转导通路, 并通过调控Ⅳ型菌毛合成促使细胞发生趋光运动行为。具体信号通路需要进一步研究。
集胞藻基因组上编码一个蓝光受体蛋白PixD(Phototaxis D, Slr1694), 该蛋白含有BLUF(Blue-Light Using FAD)结构域, 可以结合FAD(Flavin Adenine Dinucleotide)响应蓝光[76, 77]。敲除该基因后会使集胞藻在较弱单侧光[如5—100 μmol photons/(m2·s) 白光, 或0.7 μmol photons/(m2·s) 红光, 或5 μmol photons/(m2·s) 远红光、黄光与绿光]照射下产生避光运动, 而野生型细胞在相同条件下均发生趋光运动[76, 77], 说明pixD基因敲除会触发细胞在较弱单侧光照射下产生避光运动的信号转导。PixD作为蓝光受体, 其缺失突变株pixD-在单侧较弱光照条件下表型与pixJ1-突变株类似, 但它与PixJ1在调控细胞趋光运动中的信号转导机制显然不同。体内及体外蛋白相互作用实验结果表明, PixD可与其上游基因编码蛋白PixE(Phototaxis E, Slr1693)相互作用, 且其互作方式受光照条件而发生变化。在黑暗条件下, 二者可形成PixD10-PixE5(或PixD10-PixE4)复合体; 当接受强光 [约997 μmol photons/(m2·s) 白光] 照射后, 该复合体解聚为PixD二聚体及PixE单体, 暗示PixD与PixE在不同光照条件下的互作方式在集胞藻趋光或避光运动光信号传递中具有重要功能[77-80]。进一步研究发现, 在80 μmol photons/(m2·s) 单侧白光照射下, pixE-单突变株及pixD-E-双突变株均与野生型藻株发生类似的趋光运动, 而该条件下pixD-单突变株发生避光运动; 在243 μmol photons/(m2·s) 蓝光照射条件下野生型集胞藻会发生避光运动, 而pixD-E-双突变株会发生趋光运动, 结合PixD与PixE在不同光照条件下的差异互作方式, 推测PixE单体数量是细胞在单侧光照条件下发生趋光或避光运动的重要影响因素[81]。在单侧光照较弱时[如80 μmol photons/(m2·s) 单侧白光], 野生型细胞内PixD与PixE绝大多数以复合体的形式存在, 此时PixE无法与Ⅳ型菌毛合成动力蛋白PilB1相互作用[81], 细胞内感光蛋白(可能为PixJ1)感应单侧光照后通过信号转导途径调控细胞表层Ⅳ型菌毛在光照一侧合成相对增多, 促使细胞发生趋光运动。而在相同条件下, pixD敲除会使突变株细胞内PixE单体含量增加, PixE定位于细胞质膜周围可以直接与PilB1相互作用, 从而调控细胞表层Ⅳ型菌毛在背光一侧合成增多, 细胞发生避光运动[75, 81]。对比较弱单侧光照射, 在单侧较强蓝光照射时 [如243 μmol photons/(m2·s)], 野生型集胞藻内PixD感应蓝光后与PixE解聚, PixE单体含量增加会与PilB1相互作用, 从而调控细胞表层Ⅳ型菌毛在背光一侧合成相对增多, 细胞发生避光运动。该较强蓝光条件下, pixD-E-双突变株细胞内感光蛋白(可能为PixJ1)感应光照信号后, 通过信号转导途径调控细胞表层Ⅳ型菌毛在光照一侧合成相对增多, 细胞发生趋光运动。近期集胞藻单细胞水平感光实验结果表明: 在单侧超强蓝光[1200 μmol photons/(m2·s)]照射条件下, 野生型集胞藻细胞仍会发生避光运动, 而pixD-突变株细胞在该蓝光照射条件下随机蹭动, 未展现出运动方向性, 说明PixD在集胞藻细胞感应超强蓝光发生避光运动过程中具有重要作用[82]。在该条件下, 野生型集胞藻细胞发生避光运动的机制可能与在单侧较强蓝光照射时类似, 而pixD突变后为何细胞仅发生随机蹭动, 而不像在单侧较弱光照条件下时发生避光运动, 产生该现象的机制仍不清楚。综合上述结果可知, 集胞藻应对不同光质与光强而作出的细胞趋光或避光运动的调控方式精细而复杂。通过感受不同光信号实时改变PixD与PixE的互作方式, 进而精密调节PixE单体与Ⅳ型菌毛合成动力蛋白PilB1的相互作用, 从而调控Ⅳ型菌毛在细胞表层的分布情况, 实现细胞在不同条件下的趋光或避光运动。该信号转导通路组成蛋白与转导途径与某些具有鞭毛的革兰氏阴性细菌调控细胞趋化运动的信号转导通路显著不同, 是集胞藻为适应外界复杂光环境而形成的新的信号转导通路。
集胞藻基因组上存在一个基因簇slr1212-slr1213-slr1214在感应UV-A(Ultraviolet A)并调控细胞运动过程中发挥重要作用, 该基因簇三个基因分别命名为uirS-uirR-lsiR, 其中任何一个基因突变都会使细胞向单侧UV-A [25—100 μmol photons/(m2·s)] 照射方向运动(趋光运动), 而野生型集胞藻细胞在相同条件下向UV-A照射反方向运动(避光运动)[83]。该信号转导途径属于典型的二元信号转导途径, UirS(UV intensity response Sensor)预测含有4个跨膜结构域, 推测可能定位于细胞质膜上。其含有GAF结构域, 可以结合发色团后感应UV-A(或紫光)和绿光[83-85]。UirS 同时含有HisKA和HATPase-c结构域, 含有此类结构域的蛋白通常可以形成二聚体并催化自身组氨酸残基磷酸化, 并将该高能磷酸基团转移到具有Response-reg结构域的蛋白中。UirR为AraC家族转录调控因子, 含有Response-reg和HTH(Helix-Turn-Helix)结构域。蛋白相互作用实验证明UirS可以与UirR(UV intensity response Regulator)发生相互作用[83]。该基因簇中编码的另外一个蛋白LsiR(Light and stress integrating response Regulator)为PatA家族蛋白, 含有Response-reg及PATAN结构域, 该家族蛋白PixE已证实可以与集胞藻Ⅳ型菌毛合成动力蛋白PilB1相互作用, 调控细胞表层Ⅳ型菌毛合成及趋光运动方向[75], 推测LsiR与PixE功能类似。转录谱实验表明在85 μmol photons/(m2·s) 单侧UV-A照射时, uirS和uirR突变株中lsiR表达量显著低于野生型, 说明lsiR低表达是使突变株在该条件下趋光运动的主要原因[83]。综合以上实验结果推测, 在16 μmol photons/(m2·s)单侧绿光、红光或<5 μmol photons/(m2·s)UV-A照射时, UirS可以与UirR在质膜上形成复合体, 此时UirR无法结合到该基因簇中的lsiR基因启动子区, 使lsiR无法表达而发挥功能, 细胞表现出趋光运动表型。当UirS接收单侧较强UV-A [25—100 μmol photons/(m2·s)] 照射后, 构象发生改变并发生自磷酸化, UirR会与UirS分离并将磷酸基团转移至UirR中Response-reg结构域, 激活UirR转录活性, UirR结合到lsiR启动子区发挥转录激活功能[83], 合成LsiR通过与Ⅳ型菌毛合成动力蛋白PilB1相互作用, 调控Ⅳ型菌毛在背光侧动态合成从而发生避光运动。当然该信号转导通路中的一些具体细节例如UirS的自磷酸化以及将磷酸基团向UirR的转移, LsiR与PilB1的相互作用都需要进一步证实。
集胞藻中蓝藻型光敏色素Cph2(Cyanobacteria phytochrome like protein2)编码基因突变后细胞会向单侧蓝光 [0.45—1.3 μmol photons/(m2·s)] 或UV-A [10 μmol photons/(m2·s)] 照射方向运动, 而野生型细胞在相同条件下静止不动, 说明Cph2在抑制细胞向单侧蓝光及UV-A方向运动的过程中发挥功能[86, 87]。Cph2的N端GAF结构域可结合发色团后响应红光及远红光, C端GAF结构域结合发色团后响应蓝光及绿光[88-91]。同时Cph2的C端还含有二鸟苷酸环化酶结构域GGDEF及环化二鸟苷酸磷酸酯酶结构域EAL, 这两个结构域可以分别促进两个三磷酸鸟苷(GTP, Guanosine triphosphate)环化形成c-di-GMP或环化二鸟苷酸水解形成线性分子5′-pGpG, 从而调控胞内环化二鸟苷酸含量[91-93]。实验表明位于Cph2 C端的GAF和GGDEF结构域可以响应蓝光后催化c-di-GMP合成, 而胞内c-di-GMP含量增加会抑制细胞运动[91]。Cph2所含EAL结构域可以水解c-di-GMP, 降低其浓度[91]。Cph2通过感应光信号调控GGDEF和EAL结构域活性控制胞内c-di-GMP含量进而影响细胞运动[91]。近期研究表明Cph2可以通过调控胞内c-di-GMP浓度影响运动相关基因表达, 在5 μmol photons/(m2·s) 蓝光照射条件下野生型集胞藻中c-di-GMP浓度较5 μmol photons/(m2·s)绿光照射条件下显著上升, pilA5-pilA6基因表达显著下降, pilA9-pilA11表达量显著上升, 而cph2突变株在相同条件下胞内c-di-GMP浓度未发生显著变化, 野生型细胞在蓝光照射时运动相关基因表达模式的改变如何影响细胞运动仍需深入研究[94]。c-di-GMP含量升高后如何抑制集胞藻细胞运动的机制并不清楚。在大肠杆菌中, YcgR蛋白可以在结合c-di-GMP后与鞭毛动力蛋白相互作用, 降低鞭毛运动速率[95, 96]。而集胞藻中并不存在YcgR同源蛋白, 也未有关于调控运动相关蛋白可以结合c-di-GMP的报道。在霍乱弧菌(Vibrio cholerae)中研究表明MshE(Mannose-sensitive haemagglutinin E)型ATP酶可以与c-di-GMP结合[97], 而该蛋白在集胞藻中的同源蛋白即为Ⅳ型菌毛合成动力蛋白PilB1, 因此推测c-di-GMP通过与PilB1结合影响PilB1功能及Ⅳ型菌毛合成进而影响集胞藻运动。近期研究表明集胞藻中Slr1143蛋白含GGDEF结构域并可催化c-di-GMP合成, 该结构域可与Cph2的C端GGDEF及EAL结构域发生相互作用, slr1143突变后细胞可在较强 [40 μmol photons/(m2·s)] 红光(640 nm)条件下发生趋光运动, 而该实验所用野生型集胞藻株在相同条件下不会发生趋光运动[98]。Slr1143的C端含有GAF结构域, 其是否可以结合发色团响应红光并不清楚, 其在单侧强红光照射条件下抑制细胞运动的机制需要进一步探究。
综上所述, 集胞藻感应光照信号后通过多种信号转导途径将信号往下传递, 并最终调控Ⅳ型菌毛合成与分布进而控制细胞运动或静止(如通过Cph2控制胞内c-di-GMP含量调控)或产生趋光或避光运动(如通过PatA家族蛋白PixE、PixG、LsiR与Ⅳ型菌毛合成动力蛋白PilB1相互作用调控)。已报道光信号转导通路最终都是通过调控Ⅳ型菌毛合成蛋白PilB1实现对集胞藻蹭动调控, 研究表明PilB1在集胞藻运动过程中定位于运动方向一侧的细胞前端, 说明PilB1在细胞内的定位对细胞完成趋光或避光运动行为具有重要作用[99]。而调控Ⅳ型菌毛合成的另外一个动力蛋白PilT1是否参与到信号转导途径中需要进一步研究。集胞藻中还存在一个PilT1同源蛋白PilT2, PilT2编码基因突变后细胞在单侧白光照射条件下会产生避光运动[42], 而PilT2本身不含感光结构域, 其影响细胞趋光运动的机制也需要进一步揭示。集胞藻参与趋光运动感光与信号转导基因及功能在表 2中列出。
表 2 集胞藻参与趋光运动感光与信号转导基因及功能Table 2. Genes involved in light sensing and signal transduction of phototaxis in Synechocystis基因编号
Gene code基因名称
Gene name蛋白结构域
Domain功能概述
Function参与趋光运动感光基因 sll0041 pixJ1, taxD1 GAF, MCPsignal 感应蓝绿光, 参与趋光运动 sll0821 cph2 GAF, GGDEF, EAL, PHY 感应红光远红光及蓝绿光,
抑制细胞向蓝光和UV-A运动slr1694 pixD BLUF 感应蓝光, 参与趋光运动 slr1212 uirS GAF, PAS, HisKA, HATPase-c 感应UV-A与绿光,
参与UV-A照射下的避光运动参与趋光运动信号转导基因 sll0038 pixG,
taxP1Response-reg, PATAN 趋光运动信号转导 sll0039 pixH,
taxY1Response-reg 趋光运动信号转导 sll0042 pixJ2,
taxD’MCPsignal 趋光运动信号转导 sll0043 pixL,
taxAY1CheW, Response-regHATPase-c, Hpt 趋光运动信号转导 slr1143 cip1 GAF, GGDEF 抑制趋光运动信号转导 slr1693 pixE Response-reg, PATAN 趋光运动信号转导 slr1213 uirR Response-reg, HTH-18 转录调控因子, 避光运动信号转导 slr1214 lsiR Response-reg, PATAN 避光运动信号转导 sll1533 pilT2 T2SSE, AAA-16 趋光运动信号转导 其他调控集胞藻运动相关蛋白 集胞藻中还存在一些蛋白在细胞运动中起到重要作用, 这些蛋白并不直接参与Ⅳ型菌毛的组成或者装配过程, 但这些蛋白可以在转录或转录后水平上调控直接参与Ⅳ型菌毛的组成或者装配蛋白的表达及功能, 或者参与到影响细胞运动的其他过程。这些蛋白编码基因的突变均会使集胞藻细胞丧失运动能力。
Sigma因子 Bhaya等[29]报道集胞藻中SigF(Sigma factor F, SigF)对细胞运动是必需的, sigF突变株中Ⅳ型菌毛组成亚基PilA1编码基因表达量显著降低, 细胞表层Ⅳ型菌毛消失, 细胞丧失运动能力。
腺苷酸环化酶及cAMP依赖型转录调控因子 集胞藻中腺苷酸环化酶Cya1(Adenylate cyclase 1, Slr1991)突变也会使细胞丧失趋光运动能力, 而在外源添加cAMP(Cyclic Adenosine monophosphate)后, cya1突变株恢复趋光运动能力, 说明第二信使cAMP及腺苷酸环化酶Cya1在调控细胞趋光运动方面具有重要功能[100]。集胞藻中cAMP结合蛋白Sycrp1(Synechocystis cAMP Receptor Protein 1, Sycrp1)对细胞运动是必需的[49]。Sycrp1作为CRP(cAMP Receptor Protein)家族转录调控因子, 含有c-NMP binding结构域及HTH(Helix-Turn-Helix)结构域, 体外实验表明Sycrp1的c-NMP binding结构域可以结合cAMP[101]。sycrp1突变株中2个与集胞藻细胞运动相关的操纵子转录水平显著下调[102]。一个是编码Ⅳ型菌毛少量菌毛亚基蛋白的pilA9-pilA10-pilA11-slr2018操纵子[102], 该操纵子中PilA9、PilA10和PilA11直接参与到Ⅳ型菌毛装配过程中。另外一个是slr1667-slr1668操纵子, 该操纵子中的两个基因分别命名为cccS和cccP, 这两个基因的突变株表层Ⅳ型菌毛数显著减少并且均丧失了运动能力[102, 103]。免疫胶体金技术证明CccS主要定位于细胞表层, CccP主要定位于周质空间, 这2个蛋白可能对集胞藻细胞表层结构形成具有重要作用[103]。近期研究表明Slr1668可能作为一套转运系统的重要组分对少量Ⅳ型菌毛亚基分泌起作用[104]。Sycrp1可以直接结合到slr1667-slr1668操纵子启动子区域调控该启动子表达[102]。集胞藻中存在一个与Sycrp1具有较高同源性的蛋白Sycrp2(Synechocystis cAMP Receptor Protein 2), 体外实验表明Sycrp2中c-NMP binding 结构域无法与cAMP结合[101], 但sycrp2突变株也会影响集胞藻运动情况[94, 105]。进一步研究表明sycrp2突变株中pilA9-pilA10-pilA11-slr2018操纵子转录水平显著下调, 且Sycrp2可以与Sycrp1发生相互作用, Sycrp1可以直接结合到pilA9-pilA10-pilA11-slr2018操纵子的启动子区, Sycrp2与Sycrp1以异源二聚体的形式共同调控该操纵子表达, 进而影响细胞运动[105]。由上述实验结果可知, cAMP通过与CRP家族转录调控因子Sycrp1结合实现对影响Ⅳ型菌毛合成相关基因转录水平的调控是cAMP调控集胞藻趋光运动的一条重要途径。
信号转导蛋白 集胞藻基因组上存在一个操纵子slr1041-slr1042-slr1043-slr1044, 分别命名为pilG-pilH-pilI-pilJ。该操纵子中相关基因突变会影响细胞表层Ⅳ型菌毛合成进而影响细胞运动[106]。其中pilG突变会使细胞运动能力减弱, pilH、pilI和pilJ突变会使细胞丧失运动能力, pilH突变会使细胞表层Ⅳ型菌毛增多, pilI和pilJ突变会使细胞表层Ⅳ型菌毛显著减少[106]。PilG属于CheY类似蛋白, 并且属于PatA家族蛋白, PilH属于CheY类似蛋白, PilI属于CheW类似蛋白, PilJ为含有MCP结构域蛋白, 该操纵子编码蛋白种类与pixJ2所在操纵子类似, 但缺少直接感光蛋白, 其调控集胞藻运动机制需要进一步研究。另外, 依据蛋白序列同源性比对, 集胞藻中PilL同源蛋白由两个独立开放阅读框编码, 分别是pilL-N(slr0073)和pilL-C(slr0322)。pilL-N基因突变会使细胞表层Ⅳ型菌毛增多, 细胞运动能力丧失[106]。pilL-C基因突变会使细胞表层Ⅳ型菌毛几乎消失, 细胞运动能力丧失[106]。PilL-N含有Hpt(Histidine-containing phosphotransfer)结构域, 该结构域在二元信号转导过程中磷酸基团转移过程中发挥重要作用。PilL-N含有H-kinase_dim, HATPase_c及Response-reg结构域, 这些结构域在二元信号转导过程中组氨酸激酶二聚化, 自磷酸化及磷酸基团转移等方面具有重要功能。这两个蛋白具体怎样影响集胞藻中Ⅳ型菌毛合成并影响细胞运动需要进一步探究。
分子伴侣、ABC转运子、丝氨酸/苏氨酸蛋白激酶和未知功能蛋白。Bhaya等[107]利用转座子随机插入突变方式在集胞藻中筛选到很多丧失运动能力基因突变株, 这些基因包括sll0058、sll0415、sll0564、sll0565、sll1575, slr1301、slr1964、sll0183和sll0301。其他研究组也有关于基因突变导致集胞藻细胞丧失运动能力的报道, 例如sll1384和slr1443[108, 109]。这些基因分别编码不同类型蛋白。Sll0058属于分子伴侣蛋白Hsp70( Heat shock protein family 70)家族, Sll1384属于分子伴侣蛋白Hsp40( Heat shock protein family 40)家族, 这两个蛋白可能发生相互作用并共同影响细胞运动相关过程。Sll0415属于ABC(ATP Binding Cassette)转运子, 在集胞藻中可能参与转运影响运动相关蛋白。Sll1575(SpkA, Serine/threonine protein kinase A)和Slr1443(SpkE, Serine/threonine protein kinase E)属于真核生物丝氨酸/苏氨酸蛋白激酶类似蛋白, sll1575突变会使胞内少量菌毛亚基编码基因操纵子pilA9-pilA10-pilA11转录水平显著下调, 进而影响细胞运动[110, 111]。slr1443突变会使成熟PilA1的数量减少, 表明Slr1443可能在翻译后水平影响Ⅳ型菌毛亚基合成进而影响细胞运动[108]。Sll0564属于泛醌甲基转移酶类似蛋白。Sll0565编码基因在基因组上位于Sll0564编码基因下游, Sll0565富含谷氨酸和谷氨酰胺。Slr1301也富含谷氨酸和谷氨酰胺, 并可以形成螺旋线圈结构。Slr1964富含谷氨酰胺。Sll0183和Sll0301含有五肽重复结构域。这些蛋白多数属于功能未知蛋白, 含有一些未知功能结构域, 其影响细胞运动的具体机制需要进一步研究。
2.3 聚球藻参与趋光运动相关蛋白及其作用机制
聚球藻趋光运动机制研究较集胞藻少, Yang等[31]从野外环境中分离到一株聚球藻UTEX3055并对其趋光运动机制进行了深入研究。聚球藻UTEX3055基因组测序与聚球藻PCC7942相似度达98.5%, 但与聚球藻PCC7942不能发生运动不同, 该种藻可以发生趋光运动[31]。该藻基因组上一个由六个基因构成的基因簇UTEX3055_0945-0950, 对该藻的趋光运动具有重要作用[31]。该基因簇与集胞藻中sll0038-sll0043基因簇类似, 所编码蛋白为参与某些细菌趋化运动过程同源蛋白, 其中UTEX3055_0945-0946编码CheY类似蛋白, UTEX3055_0947、UTEX3055_0950编码CheW类似蛋白, UTEX3055_0948编码含有MCP及GAF等结构域蛋白, UTEX3055_0949编码CheA类似蛋白[31]。UTEX3055_0945-0946突变细胞丧失趋光运动能力, 在50 μmol photons/(m2·s)单侧白光照射条件下细胞并未向光源方向移动, 野生型细胞在相同条件下展现出趋光运动表型[31]。与集胞藻sll0038-sll0043基因簇中相关基因突变后表型不同, UTEX3055_0945-0946突变细胞并未展现出避光运动表型[31]。UTEX3055_0947, UTEX3055_0950突变并未影响细胞趋光运动能力[31]。UTEX3055_0948与UTEX3055_0949突变后细胞在50 μmol photons/(m2·s) 单侧白光照射条件下丧失趋光运动能力, 但未表现出避光运动表型[31]。UTEX3055_0948编码蛋白与集胞藻中PixJ1/TaxD1类似但并不相同, UTEX3055_0948编码蛋白N端含有5个GAF(编号GAF1-5)结构域, 且都包含在发色团结合及感光方面发挥重要作用的两个半胱氨酸残基[31]。体外表达GAF2发现其可以结合发色团响应蓝光和绿光[31]。通过组合敲除不同GAF结构域发现, 同时敲除5个GAF结构域细胞与UTEX3055_0948突变表型一致, 细胞在50 μmol photons/(m2·s)单侧白光照射条件下不会发生趋光或避光运动, 单独敲除GAF4或同时敲除GAF4及GAF5细胞在单侧白光照射条件下发生避光运动, 同时敲除GAF1-3或GAF2-4或GAF1-4或GAF1-3, GAF5细胞在单侧白光照射条件下表现出与野生型类似的趋光运动现象, 暗示聚球藻UTEX3055通过调控UTEX3055_0948编码蛋白中不同GAF结构域的功能调节细胞发生趋光或避光运动[31], 与集胞藻通过多条信号转导途径调节细胞发生趋光或避光运动相比, 该调节途径相对简单。聚球藻UTEX3055中是否存在其他调控细胞趋光运动的信号转导途径需要进一步探究。聚球藻UTEX3055细胞呈棒状, 但其感光机制可能与呈球状的集胞藻类似, 单侧光线从一侧射入经细胞类似透镜汇聚后落于细胞背光侧[31], 聚球藻UTEX3055细胞内感光蛋白(如UTEX3055_0948编码蛋白)感应光照信号并通过信号转导途径调控细胞发生趋光或避光运动。
2.4 蓝藻蹭动意义
蓝藻借助Ⅳ型菌毛伸缩沿固体表面发生蹭动响应外界环境变化并作出反应是长期进化形成的积极适应环境变化的有效策略。蓝藻作为自养型光合放氧生物, 对外界环境中光照及无机营养盐需求较高, 淡水单细胞蓝藻生存环境较开放海洋中聚球藻更为复杂多变, 因此其通过蹭动寻求更为有利的光照或营养环境对实现自身更好地生长尤为重要。对单细胞蓝藻趋光运动调控机制研究较为清晰, 单细胞蓝藻类似透镜感应光照方向并通过自身感光蛋白感应光照信号, 通过一系列信号转导途径调控细胞表层Ⅳ型菌毛合成及分布, 进而实现细胞趋光或避光运动, 体现了单细胞蓝藻响应光照并作出反应的精细调控过程。目前对蓝藻蹭动研究主要基于实验室数据, 并且多数为对单侧光照响应(趋光或避光运动), 蓝藻是否像某些细菌(如大肠杆菌)一样响应化学物质浓度存在趋化运动(利用Ⅳ型菌毛)反应需要进一步研究。在集胞藻中UirS除可以感应光信号外, 还可以与乙烯结合, 因此该蛋白又命名为SynEtr1(Synechocystis Ethylene Response1), SynEtr1结合乙烯的结构域为位于N端的3个跨膜结构域, SynEtr1结合乙烯后可以促进细胞向单侧光照方向移动[112], 说明集胞藻可以响应化学物质浓度影响趋光运动, 但集胞藻能否响应环境中不同浓度乙烯并通过运动作出反应并不清楚。另外, 在自然环境中对单细胞蓝藻能否通过蹭动有效改善自身生存环境, 促进自身生长也需要实验数据揭示。
3. 蓝藻滑动
3.1 丝状蓝藻滑动现象描述
蓝藻滑动通常指某些丝状蓝藻沿固体表面滑动的运动形式, 如某些颤藻目或念珠藻目中的丝状蓝藻沿固体表面滑动。这些丝状蓝藻在各个时期都会滑动, 并且其滑动时不会借助鞭毛或Ⅳ型菌毛, 藻丝一般在光滑表面上沿着丝状蓝藻长轴方向运行, 速率通常不超过11 μm/s[4, 113]。颤藻目和念珠藻目中的丝状蓝藻滑动形式并不完全一样, 颤藻科中某些蓝藻如颤藻属(Oscillatoria)、席藻属(Phormidium)和鞘丝藻属(Lyngbya)中的某些种类, 藻丝滑动的同时会发生沿藻丝长轴方向的旋转运动, 旋转运动方向与藻种类有关, 有些顺时针旋转, 如Phormidium uncinatum, 有些逆时针旋转如Oscillatoria princeps[114]。而念珠藻科某些蓝藻如鱼腥藻属(Anabaena)中的某些种类藻丝滑动时并不旋转, 而可以横向或侧向运动, 也可沿着底物表层按U型滑动[115]。丝状蓝藻在未受到外界环境刺激时, 可以沿某个方向运动5—8min, 然后转换方向运动[3, 113]。蓝藻滑动时通常伴随着黏液分泌, 覆盖丝状蓝藻整个表层, 并且会在蓝藻沿表层运动后留下印记[113, 116]。
3.2 丝状蓝藻滑动相关蛋白及作用机制
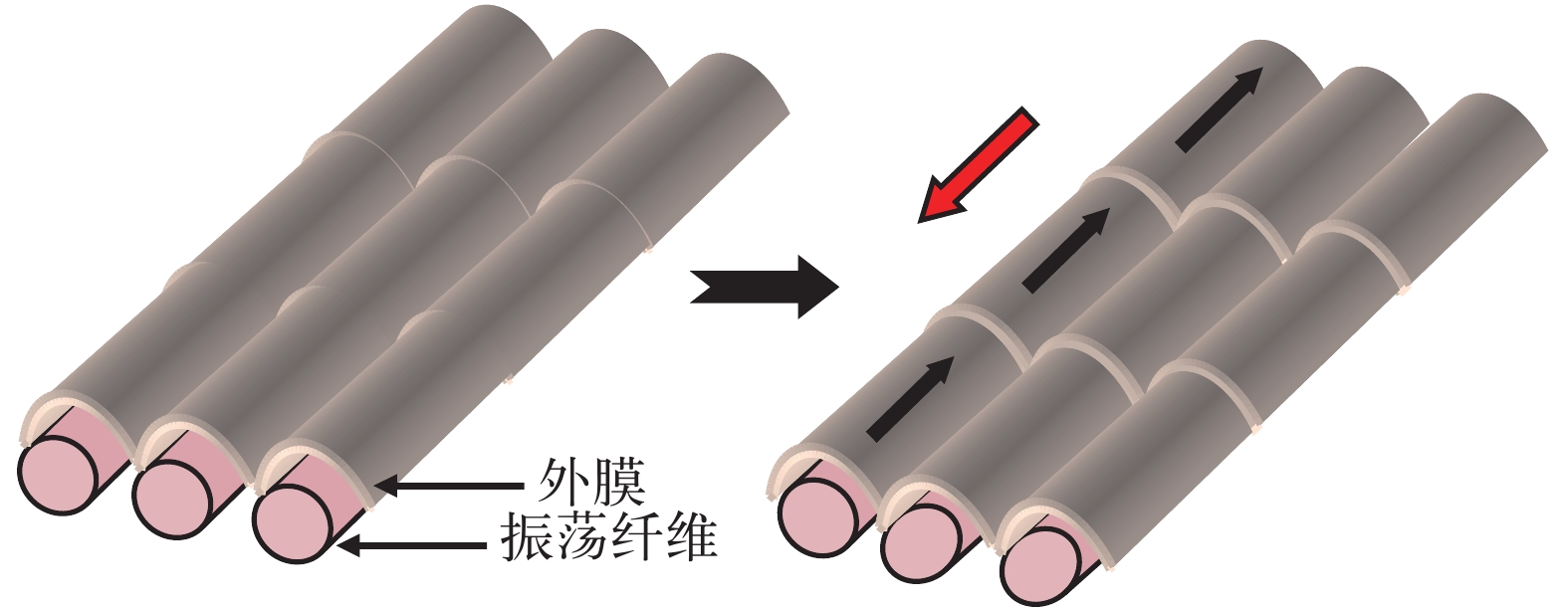
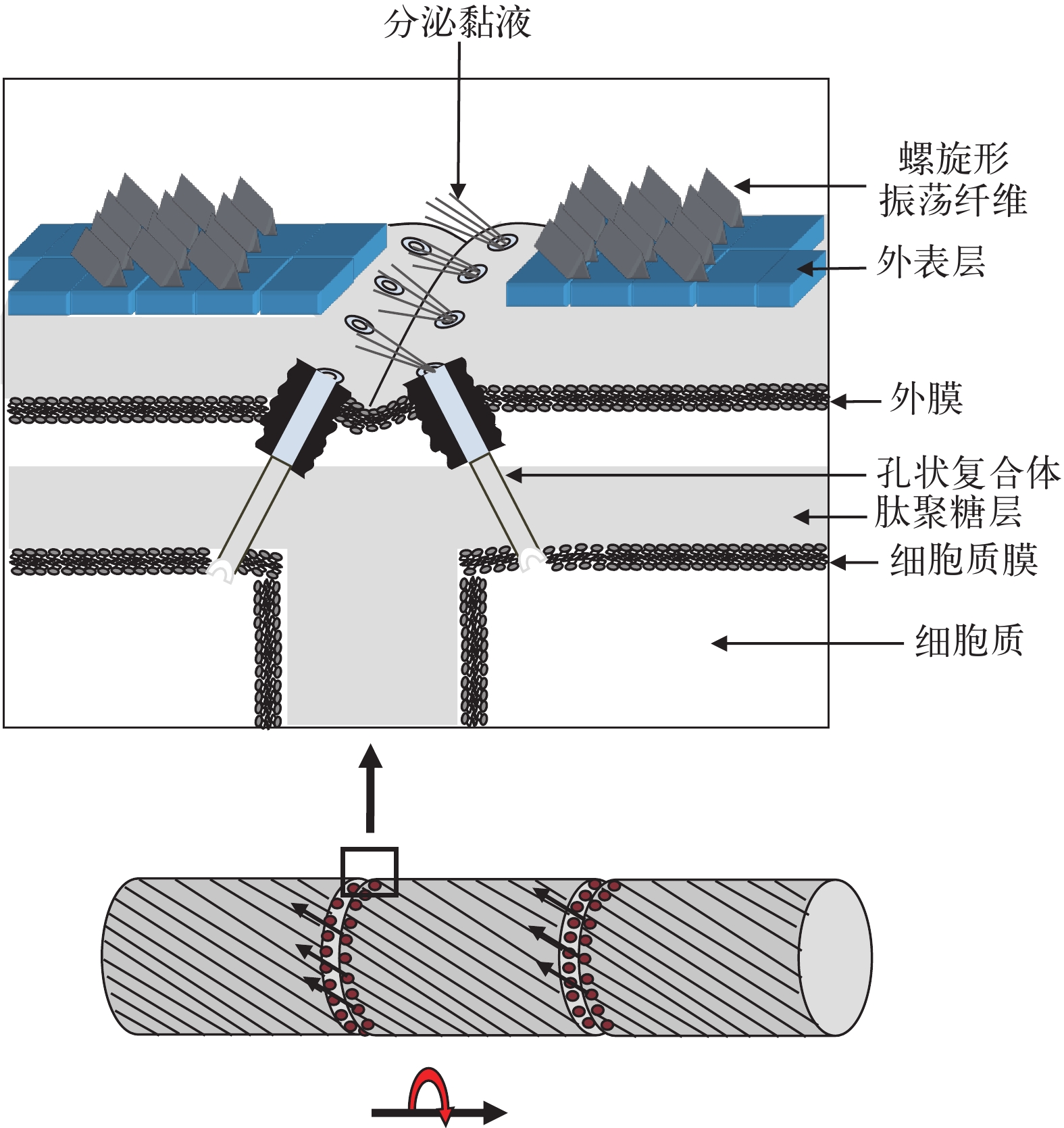
目前关于丝状蓝藻滑动现象产生机制有两种模型解释。一种是表层波动模型(图 3), 该模型提出基于的实验证据是颤藻属(Oscillatoria), 鞘丝藻属(Lyngbya), 席藻属(Phormidium)中某些种类藻外膜上存在呈螺旋状排列的纤维丝, 这些纤维丝在不同藻中的直径不尽相同(5—30 nm), 所处位置也不完全一样。有些位于肽聚糖层与外膜层之间, 例如Oscillatoria animalis, Oscillatoria strain A2, 有些位于外膜外层S-layer上, 例如Phormidium uncinatum。这些纤维丝收缩会在表层产生波动力, 进而使细胞沿着底物接触面滑动[114, 117-119]。席藻(Phormidium uncinatum)中纤维丝的组成成分已得以鉴定, 其表层纤维丝由一种称为振荡蛋白的糖蛋白构成, 该蛋白含有钙离子结合位点, 由646个氨基酸残基组成, 该蛋白以螺旋状排列方式定位于S-layer上[120]。表层波动模型中纤维丝收缩时的动力来源并不清楚, 有些种类纤维丝位于外膜外层S-layer上, 很难与提供质子动力或钠动力的细胞质膜产生关联。另外对于只沿藻丝长轴方向滑动而不发生自身转动的藻类其藻丝表层并不存在螺旋排列纤维, 这些藻类的滑动机理无法用该模型解释。另外一种解释丝状蓝藻滑动机制的模型是黏液喷出模型(图 4), 该模型基于的实验依据是一些丝状蓝藻细胞连接处存在一些孔状结构, 这些孔状结构按一定角度呈环形排列于细胞间隔膜处, 穿过细胞膜, 肽聚糖层及外膜[116]。研究者推测黏液就是由这些孔状结构喷出, 位于同一侧的孔状结构可以同时喷出黏液并产生推动力推动藻丝表层沿着底物表层向前滑动, 方向改变时位于另一侧的孔状结构同时喷出黏液使藻丝沿着底物表层向反方向运动[116]。由于某些种类藻丝表层存在螺旋状排列的纤维丝, 所以所喷出黏液可以沿螺旋状纤维丝流动, 使藻丝产生向前推动力的同时产生与藻丝长轴方向垂直的侧向力, 使藻丝发生旋转运动, 旋转运动方向与纤维丝排列方向有关[116]。黏液喷出提供动力使蓝藻滑动的机制应该与黄色黏球菌有相似之处。Wolgemuth等[121]认为黏液在细胞质膜处以脱水形式形成类似聚电解质凝胶, 然后进入孔状结构, 当从孔状结构分泌到胞外时, 骤然吸水膨胀进而产生推力。黏液喷出模型中缺少黏液从孔状复合体喷出的直接证据, 另外位于同一侧的孔状复合体如何协同作用喷出黏液并不清楚。对这2种模型的验证均需要更多的实验证据。
![]() 图 3 解释丝状蓝藻滑动的表层波动模型模式图位于丝状蓝藻细胞外膜下的振荡纤维可以收缩, 带动外膜及表层波动, 细胞可以沿与膜波动相反的方向运动。细黑色箭头表示外膜波动产生方向, 红色箭头表示细胞运动方向Figure 3. A model diagram to display surface waves producing by sliding filamentous cyanobacteria cellsThe oscillating fibers locating under outer membrane of filamentous cyanobacteria cells can contract and produce waves on outer membrane and surface. Cells can move in the opposite direction of membrane waves. Thin black arrows indicate the direction of outer membrane fluctuant waves, and the red arrow indicates the direction of cell movement
图 3 解释丝状蓝藻滑动的表层波动模型模式图位于丝状蓝藻细胞外膜下的振荡纤维可以收缩, 带动外膜及表层波动, 细胞可以沿与膜波动相反的方向运动。细黑色箭头表示外膜波动产生方向, 红色箭头表示细胞运动方向Figure 3. A model diagram to display surface waves producing by sliding filamentous cyanobacteria cellsThe oscillating fibers locating under outer membrane of filamentous cyanobacteria cells can contract and produce waves on outer membrane and surface. Cells can move in the opposite direction of membrane waves. Thin black arrows indicate the direction of outer membrane fluctuant waves, and the red arrow indicates the direction of cell movement![]() 图 4 解释丝状蓝藻滑动的黏液喷出模型模式图螺旋形排列的振荡纤维位于细胞外表层, 分泌黏液的孔状复合体位于细胞连接处, 该复合体穿过外膜和肽聚糖层并延伸到细胞质中。黏液从孔状复合体按如图所示的方向分泌出来, 沿螺旋形排列的振荡纤维流动, 黏液联系孔状复合体与底物表层, 使细胞朝黏液分泌相反的方向运动。细胞自身由于螺旋形排列的振荡纤维运动而旋转Figure 4. The slime extrusion model diagram for gliding in filamentous cyanobacteriaHelically arranged oscillating fibers are located in the outer layer of cells. The junctional pore complexes secreting slimes are located at the septum of cells, and the complexes pass through the outer membrane and peptidoglycan layer, and extends into the cytoplasm. Slimes are secreted from the pore complexes in the direction shown in the figure, and flow along the helically arranged oscillating fibers. Slimes connect junctional pore complex and substrate surface, and let cells move in the opposite direction of slimes’ secretion. The cell itself rotates due to the movement of the helically arranged oscillating fibers
图 4 解释丝状蓝藻滑动的黏液喷出模型模式图螺旋形排列的振荡纤维位于细胞外表层, 分泌黏液的孔状复合体位于细胞连接处, 该复合体穿过外膜和肽聚糖层并延伸到细胞质中。黏液从孔状复合体按如图所示的方向分泌出来, 沿螺旋形排列的振荡纤维流动, 黏液联系孔状复合体与底物表层, 使细胞朝黏液分泌相反的方向运动。细胞自身由于螺旋形排列的振荡纤维运动而旋转Figure 4. The slime extrusion model diagram for gliding in filamentous cyanobacteriaHelically arranged oscillating fibers are located in the outer layer of cells. The junctional pore complexes secreting slimes are located at the septum of cells, and the complexes pass through the outer membrane and peptidoglycan layer, and extends into the cytoplasm. Slimes are secreted from the pore complexes in the direction shown in the figure, and flow along the helically arranged oscillating fibers. Slimes connect junctional pore complex and substrate surface, and let cells move in the opposite direction of slimes’ secretion. The cell itself rotates due to the movement of the helically arranged oscillating fibers3.3 藻殖段滑动现象描述
藻殖段(Hormogonia)是某些丝状蓝藻在特定时期由长藻丝分化形成的短丝状体[122]。念珠藻目(Nostocales)和真枝藻目(Stigonematales)中某些属内的丝状蓝藻, 如念珠藻属(Nostoc), 侧生藻属(Fischerella)均可在特定环境下产生藻殖段。以点状念珠藻(Nostoc punctiforme)为例, 长丝状营养藻丝并不能运动, 但当外界环境变化时, 长丝状营养藻丝片段化并分化产生的藻殖段可以运动, 这与上文描述的颤藻属鱼腥藻属藻丝一直处于运动状态不同[123]。藻殖段运动发生在特定时期, 通常是分化形成的前48—72h, 而后停止运动分化产生异形胞并形成长丝状营养藻丝[123]。藻殖段滑动速度在0.5—3 μm/s, 运动有时会伴随沿藻丝长轴方向的转动, 藻殖段滑动过后也会留下黏液痕迹[124-126]。
3.4 藻殖段滑动相关蛋白及作用机制
Robinson等[126]报道真枝藻目中的层理鞭枝藻(Mastigocladus laminosus)所形成藻殖段可能利用细胞连接处呈环形排列的孔状结构喷出黏液提供动力使其滑动, 这类似于解释一直处于滑动状态的颤藻目和念珠藻目中某些藻类动力提供机制的黏液喷出模型。但该种藻所形成藻殖段细胞连接处的孔状结构与颤藻目中某些丝状藻类的孔状复合体是否为同一类结构并不清楚, 并且孔状复合体具体组成成分及形成机制需要提供更多实验证据。
某些念珠藻目蓝藻如Calothrix sp PCC 7601及点状念珠藻在藻殖段形成时, 细胞表层会形成Ⅳ型菌毛类似结构[125, 127], 暗示这些藻类藻殖段滑动可能与Ⅳ型菌毛有关。可进行遗传操作的点状念珠藻是探索藻殖段滑动机制的良好材料。点状念珠藻基因组中存在15种与Ⅳ型菌毛合成相关蛋白的编码基因, 这些基因多数在点状念珠藻产生藻殖段时显著上调表达[125, 128]。当插入失活pilA、pilB、pilT1和pilQ同源基因时藻殖段会丧失运动能力[125, 128], 这说明藻殖段运动需要Ⅳ型菌毛参与。点状念珠藻藻殖段细胞内Ⅳ型菌毛组成亚基PilA呈环状定位于发生滑动藻殖段细胞的细胞连接处, 并且藻殖段内每个细胞连接处的PilA通常定位于藻殖段同一侧, 暗示藻殖段滑动时位于细胞连接处同一侧的Ⅳ型菌毛会协同作用[128]。
点状念珠藻藻殖段运动及分化过程联系紧密, 受相关基因严格调控, 通常影响点状念珠藻藻殖段运动的基因对其分化过程也会产生影响。除上述Ⅳ型菌毛同源基因外, 点状念珠藻基因组上有两个基因簇与藻殖段运动及分化有关。一个是有5个基因组成的hmp(Hormogonia motility and polysaccharide)基因簇, 编码趋化运动信号转导相关蛋白的同源蛋白, 其中HmpA和HmpB为CheY类似蛋白, HmpC为CheW类似蛋白, HmpD为含有MCP结构域蛋白, HmpE为CheA类似蛋白, 突变株表型检测表明, 除HmpA外, HmpB-E对藻殖段运动及分化是必需的[129-130]。这些基因编码蛋白定位于藻殖段细胞连接处, 并呈环装排列, 可能参与调控Ⅳ型菌毛的合成[130]。另外一个基因簇为hps(Hormogonia polysaccharide secretion system)基因簇, 编码糖基转移蛋白和少量菌毛亚基同源蛋白[128]。该基因簇中hpsA-G对藻殖段运动都是必需的, hpsB-D编码少量菌毛亚基同源蛋白, 可能辅助Ⅳ型菌毛组成亚基PilA组装并发挥功能[128]。当在hpsE-G糖基转移酶编码基因突变株培养环境中添加培养过野生型点状念珠藻藻殖段培养基时, 其运动能力可得以恢复, 暗示藻殖段分泌多糖可以促进藻殖段运动, 而不是为藻殖段运动提供动力[128]。另外点状念珠藻基因组上编码的独立开放阅读框hpsH, 该基因编码蛋白与HpsB-D具有同源性, 也可能辅助Ⅳ型菌毛组成亚基PilA组装并发挥功能, 但hpsH突变藻殖段仍然可以运动[128]。
近期研究表明还有一些蛋白对点状念珠藻藻殖段运动或分化有重要作用, 例如HmpF、HmpU、HmpV、HmpW、OGTA(O-linked-β-N-acetylglucosamine transferase A)、SigC、SigF和SigJ[131-134]。点状念珠藻藻殖段中HmpF与菌毛组成亚基PilA具有类似的定位方式, 均定位于藻细胞的一端, hmpF突变株运动能力完全丧失, 且突变株中胞外PilA含量显著下降, 说明HmpF对点状念珠藻藻殖段Ⅳ型菌毛正常定位及功能发挥具有重要作用[131]。hmpU和hmpV突变株藻殖段运动受到抑制, 菌毛组成亚基PilA在胞外的积累减少; hmpW突变株藻殖段运动能力增强; hmpV突变后pilB、hmpF和hpsE等与藻殖段运动相关基因表达量显著下调; HmpW与HmpV可以发生相互作用, HmpU与HmpW分别含有丝氨酸磷酸酶结构域和丝氨酸激酶结构域, 它们可能通过调控HmpV的磷酸化状态从而影响下游与藻殖段运动相关基因表达或蛋白定位, 进而影响藻殖段运动[132]。OGTA是点状念珠藻乙酰氨基葡萄糖转移酶类似蛋白, 该蛋白编码基因突变会在转录后水平影响点状念珠藻藻殖段菌毛组成亚基PilA积累, 进而影响藻殖段运动[133]。点状念珠藻中SigC、SigF和SigJ编码基因突变均会使藻殖段完全丧失运动能力, sigC突变株中胞外菌毛组成亚基PilA积累量显著下降, sigF和sigJ突变株中胞内外菌毛组成亚基PilA含量均显著下降; SigJ可增强大多数藻殖段发育过程中特异性表达基因(包括Ⅳ型菌毛合成相关基因等)的转录表达, SigC主要影响细胞分裂相关基因表达, SigF主要调控菌毛组成亚基编码基因pilA的转录水平, 这些蛋白通过在转录水平直接或间接影响藻殖段运动或分化相关基因表达进而影响藻殖段运动或分化[134]。
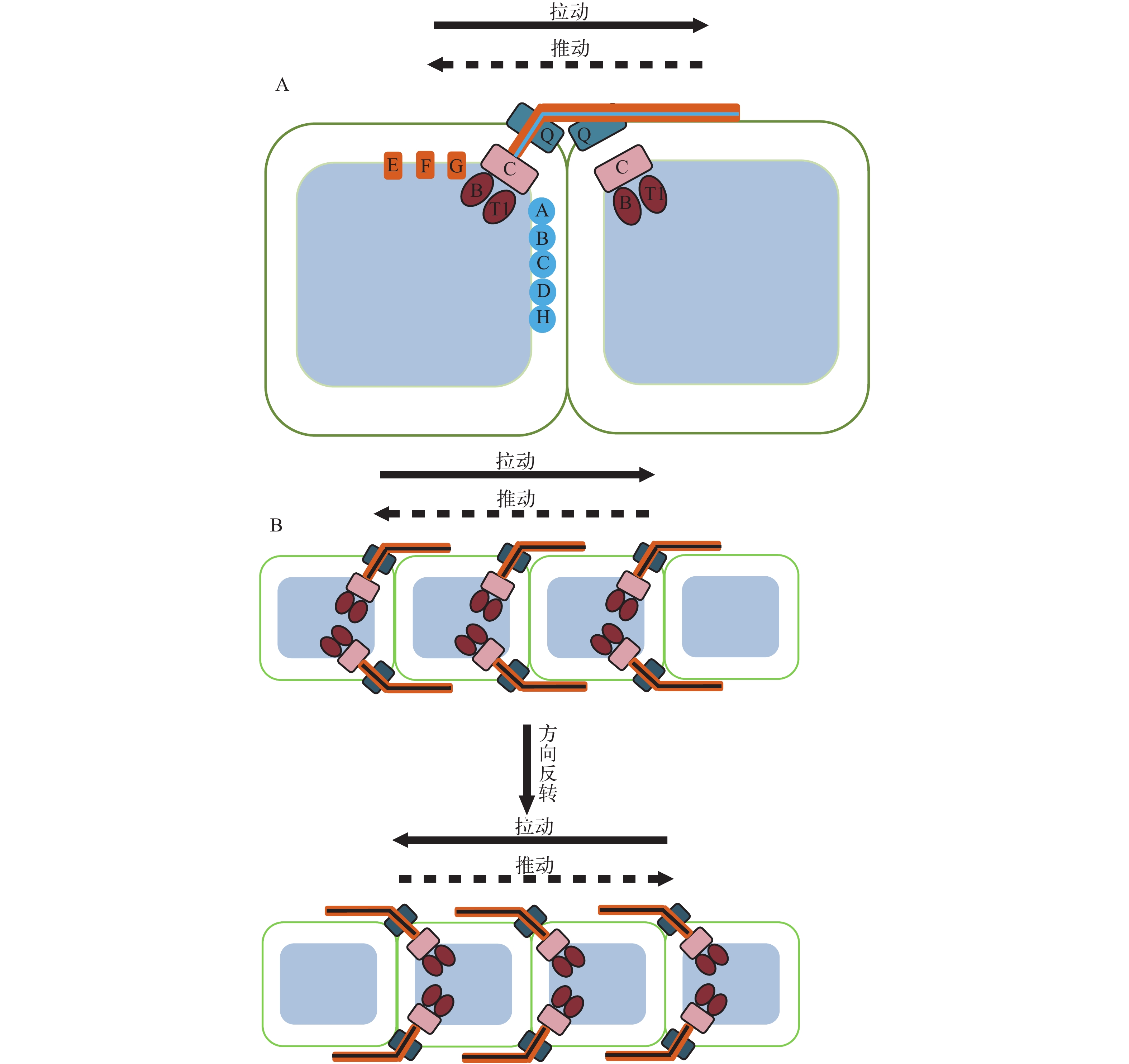
Khayatan等[128]对藻殖段运动机制提出了一种模型解释(图 5), 认为藻殖段细胞连接处的孔状结构由Ⅳ型菌毛合成及多糖分泌蛋白复合体构成, 藻殖段运动可能依靠定位于藻殖段细胞间连接孔复合体的Ⅳ型菌毛延长与收缩提供动力, 而菌毛与介质表层的黏附需要借助分泌至胞外的多糖实现。藻殖段细胞如何协同调控位于藻殖段细胞间连接孔复合体的Ⅳ型菌毛组装过程, 从而实现整个藻殖段的运动需要进一步研究。藻殖段滑动形式与丝状蓝藻相似, 而其滑动时借助Ⅳ型菌毛与集胞藻蹭动类似, 说明藻殖段滑动是一种相对特殊的运动形式。
![]() 图 5 丝状蓝藻藻殖段滑动模式图A. 藻殖段多糖由HPS-E、F和G(在图中表示为橙色E、F和G)合成并且在菌毛伸长缩短的过程中和菌毛一起经PilQ(在图中表示为暗蓝色Q)分泌出胞外。菌毛主要由PilA(在图中表示为亮蓝色A)构成, 也有可能包含其他少量菌毛亚基例如HPS-B、C、D和H(在图中表示为亮蓝色B、C、D和H)。多糖紧紧黏附菌毛和藻殖段细胞表层, 在同菌毛一起伸长缩短的过程中沿藻殖段长轴方向移动并促使细胞沿长轴方向滑动。PilC(在图中表示为粉色C)位于质膜上组成菌毛合成复合体平台, 并与PilB和PilT1(在图中表示为棕色B, T1)相互作用使菌毛伸长或缩短。若藻殖段运动方向与菌毛延伸方向相同, 表明通过菌毛拉动藻殖段细胞向前运动, 若藻殖段运动方向与菌毛延伸方向相反, 表明通过菌毛推动藻殖段细胞向前运动。B. 藻殖段细胞连接处位于每个细胞同一侧的菌毛多糖合成复合体协同作用, 使藻殖段朝一个方向滑动, 运动方向取决于发生协同作用的菌毛多糖合成复合体在细胞中所处位置Figure 5. A model of the hormogonium gliding motility in filamentous cyanobacteriaA. The polysaccharides of hormogonium are synthesized by HPS-E, F, G (shown as orange E, F, G in the figure), and secreted to the outside of the cell through PilQ (shown as dark blue Q in the figure) during the process of the pili elongation and retraction. The pili mainly consist of PilA (shown as bright blue A in the figure), may also consist of minor pilins, such as HPS-B, C, D, H (shown as bright blue B, C, D, H in the figure). Polysaccharides tightly adhere to the surface of pili and hormogonium cell, and move along the long axis of the cell during the process of the pili elongation and retraction, which helps to propel the cells to move along the long axis of the hormogonium. PilC (shown as pink C in the figure) located in the plasma membrane to form the pili synthesis complex platform, and interacts with PilB and PilT1 (shown as brown B, T1 in the figure) to promote the elongation and retraction of the pili. If the movement direction of the hormogonium cell is the same as the extension direction of the pili, it indicates that the pili pull hormogonium cells forward. If the movement direction of the hormogonium cell is in the opposite of the extension direction of the pili, it indicates that the pili push hormogonium cell forward. B. The complexes located at the junction and the same direction of each hormogonium cell synthesizes polysaccharides and pili synergistically, and make hormogonium move toward the same direction. The direction of movement depends on the position of the pili polysaccharide synthesis synergistic complexes in the cell
图 5 丝状蓝藻藻殖段滑动模式图A. 藻殖段多糖由HPS-E、F和G(在图中表示为橙色E、F和G)合成并且在菌毛伸长缩短的过程中和菌毛一起经PilQ(在图中表示为暗蓝色Q)分泌出胞外。菌毛主要由PilA(在图中表示为亮蓝色A)构成, 也有可能包含其他少量菌毛亚基例如HPS-B、C、D和H(在图中表示为亮蓝色B、C、D和H)。多糖紧紧黏附菌毛和藻殖段细胞表层, 在同菌毛一起伸长缩短的过程中沿藻殖段长轴方向移动并促使细胞沿长轴方向滑动。PilC(在图中表示为粉色C)位于质膜上组成菌毛合成复合体平台, 并与PilB和PilT1(在图中表示为棕色B, T1)相互作用使菌毛伸长或缩短。若藻殖段运动方向与菌毛延伸方向相同, 表明通过菌毛拉动藻殖段细胞向前运动, 若藻殖段运动方向与菌毛延伸方向相反, 表明通过菌毛推动藻殖段细胞向前运动。B. 藻殖段细胞连接处位于每个细胞同一侧的菌毛多糖合成复合体协同作用, 使藻殖段朝一个方向滑动, 运动方向取决于发生协同作用的菌毛多糖合成复合体在细胞中所处位置Figure 5. A model of the hormogonium gliding motility in filamentous cyanobacteriaA. The polysaccharides of hormogonium are synthesized by HPS-E, F, G (shown as orange E, F, G in the figure), and secreted to the outside of the cell through PilQ (shown as dark blue Q in the figure) during the process of the pili elongation and retraction. The pili mainly consist of PilA (shown as bright blue A in the figure), may also consist of minor pilins, such as HPS-B, C, D, H (shown as bright blue B, C, D, H in the figure). Polysaccharides tightly adhere to the surface of pili and hormogonium cell, and move along the long axis of the cell during the process of the pili elongation and retraction, which helps to propel the cells to move along the long axis of the hormogonium. PilC (shown as pink C in the figure) located in the plasma membrane to form the pili synthesis complex platform, and interacts with PilB and PilT1 (shown as brown B, T1 in the figure) to promote the elongation and retraction of the pili. If the movement direction of the hormogonium cell is the same as the extension direction of the pili, it indicates that the pili pull hormogonium cells forward. If the movement direction of the hormogonium cell is in the opposite of the extension direction of the pili, it indicates that the pili push hormogonium cell forward. B. The complexes located at the junction and the same direction of each hormogonium cell synthesizes polysaccharides and pili synergistically, and make hormogonium move toward the same direction. The direction of movement depends on the position of the pili polysaccharide synthesis synergistic complexes in the cell3.5 丝状蓝藻滑动意义
丝状蓝藻滑动较单细胞蓝藻泳动或蹭动形式及调控机制更为复杂, 涉及多个细胞协同作用。丝状蓝藻滑动是较复杂蓝藻对外部环境变化产生反应并积极适应外界环境的体现, 通过滑动可以响应外部环境中化学物质浓度和光照强度。研究表明Oscillatoria sp.可以在光照条件下产生向较高浓度二氧化碳、碳酸氢盐及氧气环境滑动的表型[135], Oscillatoria terebriformis滑动则受到环境中果糖及硫化物浓度升高的抑制[136, 137], 说明这些丝状蓝藻可以感应环境中物质浓度并作出反应。丝状蓝藻滑动通常受外部光照环境影响, Phonnidium uncinatum藻丝在变光环境下, 无论光照突然增强或减弱, 藻丝都会改变原来的滑动方向, 光信号的感应可能与跨类囊体膜的质子梯度有关, 钙离子在信号感应中也具有重要作用, 但具体机制还不清楚[138]。Anabaena variabilis藻丝在弱光条件下发生趋光运动, 在强光条件下发生避光运动, 该反应与单细胞藻类如集胞藻光反应类似, 但其光信号感应机制并不清楚[139]。这种对不同光照强度的感应及相应滑动方向的调整可以使藻丝在自然界中选择具有合适光照强度的区域生长, 避免光照过强或过弱影响藻丝生长。自然界中很多丝状蓝藻生活在一些处于极端环境例如热泉、高盐沙漠湖泊微生物垫的上层, 具有滑动能力能使其在微生物垫中平行或垂直移动, 从而在这样的微环境中寻求最利于自身生长的光照或营养盐环境, 例如, 在墨西哥Guerrero Negro[140]发现的高盐底栖微生物垫, 其中的丝状蓝藻Oscillatoria sp. 和Spirulinasubsalsa在白天光照较强时会迁移至微生物垫下层而在黄昏光照较弱时又迁移回表层; 而在法国Salins-de-Giraud[141]发现的高盐微生物垫中的丝状蓝藻Microcoleus chthonoplastes白天聚集并迁移至微生物垫上层, 而夜晚会均匀散开; 生活在美国休伦湖沉水坑主要由颤藻目丝状蓝藻形成的微生物垫中, 藻丝展现出较为明显趋光运动现象, 并且迁移至光区的丝状蓝藻光合作用产量较停留在暗区的丝状蓝藻高[142]。这些生活在不同环境中的丝状蓝藻感应外界环境变化, 并通过滑动改变自身位置获得利于自身的生长条件。
藻殖段作为某些丝状蓝藻营养藻丝响应外界营养盐浓度、光照等条件后形成的短丝状体, 其滑动行为对蓝藻在环境中的快速传播及与植物形成共生体具有重要作用[143]。有研究表明藻殖段对植物分泌物具有类似趋化运动的行为[143], 且藻殖段存在明显的趋光运动现象[144], 说明藻殖段可以有效感知外界环境信号并通过滑动做出响应。点状念珠藻藻殖段可在单侧白光照射条件下发生趋光运动, 其基因组上编码趋化运动信号转导同源蛋白的基因簇npF2161-npF2168与藻殖段趋光运动有关, 其中NpF2164和NpF2165是藻殖段趋光运动所必需的[142]。NpF2164含有MCP结构域及7个GAF结构域, 靠近C端的第7个GAF可以结合发色团感应橙光和绿光[145], 该蛋白可能感应外界光照并将信号往下游传递最终实现藻殖段趋光运动。该藻基因组上还存在其他编码趋化运动信号转导同源蛋白的基因簇, 这些基因簇是否调控藻殖段趋化运动需要进一步研究。以藻殖段滑动为基础的趋化运动及趋光运动行为对其适应外界多变环境具有重要作用。
4. 展望
从19世纪早期发现蓝藻运动现象至今, 关于蓝藻运动及其调控机制研究已取得很大进展。参考其他细菌运动调控机制, 以集胞藻为实验材料对蓝藻蹭动中的趋光运动机制研究最为深入, 关于蓝藻泳动及滑动, 研究者也提出了可能的作用机制模型。围绕蓝藻运动研究, 仍有许多问题需要研究者解答, 主要包括: (1)蓝藻蹭动现象中关于集胞藻的趋光运动信号转导过程相对清晰, 但仍有很多细节问题需要解答, 例如集胞藻中感光蛋白感应外界光信号后将信号传递下去, 通过CheY类似PatA家族蛋白(含PATAN结构域)与Ⅳ型菌毛合成动力蛋白PilB1相互作用影响Ⅳ型菌毛合成与分布, 该相互作用过程如何具体影响Ⅳ型菌毛合成? PatA家族蛋白与PilB1相互作用是促进Ⅳ型菌毛合成还是抑制Ⅳ型菌毛合成? 集胞藻中PatA家族蛋白有六个, 均含有Response-reg和PATAN结构域, 已报道实验数据表明该家族中PixE可以与PilB1相互作用, 另外五个是否都与PilB1相互作用? 该家族中PixE与LsiR对细胞在单侧光照下的避光运动至关重要, 而PixG对细胞单侧光照下的趋光运动至关重要, 如果它们都通过与PilB1相互作用调控Ⅳ型菌毛合成与分布进而实现集胞藻趋光或避光运动, 它们如何协调该过程? (2)信号分子环化腺苷酸(cAMP)及环化二鸟苷酸(c-di-GMP)在调控集胞藻蹭动中具体功能及作用机制是什么? cAMP是否只是通过与CRP类转录调控因子结合影响Ⅳ型菌毛合成相关蛋白编码基因转录水平影响集胞藻蹭动? c-di-GMP是否直接通过与PilB1或PilT1的结合影响集胞藻蹭动? (3)集胞藻中使运动能力丧失的各突变基因所编码蛋白在细胞运动过程中的具体功能是什么? (4)海洋聚球藻泳动不借助鞭毛或Ⅳ型菌毛, 其动力究竟如何产生? 实验数据表明培养基中钠离子和钙离子浓度对海洋聚球藻泳动至关重要, 钠动力和钙离子去极化如何协同产生推力需要进一步研究。SwmA和SwmB等泳动必需蛋白在泳动过程中的具体功能需要进一步揭示。两种关于促使海洋聚球藻泳动的波动机理解释模型需要寻找更多实验证据支持。(5)丝状蓝藻滑动过程中的动力如何产生? 关于丝状蓝藻滑动动力产生机制的两种模型解释需要提供更多实验证据。(6)藻殖段滑动与单细胞蓝藻蹭动均需要Ⅳ型菌毛参与, 但二者运动形式以及参与调控Ⅳ型菌毛合成的蛋白并不完全相同, 藻殖段中各细胞如何调控Ⅳ型菌毛合成过程进而实现整个藻殖段滑动需要进一步实验探究。(7)蓝藻运动在自然环境中的生态意义是什么? 能否有效地使蓝藻更好地适应环境? 如躲避高光或UV胁迫, 选择更合适的光照环境, 或通过运动获得更利于自身生长的营养环境。对部分丝状蓝藻在不同环境微生物垫中滑动研究发现, 这些丝状蓝藻通过滑动可以改变自身在微生物垫中所处位置, 从而改变获取光照条件进而增强光合效率。而对海洋聚球藻泳动, 淡水单细胞蓝藻蹭动以及藻殖段滑动过程, 仍然缺少证据表明这些藻类因为运动直接获益。经过研究者不断探索, 相信以上问题在不久的将来可以逐步解决。
-
图 1 海洋聚球藻细胞表层波动产生泳动的模式图[14,16—18]
A. 钙离子去极化过程使细胞外膜局部膨胀并回缩, 波动沿外膜表层扩散并产生推动力; B. 骨架蛋白复合体在胞内形成连续闭环轨道, 动力蛋白复合体形成跨质膜结构连接骨架蛋白和周质空间运动蛋白复合体, 动力蛋白复合体沿轨道运动带动周质空间运动蛋白复合体运动并使肽聚糖层及外膜变形膨胀并波动, 产生推动力推动细胞泳动
Figure 1. Models for causing surface waves and swimming of marine Synechococcus cells[14,16—18]
A. The process of calcium depolarization may serve to generate local swellings and waves on the cell surface, and the waves propagate along the surface to produce driving forces; B. Cytoskeletal protein complexes form a continuous helical track in the cell, and motor protein complexes form transmembrane structure which links cytoskeletal protein complexes and periplasmic space motility related protein complexes. Motor protein complexes travel along the helical track and drive the periplasmic space motility related protein complexes to cause deformations and local swellings of peptidoglycan layer and outer membrane, and subsequently produce surface waves and driving forces to propel cells to swim
图 2 集胞藻趋光运动感光信号转导及Ⅳ型菌毛合成模式图
集胞藻通过多条信号转导途径感应光照信号并调控Ⅳ型菌毛合成。PixJ1、UirS和PixD均可结合发色团感应光照信号并将信号往下游传递。PixJ1响应光照信号后可能通过与其编码基因所在操纵子中其他编码蛋白相互作用, 最终将磷酸基团转移至CheY类似蛋白PixG及PixH、PixG可能通过与PilB1相互作用调控Ⅳ型菌毛合成。UirS感应光照信号后可自磷酸化, 并可将磷酸基团转移至UirR, UirR磷酸化后可以转录激活LsiR编码基因表达, LsiR可能通过与PilB1相互作用调控Ⅳ型菌毛合成。PixD感应光照信号后改变与PixE的相互作用方式, PixE可以与PilB1相互作用调控Ⅳ型菌毛合成。PilG-PilJ及PilL-N、PilL-C所形成的信号转导通路与PixJ1所在通路类似, 但该通路中缺少直接感光蛋白。PixE与PilB1的相互作用已被实验证实, 用实线表示, 其余CheY类似蛋白与PilB1相互作用未被实验证实, 用虚线表示。PilC、PilM、PilN和PilO在质膜上形成Ⅳ型菌毛合成复合体平台, PilA1在PilD的作用下形成成熟菌毛亚基, 在PilC处添加到Ⅳ型菌毛上, 并通过PilQ在外膜上形成多聚体孔状结构使Ⅳ型菌毛延伸至胞外。PilB1和PilT1作为ATP水解酶, 可以形成六聚体结构, 并与质膜Ⅳ型菌毛合成复合体平台相互作用, 为菌毛伸长或缩短提供能量。Hfq可以与PilB1相互作用, 辅助PilB1促使Ⅳ型菌毛伸长
Figure 2. Photosensing signal transduction pathways in phototaxis and synthesis model of type Ⅳ pili in Synechocystis
Synechocystis cells sense and transduce light signal to regulate the synthesis of type Ⅳ pili through several pathways. PixJ1, UirS, PixD combine chromophore to sense and transduce light signal to downstream pathways. PixJ1 senses light and interacts with proteins encoded by genes in the same operon with pixJ1, and transduces the phosphate group to CheY-like proteins PixG and PixH, then PixG may regulate the synthesis of type Ⅳ pili through interacting with PilB1. UirS senses light and causes self phosphorylation, and transduces the phosphate group to UirR. The phosphorylation of UirR activates transcription of lsiR, and LsiR may regulate the synthesis of type Ⅳ pili through interacting with PilB1. PixD senses light and changes the way of interaction with PixE, and PixE can regulate the synthesis of type Ⅳ pili through interacting with PilB1. The signal transduction pathway composed by PilG-PilJ, PilL-N and PilL-C is similar to the pathway composed by PixJ1 and related proteins, but it lacks light sensing protein. The interaction between PixE and PilB1 had been confirmed, marked with solid line. The interactions between PilB1 and other CheY-like proteins still need to be experimentally verificated, marked with dotted line. PilC, PilM, PilN, PilO form platform complex in plasma membrane for the synthesis of type Ⅳ pili. The mature pili subunit PilA1 was formed through processing by PilD, added to the pili base at the position of PilC, and extended out of the cell through the pore-like structure of PilQ polymer located in the outer membrane. PilB1 and PilT1 are ATP hydrolases, and form hexamer structures, which interact with platform complex, and provide energy for the elongation or retraction of pili. Hfq assists the elongation of type Ⅳ pili by interacting with PilB1
图 3 解释丝状蓝藻滑动的表层波动模型模式图
位于丝状蓝藻细胞外膜下的振荡纤维可以收缩, 带动外膜及表层波动, 细胞可以沿与膜波动相反的方向运动。细黑色箭头表示外膜波动产生方向, 红色箭头表示细胞运动方向
Figure 3. A model diagram to display surface waves producing by sliding filamentous cyanobacteria cells
The oscillating fibers locating under outer membrane of filamentous cyanobacteria cells can contract and produce waves on outer membrane and surface. Cells can move in the opposite direction of membrane waves. Thin black arrows indicate the direction of outer membrane fluctuant waves, and the red arrow indicates the direction of cell movement
图 4 解释丝状蓝藻滑动的黏液喷出模型模式图
螺旋形排列的振荡纤维位于细胞外表层, 分泌黏液的孔状复合体位于细胞连接处, 该复合体穿过外膜和肽聚糖层并延伸到细胞质中。黏液从孔状复合体按如图所示的方向分泌出来, 沿螺旋形排列的振荡纤维流动, 黏液联系孔状复合体与底物表层, 使细胞朝黏液分泌相反的方向运动。细胞自身由于螺旋形排列的振荡纤维运动而旋转
Figure 4. The slime extrusion model diagram for gliding in filamentous cyanobacteria
Helically arranged oscillating fibers are located in the outer layer of cells. The junctional pore complexes secreting slimes are located at the septum of cells, and the complexes pass through the outer membrane and peptidoglycan layer, and extends into the cytoplasm. Slimes are secreted from the pore complexes in the direction shown in the figure, and flow along the helically arranged oscillating fibers. Slimes connect junctional pore complex and substrate surface, and let cells move in the opposite direction of slimes’ secretion. The cell itself rotates due to the movement of the helically arranged oscillating fibers
图 5 丝状蓝藻藻殖段滑动模式图
A. 藻殖段多糖由HPS-E、F和G(在图中表示为橙色E、F和G)合成并且在菌毛伸长缩短的过程中和菌毛一起经PilQ(在图中表示为暗蓝色Q)分泌出胞外。菌毛主要由PilA(在图中表示为亮蓝色A)构成, 也有可能包含其他少量菌毛亚基例如HPS-B、C、D和H(在图中表示为亮蓝色B、C、D和H)。多糖紧紧黏附菌毛和藻殖段细胞表层, 在同菌毛一起伸长缩短的过程中沿藻殖段长轴方向移动并促使细胞沿长轴方向滑动。PilC(在图中表示为粉色C)位于质膜上组成菌毛合成复合体平台, 并与PilB和PilT1(在图中表示为棕色B, T1)相互作用使菌毛伸长或缩短。若藻殖段运动方向与菌毛延伸方向相同, 表明通过菌毛拉动藻殖段细胞向前运动, 若藻殖段运动方向与菌毛延伸方向相反, 表明通过菌毛推动藻殖段细胞向前运动。B. 藻殖段细胞连接处位于每个细胞同一侧的菌毛多糖合成复合体协同作用, 使藻殖段朝一个方向滑动, 运动方向取决于发生协同作用的菌毛多糖合成复合体在细胞中所处位置
Figure 5. A model of the hormogonium gliding motility in filamentous cyanobacteria
A. The polysaccharides of hormogonium are synthesized by HPS-E, F, G (shown as orange E, F, G in the figure), and secreted to the outside of the cell through PilQ (shown as dark blue Q in the figure) during the process of the pili elongation and retraction. The pili mainly consist of PilA (shown as bright blue A in the figure), may also consist of minor pilins, such as HPS-B, C, D, H (shown as bright blue B, C, D, H in the figure). Polysaccharides tightly adhere to the surface of pili and hormogonium cell, and move along the long axis of the cell during the process of the pili elongation and retraction, which helps to propel the cells to move along the long axis of the hormogonium. PilC (shown as pink C in the figure) located in the plasma membrane to form the pili synthesis complex platform, and interacts with PilB and PilT1 (shown as brown B, T1 in the figure) to promote the elongation and retraction of the pili. If the movement direction of the hormogonium cell is the same as the extension direction of the pili, it indicates that the pili pull hormogonium cells forward. If the movement direction of the hormogonium cell is in the opposite of the extension direction of the pili, it indicates that the pili push hormogonium cell forward. B. The complexes located at the junction and the same direction of each hormogonium cell synthesizes polysaccharides and pili synergistically, and make hormogonium move toward the same direction. The direction of movement depends on the position of the pili polysaccharide synthesis synergistic complexes in the cell
表 1 集胞藻参与Ⅳ型菌毛合成基因及功能
Table 1 Genes and its functions involved in the synthesis of type Ⅳ pili in Synechocystis
基因编号
Code基因名称
Gene name功能概述
Functionsll1694 pilA1 Ⅳ型菌毛组成主要亚基 slr2015 pilA9 Ⅳ型菌毛组成辅助亚基 slr2016 pilA10 Ⅳ型菌毛组成辅助亚基 slr2017 pilA11 Ⅳ型菌毛组成辅助亚基 slr0063 pilB1 ATP水解酶, 作用于菌毛伸长 slr0161 pilT1 ATP水解酶, 作用于菌毛缩短 slr0162—0163 pilC Ⅳ型菌毛合成质膜平台复合体亚基 slr1120 pilD 菌毛亚基前导肽切除及N端甲基化 slr1274 pilM Ⅳ型菌毛合成质膜平台复合体亚基 slr1275 pilN Ⅳ型菌毛合成质膜平台复合体亚基 slr1276 pilO Ⅳ型菌毛合成质膜平台复合体亚基 slr1277 pilQ Ⅳ型菌毛合成外膜孔状复合体亚基 ssr3341 hfq 辅助菌毛伸长 表 2 集胞藻参与趋光运动感光与信号转导基因及功能
Table 2 Genes involved in light sensing and signal transduction of phototaxis in Synechocystis
基因编号
Gene code基因名称
Gene name蛋白结构域
Domain功能概述
Function参与趋光运动感光基因 sll0041 pixJ1, taxD1 GAF, MCPsignal 感应蓝绿光, 参与趋光运动 sll0821 cph2 GAF, GGDEF, EAL, PHY 感应红光远红光及蓝绿光,
抑制细胞向蓝光和UV-A运动slr1694 pixD BLUF 感应蓝光, 参与趋光运动 slr1212 uirS GAF, PAS, HisKA, HATPase-c 感应UV-A与绿光,
参与UV-A照射下的避光运动参与趋光运动信号转导基因 sll0038 pixG,
taxP1Response-reg, PATAN 趋光运动信号转导 sll0039 pixH,
taxY1Response-reg 趋光运动信号转导 sll0042 pixJ2,
taxD’MCPsignal 趋光运动信号转导 sll0043 pixL,
taxAY1CheW, Response-regHATPase-c, Hpt 趋光运动信号转导 slr1143 cip1 GAF, GGDEF 抑制趋光运动信号转导 slr1693 pixE Response-reg, PATAN 趋光运动信号转导 slr1213 uirR Response-reg, HTH-18 转录调控因子, 避光运动信号转导 slr1214 lsiR Response-reg, PATAN 避光运动信号转导 sll1533 pilT2 T2SSE, AAA-16 趋光运动信号转导 -
[1] Hedges S B, Chen H, Kumar S, et al. A genomic timescale for the origin of eukaryotes [J]. BMC Evolutionary Biology, 2001, 1(1): 4. doi: 10.1186/1471-2148-1-4
[2] Löffelhardt W, Bohnert H J. Molecular Biology of Cyanelles [M]. The Molecular Biology of Cyanobacteria. Dordrech: Springer, 1994: 65-89.
[3] Castenholz R W. Motility and Taxes [M]//Carr N G, Whitton B A (Eds.), The Biology of Cyanobacteria. Berkeley: University of California Press, 1982: 413-440.
[4] Castenholz R W. Order Nostocales [M]//Staley J T (Eds.), Bergey’s Manual of Systematic Bacteriology. Baltimore: Williams and Wilkins, 1989: 1780-1782.
[5] Semmler A B T, Whitchurch C B, Mattick J S. A re-examination of twitching motility in Pseudomonas aeruginosa [J]. Microbiology, 1999, 145(10): 2863-2873. doi: 10.1099/00221287-145-10-2863
[6] Zusman D R, Scott A E, Yang Z, et al. Chemosensory pathways, motility and development in Myxococcus xanthus [J]. Nature Reviews Microbiology, 2007, 5(11): 862. doi: 10.1038/nrmicro1770
[7] Brahamsha B, Bhaya D. Motility in Unicellular and Filamentous Cyanobacteria [M]//Flores E, Herrero A (Eds.), The Cell Biology of Cyanobacteria. Dorset: Caister Academic Press, 2013: 233-262.
[8] Waterbury J B, Willey J M, Franks D G, et al. A cyanobacterium capable of swimming motility [J]. Science, 1985, 230(4721): 74-76. doi: 10.1126/science.230.4721.74
[9] Brahamsha B. An abundant cell-surface polypeptide is required for swimming by the nonflagellated marine cyanobacterium Synechococcus [J]. Proceedings of the National Academy of Sciences, 1996, 93(13): 6504-6509. doi: 10.1073/pnas.93.13.6504
[10] McCarren J, Heuser J, Roth R, et al. Inactivation of swmA results in the loss of an outer cell layer in a swimming Synechococcus strain [J]. Journal of Bacteriology, 2005, 187(1): 224-230. doi: 10.1128/JB.187.1.224-230.2005
[11] McCarren J, Brahamsha B. SwmB, a 1.12-megadalton protein that is required for nonflagellar swimming motility in Synechococcus [J]. Journal of Bacteriology, 2007, 189(3): 1158-1162. doi: 10.1128/JB.01500-06
[12] McCarren J, Brahamsha B. Swimming motility mutants of marine Synechococcus affected in production and localization of the S-layer protein SwmA [J]. Journal of Bacteriology, 2009, 191(3): 1111-1114. doi: 10.1128/JB.01401-08
[13] Willey J M, Waterbury J B, Greenberg E P. Sodium-coupled motility in a swimming cyanobacterium [J]. Journal of Bacteriology, 1987, 169(8): 3429-3434. doi: 10.1128/JB.169.8.3429-3434.1987
[14] Pitta T P, Sherwood E E, Kobel A M, et al. Calcium is required for swimming by the nonflagellated cyanobacterium Synechococcus strain WH8113 [J]. Journal of Bacteriology, 1997, 179(8): 2524-2528. doi: 10.1128/JB.179.8.2524-2528.1997
[15] Ehlers K M, Samuel A D, Berg H C, et al. Do cyanobacteria swim using traveling surface waves [J]? Proceedings of the National Academy of Sciences of the United States of America, 1996, 93(16): 8340-8343.
[16] Sun M, Wartel M, Cascales E, et al. Motor-driven intracellular transport powers bacterial gliding motility [J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(18): 7559-7564. doi: 10.1073/pnas.1101101108
[17] Ehlers K, Oster G. On the mysterious propulsion of Synechococcus [J]. PLoS One, 2012, 7(5): e36081. doi: 10.1371/journal.pone.0036081
[18] Nan B, Zusman D R. Novel mechanisms power bacterial gliding motility [J]. Molecular Microbiology, 2016, 101(2): 186-193. doi: 10.1111/mmi.13389
[19] Waterbury J B, Watson S W, Valois F W, et al. Biological and ecological characterization of the marine unicellular cyanobacterium Synechococcus [J]. Canadian Journal of Fisheries and Aquatic Sciences, 1986, 214: 71-120.
[20] Eppley R W, Renger E H, Harrison W G. Nitrate and phytoplankton production in southern California coastal waters [J]. Limnology and Oceanography, 1979, 24(3): 483-494. doi: 10.4319/lo.1979.24.3.0483
[21] Eppley R W, Render E H, Harrison W G, et al. Ammonium distribution in southern California coastal waters and its role in the growth of phytoplankton [J]. Limnology and Oceanography, 1979, 24(3): 495-509. doi: 10.4319/lo.1979.24.3.0495
[22] Goldman J C. Conceptual role for microaggregates in pelagic waters [J]. Bulletin of Marine Science, 1984, 35(3): 462-476.
[23] Willey J M, Waterbury J B. Chemotaxis toward nitrogenous compounds by swimming strains of marine Synechococcus spp [J]. Applied and Environment Microbiology, 1989, 55(8): 1888-1894. doi: 10.1128/AEM.55.8.1888-1894.1989
[24] Willey J M. Characterization of swimming motility in a marine unicellular cyanobacterium [D]. Cambridge: Woods Hole Oceanographic Institution and Massachusetts Institute of Technology, 1988: 1-162.
[25] Strom S, Bright K, Fredrickson K, et al. The Synechococcus cell surface protein SwmA increases vulnerability to predation by flagellates and ciliates [J]. Limnology and Oceanography, 2017, 62(2): 784-794. doi: 10.1002/lno.10460
[26] Rippka R, Deruelles J, Waterbury J B, et al. Generic assignments, strain histories and properties of pure cultures of cyanobacteria [J]. Microbiology, 1979, 111(1): 1-61. doi: 10.1099/00221287-111-1-1
[27] Stanier R Y, Kunisawa R, Mandel M, et al. Purification and properties of unicellular blue-green algae (order Chroococcales) [J]. Bacteriological Reviews, 1971, 35(2): 171-205. doi: 10.1128/MMBR.35.2.171-205.1971
[28] Choi J S, Chung Y H, Moon Y J, et al. Photomovement of the gliding cyanobacterium Synechocystis sp. PCC 6803 [J]. Photochemistry and Photobiology, 1999, 70(1): 95-102. doi: 10.1111/j.1751-1097.1999.tb01954.x
[29] Bhaya D, Watanabe N, Ogawa T, et al. The role of an alternative sigma factor in motility and pilus formation in the cyanobacterium Synechocystis sp. strain PCC6803 [J]. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(6): 3188-3193. doi: 10.1073/pnas.96.6.3188
[30] Kondou Y, Nakazawa M, Higashi S, et al. Equal-quantum action spectra indicate fluence-rate–selective action of multiple photoreceptors for photomovement of the thermophilic cyanobacterium Synechococcus elongatus [J]. Photochemistry and Photobiology, 2001, 73(1): 90-95. doi: 10.1562/0031-8655(2001)073<0090:EQASIF>2.0.CO;2
[31] Yang Y, Lam V, Adomako M, et al. Phototaxis in a wild isolate of the cyanobacterium Synechococcus elongatus [J]. Proceedings of the National Academy of Sciences, 2018, 115(52): E12378-E12387. doi: 10.1073/pnas.1812871115
[32] Ng W O, Grossman A R, Bhaya D. Multiple light inputs control phototaxis in Synechocystis sp. strain PCC6803 [J]. Journal of Bacteriology, 2003, 185(5): 1599-1607. doi: 10.1128/JB.185.5.1599-1607.2003
[33] Fiedler B, Börner T, Wilde A. Phototaxis in the cyanobacterium Synechocystis sp. PCC 6803: role of different photoreceptors [J]. Photochemistry and Photobiology, 2005, 81(6): 1481-1488. doi: 10.1562/2005-06-28-RA-592
[34] Chau R M W, Bhaya D, Huang K C. Emergent phototactic responses of cyanobacteria under complex light regimes [J]. MBio, 2017, 8(2): e02330-16.
[35] Kong S G, Arai Y, Suetsugu N, et al. Rapid severing and motility of chloroplast-actin filaments are required for the chloroplast avoidance response in Arabidopsis [J]. The Plant Cell, 2013, 25(2): 572-590. doi: 10.1105/tpc.113.109694
[36] Wall D, Kaiser D. Type IV pili and cell motility [J]. Molecular Microbiology, 1999, 32(1): 1-10. doi: 10.1046/j.1365-2958.1999.01339.x
[37] Youderian P. Bacterial motility: secretory secrets of gliding bacteria [J]. Current Biology, 1998, 8(12): R408-R411. doi: 10.1016/S0960-9822(98)70264-7
[38] Merz A J, So M, Sheetz M P. Pilus retraction powers bacterial twitching motility [J]. Nature, 2000, 407(6800): 98-102. doi: 10.1038/35024105
[39] Skerker J M, Berg H C. Direct observation of extension and retraction of type IV pili [J]. Proceedings of the National Academy of Sciences, 2001, 98(12): 6901-6904. doi: 10.1073/pnas.121171698
[40] Maier B, Potter L, So M, et al. Single pilus motor forces exceed 100 pN [J]. Proceedings of the National Academy of Sciences, 2002, 99(25): 16012-16017. doi: 10.1073/pnas.242523299
[41] Chang Y W, Rettberg L A, Treuner-Lange A, et al. Architecture of the type IVa pilus machine [J]. Science, 2016, 351(6278): 2001. doi: 10.1126/science.aad2001
[42] Bhaya D, Bianco N R, Bryant D, et al. Type IV pilus biogenesis and motility in the cyanobacterium Synechocystis sp. PCC6803 [J]. Molecular Microbiology, 2000, 37(4): 941-951. doi: 10.1046/j.1365-2958.2000.02068.x
[43] Chen Z, Li X T, Tan X M, et al. Recent advances in biological functions of thick pili in the cyanobacterium Synechocystis sp. PCC 6803 [J]. Frontiers in Plant Science, 2020(11): 241.
[44] Yoshihara S, Geng X X, Okamoto S, et al. Mutational analysis of genes involved in pilus structure, motility and transformation competency in the unicellular motile cyanobacterium Synechocystis sp. PCC6803 [J]. Plant and Cell Physiology, 2001, 42(1): 63-73. doi: 10.1093/pcp/pce007
[45] Strom M S, Lory S. Amino acid substitutions in pilin of Pseudomonas aeruginosa. Effect on leader peptide cleavage, amino-terminal methylation, and pilus assembly [J]. Journal of Biological Chemistry, 1991, 266(3): 1656-1664.
[46] Strom M S, Lory S. Kinetics and sequence specificity of processing of prepilin by PilD, the type IV leader peptidase of Pseudomonas aeruginosa [J]. Journal of Bacteriology, 1992, 174(22): 7345-7351. doi: 10.1128/JB.174.22.7345-7351.1992
[47] Lory S, Strom M S. Structure-function relationship of type-IV prepilin peptidase of Pseudomonas aeruginosa–a review [J]. Gene, 1997, 192(1): 117-121. doi: 10.1016/S0378-1119(96)00830-X
[48] Mattick J S. Type IV pili and twitching motility [J]. Annual Reviews of Microbiology, 2002, 56(1): 289-314. doi: 10.1146/annurev.micro.56.012302.160938
[49] Yoshimura H, Yoshihara S, Okamoto S, et al. A cAMP receptor protein, SYCRP1, is responsible for the cell motility of Synechocystis sp. PCC 6803 [J]. Plant and Cell Physiology, 2002, 43(4): 460-463. doi: 10.1093/pcp/pcf050
[50] Nguyen Y, Sugiman-Marangos S, Harvey H, et al. Pseudomonas aeruginosa minor pilins prime type IVa pilus assembly and promote surface display of the PilY1 adhesin [J]. Journal of Biological Chemistry, 2015, 290(1): 601-611. doi: 10.1074/jbc.M114.616904
[51] Whitchurch C B, Hobbs M, Livingston S P, et al. Characterisation of a Pseudomonas aeruginosa twitching motility gene and evidence for a specialised protein export system widespread in eubacteria [J]. Gene, 1991, 101(1): 33-44. doi: 10.1016/0378-1119(91)90221-V
[52] Satyshur K A, Worzalla G A, Meyer L S, et al. Crystal structures of the pilus retraction motor PilT suggest large domain movements and subunit cooperation drive motility [J]. Structure, 2007, 15(3): 363-376. doi: 10.1016/j.str.2007.01.018
[53] Misic A M, Satyshur K A, Forest K T. P. aeruginosa PilT structures with and without nucleotide reveal a dynamic type IV pilus retraction motor [J]. Journal of Molecular Biology, 2010, 400(5): 1011-1021. doi: 10.1016/j.jmb.2010.05.066
[54] Mancl J M, Black W P, Robinson H, et al. Crystal structure of a type IV pilus assembly ATPase: insights into the molecular mechanism of PilB from Thermus thermophilus [J]. Structure, 2016, 24(11): 1886-1897. doi: 10.1016/j.str.2016.08.010
[55] McCallum M, Tammam S, Khan A, et al. The molecular mechanism of the type IVa pilus motors [J]. Nature Communications, 2017(8): 15091.
[56] Craig L, Forest K T, Maier B. Type IV pili: dynamics, biophysics and functional consequences [J]. Nature Reviews Microbiology, 2019, 17(7): 429-440. doi: 10.1038/s41579-019-0195-4
[57] Aim R A, Hallinan J P, Watson A A, et al. Fimbrial biogenesis genes of Pseudomonas aeruginosa: pilW and pilX increase the similarity of type 4 fimbriae to the GSP protein-secretion systems and pilY1 encodes a gonococcal PilC homologue [J]. Molecular Microbiology, 1996, 22(1): 161-173. doi: 10.1111/j.1365-2958.1996.tb02665.x
[58] Wolfgang M, Park H S, Hayes S F, et al. Suppression of an absolute defect in type IV pilus biogenesis by loss-of-function mutations in pilT, a twitching motility gene in Neisseria gonorrhoeae [J]. Proceedings of the National Academy of Sciences, 1998, 95(25): 14973-14978. doi: 10.1073/pnas.95.25.14973
[59] Scheuerpflug I, Rudel T, Ryll R, et al. Roles of PilC and PilE proteins in pilus-mediated adherence of Neisseria gonorrhoeae and Neisseria meningitidis to human erythrocytes and endothelial and epithelial cells [J]. Infection and Immunity, 1999, 67(2): 834-843. doi: 10.1128/IAI.67.2.834-843.1999
[60] Martin P R, Watson A A, McCaul T F, et al. Characterization of a five-cluster required for the biogenesis of type 4 fimbriae in Pseudomonas aeruginosa [J]. Molecular Microbiology, 1995, 16(3): 497-508. doi: 10.1111/j.1365-2958.1995.tb02414.x
[61] Ayers M, Sampaleanu L M, Tammam S, et al. PilM/N/O/P proteins form an inner membrane complex that affects the stability of the Pseudomonas aeruginosa type IV pilus secretin [J]. Journal of Molecular Biology, 2009, 394(1): 128-142. doi: 10.1016/j.jmb.2009.09.034
[62] McCallum M, Tammam S, Little D J, et al. PilN binding modulates the structure and binding partners of the Pseudomonas aeruginosa type IVa pilus protein PilM [J]. Journal of Biological Chemistry, 2016, 291(21): 11003-11015. doi: 10.1074/jbc.M116.718353
[63] Martin P R, Hobbs M, Free P D, et al. Characterization of pilQ, a new gene required for the biogenesis of type 4 fimbriae in Pseudomonas aeruginosa [J]. Molecular Microbiology, 1993, 9(4): 857-868. doi: 10.1111/j.1365-2958.1993.tb01744.x
[64] Bitter W, Koster M, Latijnhouwers M, et al. Formation of oligomeric rings by XcpQ and PilQ, which are involved in protein transport across the outer membrane of Pseudomonas aeruginosa [J]. Molecular Microbiology, 1998, 27(1): 209-219. doi: 10.1046/j.1365-2958.1998.00677.x
[65] Dienst D, Dühring U, Mollenkopf H J, et al. The cyanobacterial homologue of the RNA chaperone Hfq is essential for motility of Synechocystis sp. PCC 6803 [J]. Microbiology, 2008, 154(10): 3134-3143. doi: 10.1099/mic.0.2008/020222-0
[66] Schuergers N, Ruppert U, Watanabe S, et al. Binding of the RNA chaperone Hfq to the type IV pilus base is crucial for its function in Synechocystis sp. PCC 6803 [J]. Molecular Microbiology, 2014, 92(4): 840-852. doi: 10.1111/mmi.12595
[67] Schuergers N, Wilde A. Appendages of the cyanobacterial cell [J]. Life, 2015, 5(1): 700-715. doi: 10.3390/life5010700
[68] Solanki V, Kapoor S, Thakur K G. Structural insights into the mechanism of Type IVa pilus extension and retraction ATPase motors [J]. The FEBS Journal, 2018, 285(18): 3402-3421. doi: 10.1111/febs.14619
[69] Schuergers N, Lenn T, Kampmann R, et al. Cyanobacteria use micro-optics to sense light direction [J]. Elife, 2016(5): e12620.
[70] Yoshihara S, Suzuki F, Fujita H, et al. Novel putative photoreceptor and regulatory genes required for the positive phototactic movement of the unicellular motile cyanobacterium Synechocystis sp. PCC 6803 [J]. Plant and Cell Physiology, 2000, 41(12): 1299-1304. doi: 10.1093/pcp/pce010
[71] Bhaya D, Takahashi A, Grossman A R. Light regulation of type IV pilus-dependent motility by chemosensor-like elements in Synechocystis PCC6803 [J]. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(13): 7540-7545. doi: 10.1073/pnas.131201098
[72] Wadhams G H, Armitage J P. Making sense of it all: bacterial chemotaxis [J]. Nature Reviews Molecular Cell Biology, 2004, 5(12): 1024-1037. doi: 10.1038/nrm1524
[73] Reader R W, Tso W W, Springer M S, et al. Pleiotropic aspartate taxis and serine taxis mutants of Escherichia coli [J]. Microbiology, 1979, 111(2): 363-374.
[74] Yoshihara S, Katayama M, Geng X, et al. Cyanobacterial phytochrome-like PixJ1 holoprotein shows novel reversible photoconversion between blue-and green-absorbing forms [J]. Plant and Cell Physiology, 2004, 45(12): 1729-1737. doi: 10.1093/pcp/pch214
[75] Jakob A, Nakamura H, Kobayashi A, et al. The (PATAN)-CheY-like response regulator PixE interacts with the motor ATPase PilB1 to control negative phototaxis in the cyanobacterium Synechocystis sp. PCC 6803 [J]. Plant and Cell Physiology, 2020, 61(2): 296-307. doi: 10.1093/pcp/pcz194
[76] Masuda S, Ono T. Biochemical characterization of the major adenylyl cyclase, Cya1, in the cyanobacterium Synechocystis sp. PCC 6803 [J]. FEBS Letters, 2004, 577(1-2): 255-258. doi: 10.1016/j.febslet.2004.09.086
[77] Okajima K, Yoshihara S, Fukushima Y, et al. Biochemical and functional characterization of BLUF-type flavin-binding proteins of two species of cyanobacteria [J]. Journal of Biochemistry, 2005, 137(6): 741-750. doi: 10.1093/jb/mvi089
[78] Yuan H, Bauer C E. PixE promotes dark oligomerization of the BLUF photoreceptor PixD [J]. Proceedings of the National Academy of Sciences, 2008, 105(33): 11715-11719. doi: 10.1073/pnas.0802149105
[79] Tanaka K, Nakasone Y, Okajima K, et al. Time-resolved tracking of interprotein signal transduction: Synechocystis PixD–PixE complex as a sensor of light intensity [J]. Journal of the American Chemical Society, 2012, 134(20): 8336-8339. doi: 10.1021/ja301540r
[80] Ren S, Sato R, Hasegawa K, et al. A predicted structure for the PixD–PixE complex determined by homology modeling, docking simulations, and a mutagenesis study [J]. Biochemistry, 2013, 52(7): 1272-1279. doi: 10.1021/bi301004v
[81] Sugimoto Y, Nakamura H, Ren S, et al. Genetics of the blue light-dependent signal cascade that controls phototaxis in the cyanobacterium Synechocystis sp. PCC6803 [J]. Plant and Cell Physiology, 2017, 58(3): 458-465.
[82] Nakane D, Nishizaka T. Asymmetric distribution of type IV pili triggered by directional light in unicellular cyanobacteria [J]. Proceedings of the National Academy of Sciences, 2017, 114(25): 6593-6598. doi: 10.1073/pnas.1702395114
[83] Song J Y, Cho H S, Cho J I, et al. Near-UV cyanobacteriochrome signaling system elicits negative phototaxis in the cyanobacterium Synechocystis sp. PCC 6803 [J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(26): 10780-10785. doi: 10.1073/pnas.1104242108
[84] Narikawa R, Suzuki F, Yoshihara S, et al. Novel photosensory two-component system (PixA–NixB–NixC) involved in the regulation of positive and negative phototaxis of cyanobacterium Synechocystis sp. PCC 6803 [J]. Plant and Cell Physiology, 2011, 52(12): 2214-2224. doi: 10.1093/pcp/pcr155
[85] Ramakrishnan P, Tabor J J. Repurposing Synechocystis PCC6803 UirS–UirR as a UV-violet/green photoreversible transcriptional regulatory tool in E. coli [J]. ACS Synthetic Biology, 2016, 5(7): 733-740. doi: 10.1021/acssynbio.6b00068
[86] Wilde A, Fiedler B, Börner T. The cyanobacterial phytochrome Cph2 inhibits phototaxis towards blue light [J]. Molecular Microbiology, 2002, 44(4): 981-988. doi: 10.1046/j.1365-2958.2002.02923.x
[87] Moon Y J, Kim S Y, Jung K H, et al. Cyanobacterial phytochrome Cph2 is a negative regulator in phototaxis toward UV-A [J]. FEBS Letters, 2011, 585(2): 335-340. doi: 10.1016/j.febslet.2010.12.007
[88] Park C M, Kim J I, Yang S S, et al. A second photochromic bacteriophytochrome from Synechocystis sp. PCC 6803: spectral analysis and down-regulation by light [J]. Biochemistry, 2000, 39(35): 10840-10847. doi: 10.1021/bi992831r
[89] Wu S H, Lagarias J C. Defining the bilin lyase domain: lessons from the extended phytochrome superfamily [J]. Biochemistry, 2000, 39(44): 13487-13495. doi: 10.1021/bi001123z
[90] Anders K, von Stetten D, Mailliet J, et al. Spectroscopic and photochemical characterization of the red-light sensitive photosensory module of Cph2 from Synechocystis PCC 6803 [J]. Photochemistry and Photobiology, 2011, 87(1): 160-173. doi: 10.1111/j.1751-1097.2010.00845.x
[91] Savakis P, De Causmaecker S, Angerer V, et al. Light-induced alteration of c-di-GMP level controls motility of Synechocystis sp. PCC 6803 [J]. Molecular Microbiology, 2012, 85(2): 239-251. doi: 10.1111/j.1365-2958.2012.08106.x
[92] Paul R, Weiser S, Amiot N C, et al. Cell cycle-dependent dynamic localization of a bacterial response regulator with a novel di-guanylate cyclase output domain [J]. Genes & Development, 2004, 18(6): 715-727.
[93] Schmidt A J, Ryjenkov D A, Gomelsky M. The ubiquitous protein domain EAL is a cyclic diguanylate-specific phosphodiesterase: enzymatically active and inactive EAL domains [J]. Journal of Bacteriology, 2005, 187(14): 4774-4781. doi: 10.1128/JB.187.14.4774-4781.2005
[94] Wallner T, Pedroza L, Voigt K, et al. The cyanobacterial phytochrome 2 regulates the expression of motility-related genes through the second messenger cyclic di-GMP [J]. Photochemical & Photobiological Sciences, 2020, 19(5): 631-643.
[95] Ko M, Park C. Two novel flagellar components and H-NS are involved in the motor function of Escherichia coli [J]. Journal of Molecular Biology, 2000, 303(3): 371-382. doi: 10.1006/jmbi.2000.4147
[96] Boehm A, Kaiser M, Li H, et al. Second messenger-mediated adjustment of bacterial swimming velocity [J]. Cell, 2010, 141(1): 107-116. doi: 10.1016/j.cell.2010.01.018
[97] Wang Y C, Chin K H, Tu Z L, et al. Nucleotide binding by the widespread high-affinity cyclic di-GMP receptor MshEN domain [J]. Nature Communications, 2016(7): 12481.
[98] Angerer V, Schwenk P, Wallner T, et al. The protein Slr1143 is an active diguanylate cyclase in Synechocystis sp. PCC 6803 and interacts with the photoreceptor Cph2 [J]. Microbiology, 2017, 163(6): 920-930. doi: 10.1099/mic.0.000475
[99] Schuergers N, Nürnberg D J, Wallner T, et al. PilB localization correlates with the direction of twitching motility in the cyanobacterium Synechocystis sp. PCC 6803 [J]. Microbiology, 2015, 161(5): 960-966. doi: 10.1099/mic.0.000064
[100] Terauchi K, Ohmori M. An adenylate cyclase, Cyal, regulates cell motility in the cyanobacterium Synechocystis sp. PCC 6803 [J]. Plant and Cell Physiology, 1999, 40(2): 248-251. doi: 10.1093/oxfordjournals.pcp.a029534
[101] Yoshimura H, Hisabori T, Yanagisawa S, et al. Identification and characterization of a novel cAMP receptor protein in the cyanobacterium Synechocystis sp. PCC 6803 [J]. Journal of Biological Chemistry, 2000, 275(9): 6241-6245. doi: 10.1074/jbc.275.9.6241
[102] Yoshimura H, Yanagisawa S, Kanehisa M, et al. Screening for the target gene of cyanobacterial cAMP receptor protein SYCRP1 [J]. Molecular Microbiology, 2002, 43(4): 843-853. doi: 10.1046/j.1365-2958.2002.02790.x
[103] Yoshimura H, Kaneko Y, Ehira S, et al. CccS and CccP are involved in construction of cell surface components in the cyanobacterium Synechocystis sp. strain PCC 6803 [J]. Plant and Cell Physiology, 2010, 51(7): 1163-1172. doi: 10.1093/pcp/pcq081
[104] Chandra A, Joubert L M, Bhaya D. Modulation of type IV pili phenotypic plasticity through a novel Chaperone-Usher system in Synechocystis sp 6803[J]. BioRxiv, doi: 10.1101/130278.
[105] Song W Y, Zang S S, Li Z K, et al. Sycrp2 is essential for twitching motility in the cyanobacterium Synechocystis sp. strain PCC 6803 [J]. Journal of Bacteriology, 2018, 200(21): e00436-18.
[106] Yoshihara S, Geng X, Ikeuchi M. pilG gene cluster and split pilL genes involved in pilus biogenesis, motility and genetic transformation in the cyanobacterium Synechocystis sp. PCC 6803 [J]. Plant and Cell Physiology, 2002, 43(5): 513-521. doi: 10.1093/pcp/pcf061
[107] Bhaya D, Takahashi A, Shahi P, et al. Novel motility mutants of Synechocystis strain PCC 6803 generated by in vitro transposon mutagenesis [J]. Journal of Bacteriology, 2001, 183(20): 6140-6143. doi: 10.1128/JB.183.20.6140-6143.2001
[108] Kim Y H, Park Y M, Kim S J, et al. The role of Slr1443 in pilus biogenesis in Synechocystis sp. PCC 6803: involvement in post-translational modification of pilins [J]. Biochemical and Biophysical Research Communications, 2004, 315(1): 179-186. doi: 10.1016/j.bbrc.2004.01.036
[109] Chen Z, Xu X D. DnaJ-like protein gene sll1384 is involved in phototaxis in Synechocystis sp. PCC 6803 [J]. Chinese Science Bulletin, 2009, 54(23): 4381-4386.
[110] Kamei A, Yuasa T, Orikawa K, et al. A eukaryotic-type protein kinase, SpkA, is required for normal motility of the unicellular cyanobacterium Synechocystis sp. strain PCC 6803 [J]. Journal of Bacteriology, 2001, 183(5): 1505-1510. doi: 10.1128/JB.183.5.1505-1510.2001
[111] Panichkin V B, Arakawa-Kobayashi S, Kanaseki T, et al. Serine/threonine protein kinase SpkA in Synechocystis sp. strain PCC 6803 is a regulator of expression of three putative pilA operons, formation of thick pili, and cell motility [J]. Journal of Bacteriology, 2006, 188(21): 7696-7699. doi: 10.1128/JB.00838-06
[112] Lacey R F, Binder B M. Ethylene regulates the physiology of the cyanobacterium Synechocystis sp. PCC 6803 via an ethylene receptor [J]. Plant Physiology, 2016, 171(4): 2798-2809. doi: 10.1104/pp.16.00602
[113] Hoiczyk E. Gliding motility in cyanobacteria: observations and possible explanations [J]. Archives of Microbiology, 2000, 174(1-2): 11-17. doi: 10.1007/s002030000187
[114] Hoiczyk E, Baumeister W. Envelope structure of four gliding filamentous cyanobacteria [J]. Journal of Bacteriology, 1995, 177(9): 2387-2395. doi: 10.1128/JB.177.9.2387-2395.1995
[115] Nultsch W, Schuchart H, Höhl M. Investigations on the phototactic orientation of Anabaena variabilis [J]. Archives of Microbiology, 1979, 122(1): 85-91.
[116] Hoiczyk E, Baumeister W. The junctional pore complex, a prokaryotic secretion organelle, is the molecular motor underlying gliding motility in cyanobacteria [J]. Current Biology, 1998, 8(21): 1161-1168. doi: 10.1016/S0960-9822(07)00487-3
[117] Halfen L N, Castenholz R W. Gliding motility in the blue-green alga Oscillatoria princeps [J]. Journal of Phycology, 1971, 7(2): 133-145.
[118] Adams D G, Ashworth D, Nelmes B. Fibrillar array in the cell wall of a gliding filamentous cyanobacterium [J]. Journal of Bacteriology, 1999, 181(3): 884-892. doi: 10.1128/JB.181.3.884-892.1999
[119] Read N, Connell S, Adams D G. Nanoscale visualization of a fibrillar array in the cell wall of filamentous cyanobacteria and its implications for gliding motility [J]. Journal of Bacteriology, 2007, 189(20): 7361-7366. doi: 10.1128/JB.00706-07
[120] Hoiczyk E, Baumeister W. Oscillin, an extracellular, Ca2+-binding glycoprotein essential for the gliding motility of cyanobacteria [J]. Molecular Microbiology, 1997, 26(4): 699-708.
[121] Wolgemuth C, Hoiczyk E, Kaiser D, et al. How Myxobacteria glide [J]. Current Biology, 2002, 12(5): 369-377. doi: 10.1016/S0960-9822(02)00716-9
[122] Marsac N T. Differentiation of Hormogonia and Relationships with Other Biological Processes [M]//Bryant D A (Eds.), The Molecular Biology of Cyanobacteria. Boston: Kluwer Academic Publishers, 1994: 825-842.
[123] Meeks J C, Campbell E L, Summers M L, et al. Cellular differentiation in the cyanobacterium Nostoc punctiforme [J]. Archives of Microbiology, 2002, 178(6): 395-403. doi: 10.1007/s00203-002-0476-5
[124] Hernandez-Muniz W, Stevens S E. Characterization of the motile hormogonia of Mastigocladus laminosus [J]. Journal of Bacteriology, 1987, 169(1): 218-223. doi: 10.1128/JB.169.1.218-223.1987
[125] Duggan P S, Gottardello P, Adams D G. Molecular analysis of genes in Nostoc punctiforme involved in pilus biogenesis and plant infection [J]. Journal of Bacteriology, 2007, 189(12): 4547-4551. doi: 10.1128/JB.01927-06
[126] Robinson W B, Mealor A E, Stevens J S E, et al. Measuring the force production of the hormogonia of Mastigocladus laminosus [J]. Biophysical Journal, 2007, 93(2): 699-703. doi: 10.1529/biophysj.107.104067
[127] Damerval T, Guglielmi G, Houmard J, et al. Hormogonium differentiation in the cyanobacterium Calothrix: a photoregulated developmental process [J]. The Plant Cell, 1991, 3(2): 191-201. doi: 10.2307/3869288
[128] Khayatan B, Meeks J C, Risser D D. Evidence that a modified type IV pilus-like system powers gliding motility and polysaccharide secretion in filamentous cyanobacteria [J]. Molecular Microbiology, 2015, 98(6): 1021-1036. doi: 10.1111/mmi.13205
[129] Risser D D, Meeks J C. Comparative transcriptomics with a motility-deficient mutant leads to identification of a novel polysaccharide secretion system in Nostoc punctiforme [J]. Molecular Microbiology, 2013, 87(4): 884-893. doi: 10.1111/mmi.12138
[130] Risser D D, Chew W G, Meeks J C. Genetic characterization of the hmp locus, a chemotaxis-like gene cluster that regulates hormogonium development and motility in Nostoc punctiforme [J]. Molecular Microbiology, 2014, 92(2): 222-233. doi: 10.1111/mmi.12552
[131] Cho Y W, Gonzales A, Harwood T V, et al. Dynamic localization of HmpF regulates type IV pilus activity and directional motility in the filamentous cyanobacterium Nostoc punctiforme [J]. Molecular Microbiology, 2017, 106(2): 252-265. doi: 10.1111/mmi.13761
[132] Riley K W, Gonzalez A, Risser D D. A partner-switching regulatory system controls hormogonium development in the filamentous cyanobacterium Nostoc punctiforme [J]. Molecular Microbiology, 2018, 109(4): 555-569. doi: 10.1111/mmi.14061
[133] Khayatan B, Bains D K, Cheng M H, et al. A putative O-linked β-N-acetylglucosamine transferase is essential for hormogonium development and motility in the filamentous cyanobacterium Nostoc punctiforme [J]. Journal of Bacteriology, 2017, 199(9): e00075-17.
[134] Gonzalez A, Riley K W, Harwood T V, et al. A tripartite, hierarchical sigma factor cascade promotes hormogonium development in the filamentous cyanobacterium Nostoc punctiforme [J]. mSphere, 2019, 4(3): e00231-19.
[135] Malin G, Walsby A E. Chemotaxis of a cyanobacterium on concentration gradients of carbon dioxide, bicarbonate and oxygen [J]. Microbiology, 1985, 131(10): 2643-2652. doi: 10.1099/00221287-131-10-2643
[136] Richardson L L, Castenholz R W. Diel vertical movements of the cyanobacterium Oscillatoria terebriformis in a sulfide-rich hot spring microbial mat [J]. Applied and Environment Microbiology, 1987, 53(9): 2142-2150. doi: 10.1128/AEM.53.9.2142-2150.1987
[137] Richardson L L, Castenholz R W. Chemokinetic motility responses of the cyanobacterium Oscillatoria terebriformis [J]. Applied and Environment Microbiology, 1989, 55(1): 261-263. doi: 10.1128/AEM.55.1.261-263.1989
[138] Häder D P. Signal perception and amplification in photoresponses of cyanobacteria [J]. Biophysical Chemistry, 1988, 29(1-2): 155-159. doi: 10.1016/0301-4622(88)87035-2
[139] 孔任秋, 徐旭东, 冯勃. 集胞藻的随机插入诱变和光激活异养突变株的筛选 [J]. 水生生物学报, 2001, 25(6): 537-543. doi: 10.1007/BF00408050 Kong R Q, Xu X D, Feng B. Random insertion mutagenesis of Synechocystis sp.PCC6803 and selection of lahg mutants [J]. Acta Hydrobiologica Sinica, 2001, 25(6): 537-543. [ doi: 10.1007/BF00408050
[140] Garcia-Pichel F, Mechling M, Castenholz R W. Diel migrations of microorganisms within a benthic, hypersaline mat community [J]. Applied and Environment Microbiology, 1994, 60(5): 1500-1511. doi: 10.1128/AEM.60.5.1500-1511.1994
[141] Fourçans A, Solé A, Diestra E, et al. Vertical migration of phototrophic bacterial populations in a hypersaline microbial mat from Salins-de-Giraud (Camargue, France) [J]. FEMS Microbiology Ecology, 2006, 57(3): 367-377. doi: 10.1111/j.1574-6941.2006.00124.x
[142] Biddanda B A, McMillan A C, Long S A, et al. Seeking sunlight: rapid phototactic motility of filamentous mat-forming cyanobacteria optimize photosynthesis and enhance carbon burial in Lake Huron’s submerged sinkholes [J]. Frontiers in Microbiology, 2015(6): 930.
[143] Nilsson M, Rasmussen U, Bergman B. Cyanobacterial chemotaxis to extracts of host and nonhost plants [J]. FEMS Microbiology Ecology, 2006, 55(3): 382-390. doi: 10.1111/j.1574-6941.2005.00043.x
[144] Campbell E L, Hagen K D, Chen R, et al. Genetic analysis reveals the identity of the photoreceptor for phototaxis in hormogonium filaments of Nostoc punctiforme [J]. Journal of Bacteriology, 2015, 197(4): 782-791. doi: 10.1128/JB.02374-14
[145] Kirpich J S, Chang C W, Madsen D, et al. Noncanonical photodynamics of the orange/green cyanobacteriochrome power sensor NpF2164g7 from the PtxD phototaxis regulator of Nostoc punctiforme [J]. Biochemistry, 2018, 57(18): 2636-2648. doi: 10.1021/acs.biochem.7b01252
-
期刊类型引用(0)
其他类型引用(3)



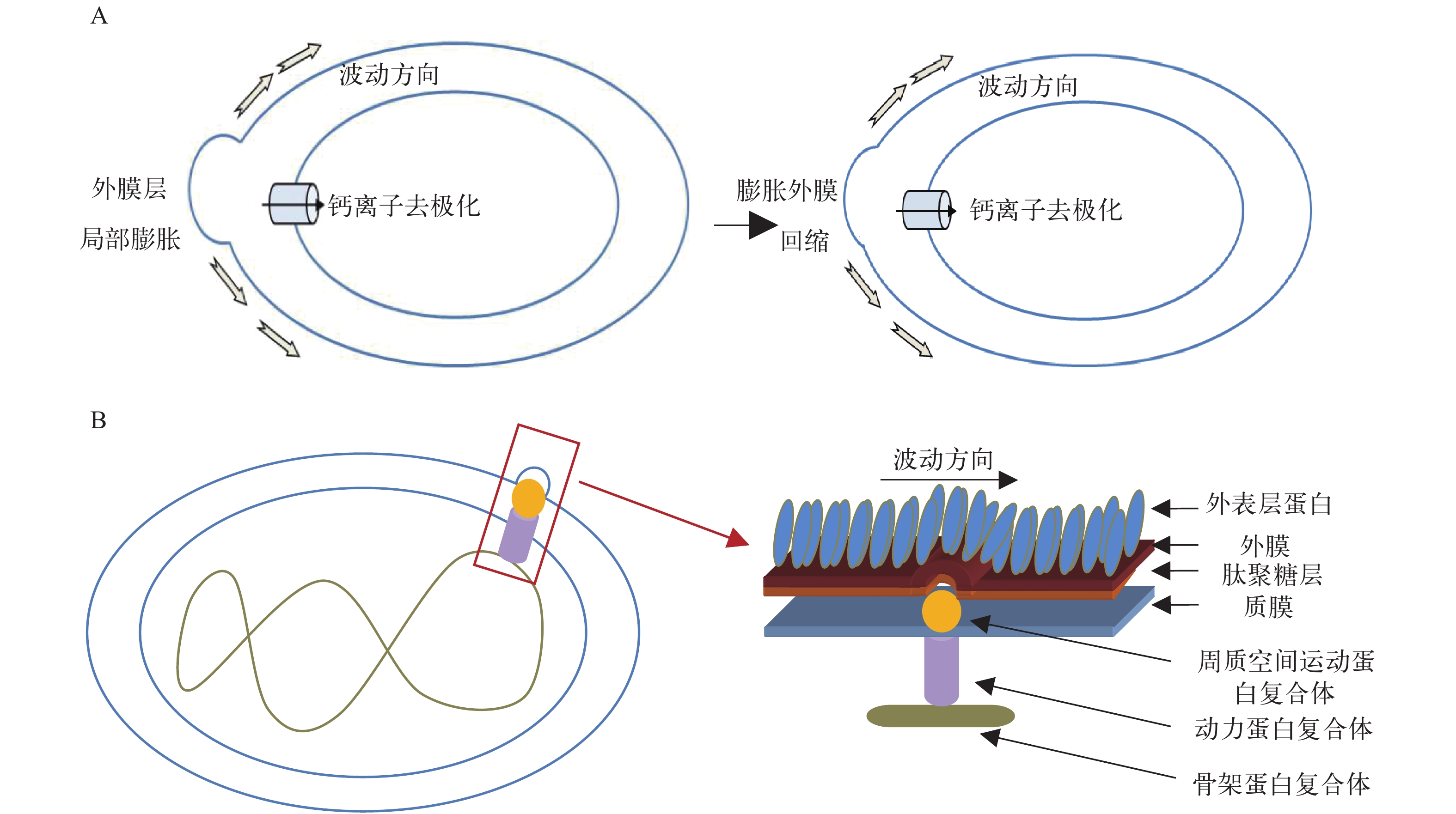
 下载:
下载: