GENETIC MONITORING OF CHANGFENG SILVER CARP BASE ON MICROSATELLITE
-
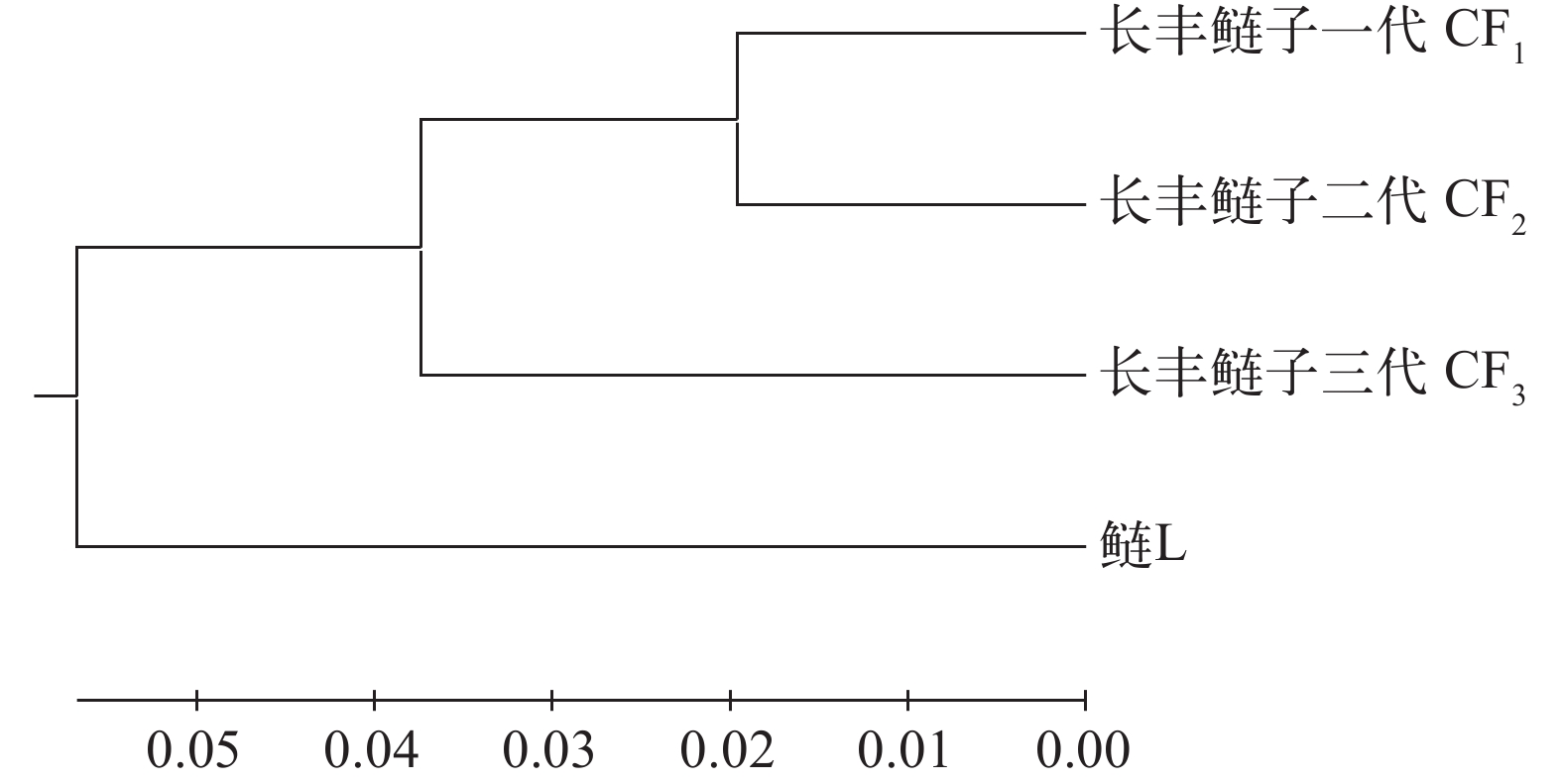
摘要: 长丰鲢(CF)为我国人工培育的鱼类新品种, 自推广应用以来取得了良好的效果。开展长丰鲢种质资源遗传监测, 对其优良性状保持具重要作用。研究采用18对微卫星引物分析了鲢(L)和长丰鲢世代间(CF1、CF2和CF3)的遗传多样性和遗传结构。结果表明: 鲢遗传多样性指数高于长丰鲢, 遗传多样性也较长丰鲢丰富。而长丰鲢子代间CF1到CF3平均等位基因数(Na)从5.7222下降到5.0556; 平均有效等位基因数(Ne)从3.2551下降到3.1461; 平均观测杂合度(Ho)从0.6975下降到0.5407; 平均期望杂合度(He)从0.6422下降到0.6235; 多态信息含量(PIC)从0.5784下降到0.5609。CF1到CF3的遗传参数是逐渐下降, 遗传多样性逐渐降低, 但群体间遗传多样性仍较高。长丰鲢子代间Fst在0.0160—0.0315, 表明其群体已出现了遗传分化, 但分化程度较低。长丰鲢各世代间遗传距离逐渐增加, 遗传相似度逐渐减小。研究表明经过连续3代利用, 长丰鲢CF1到CF3的遗传结构发生了改变, 遗传多样性呈下降趋势, 但遗传多样性水平仍较高。研究结果为长丰鲢进一步优良性状的维持提供了依据。Abstract: Changfeng silver carp (Hypophthalmichthys molitrix) (CF) is a new variety obtained by artificially selective breeding in China. Genetic monitoring the germplasm resources of Changfeng silver carp play an important role in maintaining its excellent traits. 18 microsatellite markers were used to analyze the genetic diversity and genetic structure in Changfeng silver carp (CF) populations. The results showed that the genetic diversity of silver carp (L) was higher than that of CF. The average number of alleles (Na) from CF1 to CF3 of CF offspring decreased from 5.7222 to 5.0556, and the average effective allele (Ne) were from 3.2551 to 3.1461. The average observed heterozygosity (Ho), the average expected heterozygosity (He), and the polymorphic information content (PIC) ranged from 0.6975 to 0.5407, 0.6422 to 0.6235, and 0.5784 to 0.5609, respectively. The Fst among the progeny of L and CF ranged from 0.0160 to 0.0315, indicating that the population of L has been genetically differentiated with low degree of differentiation. These results showed that the genetic structure of CF1 to CF3 exhibit slightly declining genetic diversity after three successive generations, whereas the genetic diversity was still abundant. Our study provides a basis for maintaining the genetic diversity of Changfeng silver carp.
-
Keywords:
- Genetic diversity /
- Genetic monitoring /
- Genetic structure /
- SSR /
- Silver carp /
- Changfeng silver carp
-
石斑鱼属(Epinephelus)隶属鲈形目(Perciformes), 鲈亚目(Percoidei), 鮨科(Serranidae)石斑鱼亚科(Epinephelinae), 是石斑鱼亚科中物种数量最多的一个属, 广泛分布于全球热带、亚热带海域, 是珊瑚礁的重要鱼类, 也是重要的海洋经济名贵食用鱼类。世界上石斑鱼属98种[1], 我国约40余种, 主要分布于东海及南海[2-6]。
形态上, 石斑鱼属鱼类近缘物种十分相似, 许多种类在外部形态特征如体色和斑纹都非常接近, 且许多种类体色与斑纹还会随着个体的发育发生巨大变化, 幼鱼与成鱼的形态截然不同[1, 3]。另外, 在不同的生活环境及生理应激状态下, 许多石斑鱼的体色花纹会出现明显变化, 给传统基于外部性状的系统分类带来巨大的困扰, 许多种类的分类与命名存在巨大争议[1-6]。如云纹石斑鱼(Epinephelus moara)与褐石斑鱼(Epinephelus bruneus), 斜带石斑鱼(Epinephelus coioides)与马拉巴石斑鱼(Epinephelus malabaricus), 均存在同种异名的分类争议。在分子水平研究上, Craig等[7]最先针对东太平洋及西大西洋分布石斑鱼类进行较为系统的分子分类学研究。而对于西太平洋分布的种类, 国内也有部分研究报道, 如丁少雄等[8]利用16S rRNA及庄轩等[9]利用Cyt b基因分析我国近海20余种石斑鱼属鱼类系统分类关系; 陈兴汉等[10]基于Cyt b基因分析南海10种石斑鱼属鱼类的分类关系等。但上述研究主要是围绕石斑鱼亚科的研究中涉及部分石斑鱼属的种类, 针对西太平洋石斑鱼属的详细系统演化关系研究还存在一定空白。
DNA条形码(DNA Barcoding)技术是通过一个标准目的基因的DNA序列分析而进行物种鉴定的技术, 它能在分子水平上成功区分物种, 为物种分类提供一种快速有效的辨别方法[11, 12]。在鱼类上, 该基因是线粒体细胞色素C 氧化酶亚基Ⅰ(COⅠ)基因5′端一段长度为648 bp 的片段, 在前期许多研究中已验证其可作为许多鱼类有效的DNA条形码基因。如Pereira等[13]利用COⅠ条形码序列对新热带区的254 种淡水鱼类进行分子鉴定, 其中252 种鱼类能被清晰区分, 并确定23个隐存种; 张楠等[14]基于DNA条形码技术对江门沿岸海域夏季鱼卵的鉴定, 获得鱼卵个体有效线粒体COⅠ序列信息217个, 成功鉴定鱼卵5目14科19属20种(未知种2种)。TMO-4C4是一个单拷贝的核基因, 被认为是与肌联蛋白titin相似, 与肌肉组装和静息张力有关, 最初由Streelman和Karl[15]分离, 目前国外广泛用于鱼类的系统发育研究[16-18], 解决了许多鱼类分类争议问题, 而国内利用TMO-4C4标记进行鱼类系统进化研究的相对较少[19]。本研究选择COⅠ和TMO-4C4基因, 线粒体DNA与核DNA标记相结合, 共同分析西太平洋35种石斑鱼分子系统分类关系, 同时探讨COⅠ基因在石斑鱼属物种分子鉴定的有效性, 为解决石斑鱼属分类争议问题及物种有效鉴定提供分子水平依据。
1. 材料与方法
1.1 样品采集
石斑鱼属鱼类样品于2018—2019年在中国广东、福建、海南和香港等沿海地区的水产市场及渔港码头进行野外采集, 另有部分种类采集于菲律宾、印度尼西亚和澳大利亚等西太平洋国家。最后共获得石斑鱼属鱼类样品35种共142个个体。采集标本根据《Grouper of the World》[1]、《中国鱼类分类检索》[2]和《台湾鱼类志》[3]等主要分类资料进行初步形态鉴定。所有样品取肌肉或鳍条于95%酒精中固定, 用于实验室总基因组DNA的提取。石斑鱼样品种类与来源见表 1。
表 1 实验材料的种类和采集地Table 1. Species and localities of experimental samples属Genera 种Species 采样地点Localities 数量Number 属Genera 种Species 采样地点Localities 数量Number 石斑鱼属
Epinephelus赤点石斑鱼
Epinephelus akaara广东广州、深圳;
福建漳州6 花点石斑鱼
Epinephelus maculatus广东广州、
深圳、湛江5 镶点石斑鱼
Epinephelus amblycephalus印度尼西亚、
广东深圳3 马拉巴石斑鱼
Epinephelus malabaricus广东汕头;
福建漳州4 宝石石斑鱼
Epinephelus areolatus广东广州、深圳 4 蜂巢石斑鱼
Epinephelus merra广东深圳、湛江 5 青石斑鱼
Epinephelus awoara广东阳江、
湛江、茂名6 云纹石斑鱼
Epinephelus moara广东阳江;
福建东山岛5 白背石斑鱼
Epinephelus bilobatus澳大利亚 2 吊桥石斑鱼
Epinephelus morrhua福建东山岛 2 布氏石斑鱼
Epinephelus bleekeri广东阳江 5 蓝棕石斑鱼
Epinephelus multinotatus菲律宾、中国香港 3 褐石斑鱼
Epinephelus bruneus广东深圳;
福建东山岛4 纹波石斑鱼
Epinephelus ongus斐济、广东汕头 4 网纹石斑鱼
Epinephelus chlorostigma福建东山岛 2 清水石斑鱼
Epinephelus polyphekadion广东深圳、阳江 4 萤点石斑鱼
Epinephelus coeruleopunctatus越南 2 玳瑁石斑鱼
Epinephelus quoyanus广东湛江、汕头;
福建漳州6 斜带石斑鱼
Epinephelus coioides广东深圳、
阳江、茂名6 霜点石斑鱼
Epinephelus rivulatus广东深圳 2 珊瑚石斑鱼
Epinephelus corallicola广东广州、深圳 5 吻斑石斑鱼
Epinephelus spilotoceps菲律宾 3 细点石斑鱼
Epinephelus cyanopodus越南、中国香港 4 南海石斑鱼
Epinephelus stictus越南、海南海口 5 双棘石斑鱼
Epinephelus diacanthus印度尼西亚、
菲律宾5 巨石斑鱼
Epinephelus tauvina广东深圳;
海南三亚3 小点石斑鱼
Epinephelus epistictus福建东山岛 3 三斑石斑鱼
Epinephelus trimaculatus广东广州、
湛江5 拟青石斑鱼
Epinephelus fasciatomaculosus广东阳江、
茂名5 蓝身大斑石斑鱼
Epinephelus tukula广东深圳、
福建东山岛3 黑边石斑鱼
Epinephelus fasciatus广东广州、深圳;
福建东山岛5 波纹石斑鱼
Epinephelus undulosus印度尼西亚、
中国香港3 褐点石斑鱼
Epinephelus fuscoguttatus广东广州、
汕头6 鳃棘鲈属
Plectropomus蓝点鳃棘鲈
Plectropomus areolatus广东广州 1 鞍带石斑鱼
Epinephelus lanceolatus广东深圳;
福建东山岛4 豹纹鳃棘鲈
Plectropomus leopardus广东深圳 1 纵带石斑鱼
Epinephelus latifasciatus福建东山岛 3 1.2 总基因组DNA的提取
取约50 mg酒精固定的样品组织, 利用动物组织DNA提取试剂盒 (天根生化科技有限公司), 按照使用说明书进行提取。最后提取的总基因组DNA溶解于100 μL灭菌蒸馏水, 电泳检测, –20℃保存备用。
1.3 PCR扩增和测序
本研究扩增的基因片段为线粒体COⅠ基因与核基因TMO-4C4。参考文献[20], 选择扩增COⅠ基因片段的通用引物为COⅠ-F1: 5′-TCAACYAATCAYAAAGATATYGGCAC-3′和COⅠ-R1: 5′-ACTTCYGGGTGRCCRAARAATCA-3′。扩增TMO-4C4基因片段的引物为TMO-4C4-F1: 5′-GAAAAGAGT GTTTGAAAATGA-3′和TMO-4C4-R1: 5′-CATCGTGCTCCTGGGTGACAAAGT-3′[15]。PCR反应体系总体积为50 μL, 其中包括PCR Mix反应混合液(天根生化科技有限公司)25 μL、灭菌蒸馏水21 μL、上下游引物(10 μmol/L)各1 μL、DNA样品2 μL。PCR反应条件为94℃预变性5min, 94℃变性30s, 55℃退火30s, 72℃延伸1min, 设置35个循环, 最后72℃再延伸5min。PCR产物用1%琼脂糖凝胶电泳检测, 纯化回收后送天一辉远生物技术有限公司双向测序。
1.4 数据分析方法
测序所得序列运用 BioEdit 软件查看序列结果并辅助测序峰图进行人工校正, 序列经NCBI的Blastn工具进行相似性检索, 验证序列的准确性。利用Clustal W[21]软件进行排序比对, 去掉两端冗余序列, 得到一致序列进行后续的分析计算。利用MEGA 7.0[22]软件计算序列的碱基组成、序列转换颠换值、保守位点、变异位点和遗传距离(基于Kimura-2-Parameter模型)等数据。系统进化树的构建采用最大似然法及贝叶斯法, 以石斑鱼亚科的鳃棘鲈属2种鱼类作为外类群。利用jModelTest 2.1.5进行最佳进化模型预测[23], 最大似然树在RAxML 8.0[24]软件中完成, 分支的置信度采用重复抽样分析方法, 重复抽样的次数为1000次。贝叶斯法分析在MrBayes 3.1.2[25]软件中完成, 随机选取起始树, 计算1000000代, 取样代数100, 系统进化树节点置信度由后验概率(Posterior probabilities)提供。
2. 结果
2.1 序列特征分析
扩增获得35种142个石斑鱼个体COⅠ序列同源片段为636 bp, 利用MEGA 7.0软件分析可知, 序列中 A、T、G和C 碱基平均含量分别为24.4%、29.9%、17.8%和27.9%, 其中 A+T 含量(54.3%)高于 G+C 含量(45.7%), 编码212个氨基酸, 无碱基插入与缺失。在 636 bp编码序列中, 密码子第1位4种碱基含量差异不大, 其中G-1 (30.0%)最高, T-1 (18%)最低; 密码子第2位, T-2 含量(42.0%)最高, 明显高于其他3种碱基, A-2(15.1%)最低; 密码子第3位中T-3、A-3和C-3三种碱基含量相差不大, 而G-3 含量最低(8.9%), 表现出明显的反 G 偏倚。在 G+C 含量中, 密码子第 1位点 G+C 含量(55.9%)高于第2和第3位(42.5%, 38.7%)。此外, 在长度为 636 bp 序列中, 除去外类群, 保守位点 403 个(63.4%), 变异位点 233(36.6%), 简约性信息位点 205 (32.2%)。
在TMO-4C4基因中, 同源序列为486 bp, 编码162个氨基酸。碱基A、T、G和C平均含量分别为28.2%、25.1%、27.1%和19.6%, A+T含量(53.3%)高于G + C(46.7%)。密码子第1与第2位4种碱基含量差异较大, 密码子第1位碱基含量最高为G-1 (43.0%), 最低是C-1(14.2%), 密码子第2位最高是A-2(37.6%), 最低是G-2(9.6%), 明显的反G偏倚。而密码子第3位4种碱基含量分布较为平均, 均在20%—30%。序列的保守位点443个(67.5%), 变异位点43个(32.5%), 简约性信息位点23个(18.6%)。与COⅠ基因相比, TMO-4C4基因序列保守, 变异位点数远低于COⅠ基因, 物种间序列差异性较小。
基于Kimura 2-Parameter 模型计算COⅠ与TMO-4C4基因序列转换与颠换之比值分别为4.104和2.772, 两基因转换明显大于颠换。另外, 基于DAMBE7[26]软件对COⅠ与TMO-4C4基因转换颠换突变饱和分析。根据分析所得的散点图, 随着遗传距离的增大, 转换与颠换均呈线性递增趋势, 转换增加的速率大于颠换增加的速率, 显示COⅠ与TMO-4C4基因均未突变饱和, 可用于后续进化树的分析。另外, 汇总出COⅠ与TMO-4C4基因序列核苷酸平均变异情况(表 2)。在COⅠ基因中, 相同碱基对平均555个, 转换碱基对63个, 颠换碱基对18个; TMO-4C4基因相同碱基对平均479个, 转换碱基对5个, 颠换碱基对2个。两基因转换与颠换均属第3密码子出现最多(表 2)。这可能与密码子第3位发生的碱基替换不易造成氨基酸突变, 而第 1和第 2 位点的替换容易引起氨基酸突变有关。
表 2 COⅠ与TMO-4C4基因片段碱基转换和颠换数Table 2. Numbers of transition and transversion of COⅠ and TMO-4C4 gene sequences基因
Gene密码子
位点
Codon sites碱基数
Numbers of base相同碱基对
Identical pairs (ii)转换碱基对
Transitional pairs (si)颠换碱基对
Transversio-nal pairs (sv)COⅠ 所有位点 636 555 63 18 1st位点 212 206 6 0 2nd位点 212 212 0 0 3rd位点 212 137 57 18 TMO-4C4 所有位点 486 479 5 2 1st位点 162 161 1 0 2nd位点 162 161 0 1 3rd位点 162 156 4 1 利用MEGA 7.0基于Kimura 2-parameter模型计算出35种石斑鱼COⅠ与TMO-4C4种内与种间遗传距离。在COⅠ基因中, 除去外类群, 在35种石斑鱼中, 种内遗传距离为0.000—0.008, 其中吊桥石斑鱼种内遗传距离最大, 为0.008, 其次是波纹石斑鱼与青石斑鱼, 均为0.006, 其他种类种内遗传距离均不大于0.005, 平均遗传距离为0.0027, 显著低于Hebert等[11]所推荐的物种鉴定最小种间遗传距离0.020 (2%)。35种石斑鱼属鱼类种间平均遗传距离为0.144, 是种内平均遗传距离的48倍, 其中种间遗传距离最大为玳瑁石斑鱼与网纹石斑鱼(0.202), 其次是蜂巢石斑鱼与霜点石斑鱼(0.200); 种间遗传距离最小为云纹石斑鱼与褐石斑鱼(0.027), 其次是斜带石斑鱼与马拉巴石斑鱼(0.036), 均大于Hebert等[11]设定的种间遗传距离2%的遗传差异。这表明COⅠ基因可以清晰区分石斑鱼属不同物种。
在TMO-4C4基因中, 遗传距离是0.000—0.030, 平均遗传距离为0.015, 可知核基因TMO-4C4比线粒体基因COⅠ保守很多, 遗传距离最大在南海石斑鱼与鞍带石斑鱼之间(0.032), 其次是网纹石班鱼与鞍带石斑鱼(0.029); 遗传距离最小值为0.000, 两物种间序列无显著差异, 如褐石斑鱼与云纹石斑鱼, 斜带石斑鱼与玛拉巴石斑鱼, 细点石斑鱼与蓝棕石斑鱼等。TMO-4C4序列十分保守, 在亲缘关系很近的物种间存在的遗传差异极小, 甚至无法区分物种。
2.2 分子系统进化关系分析
基于COⅠ、TMO-4C4和COⅠ+TMO-4C4联合序列, 以鳃棘鲈属中2种鳃棘鲈作为外类群进行进化树构建。根据jModelTest 2.1.5预测, 3种序列最佳核苷酸替代模型均为GTR + G + I, 系统进化树利用最大似然法及贝叶斯法2种方法进行构建。每种序列基于2种方法构建的进化树进行合并, 树上的节点分别为最大似然法支持率及贝叶斯法后验概率(图 1、图 2和图 3)。其中基于COⅠ+TMO-4C4联合序列构建的进化树各节点支持率及后验概率相对较高, 物种分类地位比较清晰, 这里主要针对COⅠ+TMO-4C4联合序列构建的系统进化树进行分析。在进化树上, 同种石斑鱼不同个体均能聚在一起, 形成该石斑鱼种内单系分支。鉴于个体样品数量多, 在进化树无法一一显示, 故进化树图片仅汇总35种石斑鱼种间聚类关系, 每种石斑鱼后标明样品个体数。35种石斑鱼大致分成两大类群, 类群Ⅰ由细点石斑鱼和蓝棕石斑鱼等22种石斑鱼聚成, 类群Ⅱ由吊桥石斑鱼和小点石斑鱼等13种石斑鱼组成。两大类群中亲缘关系较近的石斑鱼又聚成一些小分支, 部分物种在进化树上以高支持率聚成姐妹种, 如细点石斑鱼与蓝棕石斑鱼、白背石斑鱼与花点石斑鱼、吊桥石斑鱼与小点石斑鱼、萤点石斑鱼与珊瑚石斑鱼、云纹石斑鱼与褐石斑鱼、斜带石斑鱼与马拉巴石斑鱼等。
3. 讨论
3.1 COⅠ序列在石斑鱼属分类的可行性
前期研究表明, COⅠ基因在许多鱼类上已被验证可作为鉴定的标准DNA条形码序列, 可有效区分物种。目前在慈鲷科[27]、鲤科[28]、石首鱼科[29]、鲿科[30]和裂腹鱼亚科[31]等鱼类上均验证COⅠ基因可作为其物种识别标准序列。Hebert等[11]对11个门13320个物种的COⅠ基因分析得出物种内的COⅠ遗传距离基本小于2%。同时其提出, 利用 COⅠ序列有效鉴定物种的关键是种间遗传距离必须大于种内遗传距离10倍以上。本研究的35种石斑鱼共142个个体样品中, 种内平均遗传距离为0.003, 种间平均遗传距离为0.143, 种间平均遗传距离是种内平均遗传距离的47倍, 远大于Hebert 等[11, 12]提出的10倍差异值。另外, 石斑鱼属鱼类种内遗传距离为0.000—0.008, 均低于Hebert等[11]推荐的0.020(2%)作为物种鉴定最小遗传距离; 种间遗传距离为0.030—0.202, 均高于0.020(2%)的临界值, 辨析度清晰。可见COⅠ基因在石斑鱼属鱼类的分子系统分类上, 可作为有效的DNA条形码基因, 区分石斑鱼属内种间不同的物种。
3.2 基于DNA 条形码序列的石斑鱼属鱼类关系分析
石斑鱼属是石斑鱼亚科中种类最多的一个属, 我国分布40余种[2-6], 本研究采集35种, 基本囊括目前我国分类资料有记载的常见代表种类, 具有一定代表性。基于COⅠ及TMO-4C4基因构建的ML系统进化树, 35种石斑鱼形成两个平行分支。这与丁少雄等[8]利用16S rRNA和庄轩等[9]利用Cyt b构建的石斑鱼类分子系统进化结果一致。Schoelinck等[32]对分布印度-西太平洋石斑鱼亚科鱼类研究同样发现在石斑鱼属内存在2个分支。丁少雄等[8]在其西太平洋石斑鱼研究基础上结合东太平洋和大西洋石斑鱼16S rRNA数据联合分析发现, 石斑鱼属两大分支依然存在。其推测石斑鱼属共同祖先在演化初期先分化为两大支系, 两大支系种类再进一步种间分化并辐射分布于全球各海区。本研究基于COⅠ及TMO-4C4构建的进化树石斑鱼也形成两个分支, 聚类于两分支上的石斑鱼种类与前人研究基本一致[7, 8, 32], 结果也支持丁少雄等[8]关于石斑鱼系统演化的观点。
云纹石斑鱼(E. moara)与褐石斑鱼(E. bruneus)在前期分类研究中一直存在争议[1-3, 33-36]。Heemstra等[1]编著的《Groupers of the World》认为两者是同种异名。Fishbase的鱼类名录中也仅记录褐石斑鱼(E. bruneus)的信息, E. moara为其同种异名[33]。而成庆泰等[2]所著《中国鱼类系统检索》中根据侧线孔鳞数目差异、前鳃盖骨隅角有大棘还是锯齿认为褐石斑鱼和云纹石斑鱼是两个不同物种。郭明兰等[34]比较了云纹石斑鱼与褐石斑鱼体表形态及骨骼系统, 发现两者存在显著差异, 认为云纹石斑鱼是区别于褐石斑鱼的有效种。Liu等[35]基于外部形态特征及内部构造, 结合线粒体全基因组序列比较分析云纹石斑鱼与褐石斑鱼的差异, 支持云纹石斑鱼是有效的物种。本研究测定分析了形态初步鉴定为云纹石斑鱼与褐石斑鱼的COⅠ序列差异, 发现两者遗传距离为0.030, 大于 Hebert等[11]设定区分物种最小遗传距离2%。系统进化树上云纹石斑鱼与褐石斑鱼紧密聚为一支, 关系十分接近, 但两者种内不同个体均能聚成独立的物种单系分支, 没有交叉聚集在一起, 进化树的枝条长度也能有效把两种鱼区分, 因此本研究也支持前人研究的结果, 认为云纹石斑鱼和褐石斑鱼是石斑鱼属2个独立的物种, 不是同种异名[34, 35]。
斜带石斑鱼与马拉巴石斑鱼(也叫点带石斑鱼)在分类上也长期存在争议与混淆, 前期研究也存在把两种石斑鱼相互误鉴的情况[33, 36, 37]。《中国鱼类系统检索》[2]及《中国鱼类分类及分布名录》[6]仅有记载点带石斑鱼一种。而Heemstra等[1]根据幽门盲囊数的差异将斜带石斑鱼与马拉巴石斑鱼确定为2个种。沈世杰[2]主编的《台湾鱼类志》根据头部与体部是否有白斑, 胸鳍鳍条数差异将斜带石斑鱼与点带石斑鱼区分开。但由于两种鱼体表斑点与花纹过于相似, 目前许多资料对这两种石斑鱼依然存在误鉴, 在水产养殖生产中更是相互混淆, 存在混养与杂交的情况。本研究的斜带石斑鱼与马拉巴石斑鱼COⅠ基因遗传距离为0.036, 大于Hebert等[11]设定的2%临界值, 遗传分化达到种间水平。在进化树上, 两者紧密聚成姐妹分支, 但种内不同个体依然形成独立的物种单系分支, 显示是独立的物种。
在系统进化树上, 部分石斑鱼以较高的支持率聚成紧密的姐妹分支, 如细点石斑鱼与蓝棕石斑鱼、白背石斑鱼与花点石斑鱼、吻斑石斑鱼与巨石斑鱼、萤点石斑鱼与珊瑚石斑鱼等。比较这些石斑鱼相互间形态特征, 在体型、斑纹和体色上均非常接近, 形态分类上也容易发生误鉴[1, 33]。如细点石斑鱼与蓝棕石斑鱼身体均蓝色, 尾鳍截形, 主要区别是细点石斑鱼身体布满黑色细小斑点, 而蓝棕石斑鱼无此特征。在石斑鱼市场上两者均称为“蓝瓜子斑”, 容易混淆, 也存在用蓝棕石斑鱼冒充细点石斑鱼的现象。白背石斑鱼与花点石斑鱼斑纹异常相似, 两者体呈棕褐色, 身体密布有棱角褐色斑点, 浅色部分构成了网状花纹, 背鳍基部均有黑白相间斑纹, 细微区别为白背石斑鱼背鳍基部有一条贯通的白色带, 而花点石斑鱼没有。吻斑石斑鱼与巨石斑鱼体色淡黄色, 背部存在多块深色暗斑, 身体与各鳍均密布棕色小斑点, 巨石斑鱼斑点偏圆形, 吻斑石斑鱼斑点偏多边形。萤点石斑鱼与珊瑚石斑鱼体灰褐色, 头部尖, 体型修长, 尾鳍圆形, 萤点石斑鱼身体具白色不规则圆斑, 珊瑚石斑鱼具黑色斑点。在分子水平上, 部分前人研究构建的石斑鱼亚科分子系统进化树也显示这些石斑鱼存在较近的亲缘关系, 进化树上也聚集在一起[28, 39]。本研究中, 在 COⅠ遗传距离上, 细点石斑鱼与蓝棕石斑鱼(0.044)、白背石斑鱼与花点石斑鱼(0.056)、吻斑石斑鱼与巨石斑鱼(0.046)、萤点石斑鱼与珊瑚石斑鱼(0.063)相互间的遗传距离也较小, 远低于石斑鱼属种间平均遗传距离(0.144), 表明许多石斑鱼进化过程中, 外观表型进化与分子进化的趋势一致, 形态相似的种类其分子水平也接近。该结果可为揭示石斑鱼近缘物种分类关系, 澄清分类争议提供理论依据, 针对形态相似的类群, 结合分子水平信息, 可更科学有效明晰石斑鱼属各物种间的分类关系。
-
表 1 长丰鲢各龄体长及体质量组成
Table 1 Body length and body mass in each age group of Changfeng silver carp
年龄(龄)
Age性别
Sex数量(尾)Number
(Tail)体长
Body length (cm)体质量
Body weight (kg)范围Range 均值Mean value 范围Range 均值Mean value 2 ♀ 11 33.00—
37.0035.26±
1.090.65—
0.910.81±
0.072 ♂ 10 34.50—
37.0035.66±
0.900.75—
0.930.84±
0.063 ♀ 15 47.40—
56.0050.78±
2.962.20—
3.702.83±
0.473 ♂ 15 52.30—
55.5054.40±
1.302.10—
3.502.68±
0.484 ♀ 15 60.70—
69.0065.74±
3.204.50—
6.605.84±
0.624 ♂ 15 67.00—
69.5068.26±
0.895.40—
5.855.67±
0.12表 2 鲢卫星分子标记及其引物序列
Table 2 Microsatellite markers and their primers sequences in silver carp
位点
Locus重复序列
Repeat sequence引物序列
Primer sequence退火温度
Annealing temperature (℃)片段大小
Size range (bp)H121 (AC)13 AACCATTCATGCTCCCAAAC 50 150—200 AATTCAACTCTGCCCTCTGG H129 (TA)7(TG)14 TGGGGTGTCCTAACTTTTTCA 48 93—150 GGGGGTTAATTGTGCATTTG S65 (TG)10 TGAACTGGATCAGAAGACACTCA 50 100—200 GCAAACTGCAAAAATGATTCTG S78 (TGC)6 ATCTACGCGTCTGCCAGTATC 60 300—400 ACTTCACGTGATCTTTACGAACG S92 (CA)8 AACACAACGATCCAACAGAGAAT 50 100—200 GGGTCTATGGATTCTTCCTTGTC S162 (CAA)5 GCTCGACTTGTGCCTAATTATTG 50 100—200 AAAATGACAATGTTTGGTCTTGG BL5 (TG)27 CCTGTGCCTTTGAACTCTGA 52 300—500 CCCTCCACCATACTGACAAG BL52 (TG)12 CAGAATCCAGAGCCGTCAG 54 150—300 CACCGAACAGGGAACCAA BL55 (GT)14 AAGGAAAGTTGGCTGCTC 52 100—200 GGCTCTGAGGGAGATACCAC BL56 (GT)16 TTAGGTGAACCCAGCAGC 54 200—400 AAGAAGCATTAGTGCAGATGAGTAC BL58 (GT)9 TTCCTGCCTGTGCTCCAT 52 100—200 TTGCATTGATGCTGTCCC BL62 (TG)11 ATATTAACATCTGCCGAAGC 52 150—300 ACAACCAGCAGTCTGAAGC BL82 (GA)12 (TG)
4TT(TG)4GTTGCTGCTTTATCTTTGGA 51 150—300 AACCACTTCACATAGGCTTG BL101 (AC)10A7 CCATCAGACAGCCAAAGACAA 54 300—400 TGAAGGCAAGGTCAAGGTTTT BL106-2 (AC)14 TTTAATTCTTCTAGCTGGACACG 54 200—300 CACTCCTCTTCCCTCGTAAAT BL109 (TG)21 GTGTCCTGGATTCTAGCCG 54 200—300 CATGAGAGAAACACCTGAACA BL116 (CT)15 GCGGGATGAGTTTGAAGAA 53 150—300 TATGGACTGGACTGCTGGAT 表 3 18个位点在鲢和长丰鲢4个群体中的遗传多样性
Table 3 Genetic diversity of four populations of silver carp and Changfeng silver carp based on 18 polymorphic microsatellite loci
位点
Locus群体
Populaiton等位基因数
Na有效等位基因数
Ne观测杂合度
Ho期望杂合度
He多态信息含量
PIC遗传偏离指数
PHWH111 L 7 2.8557 0.5517 0.6612 1.2921 –0.1656 CF1 4 3.4335 0.6500 0.7269 1.3034 –0.1058 CF2 4 2.0962 0.5625 0.5312 0.9858 0.0589 CF3 4 2.3047 0.5000 0.5757 1.0696 –0.1315 H121 L 4 3.2727 0.3333 0.7062 1.2702 –0.5280*** CF1 3 1.7890 0.2381 0.4518 0.7027 –0.4730* CF2 2 1.7534 0.1250 0.4365 0.6211 –0.7136*** CF3 2 1.6000 0.2333 0.3814 0.5623 –0.3883* H129 L 6 3.1250 0.8667 0.6915 1.3541 0.2534*** CF1 5 2.5200 0.3810 0.6179 1.1754 –0.3834*** CF2 5 2.6912 0.5938 0.6384 1.2200 –0.0699*** CF3 5 2.5175 0.2333 0.6130 1.1880 –0.6194*** S65 L 9 5.0420 0.8333 0.8153 1.8926 0.0221 CF1 6 3.8095 1.0000 0.7564 1.4863 0.3221** CF2 7 4.4814 0.5938 0.7892 1.6346 –0.2476** CF3 6 3.7815 0.5333 0.7480 1.5442 –0.2870** S78 L 8 5.1576 1.0000 0.8198 1.7772 0.2198* CF1 7 3.6598 1.0000 0.7445 1.5330 0.3432* CF2 6 3.6835 0.9375 0.7401 1.4485 0.2667* CF3 6 3.3520 0.9667 0.7136 1.4170 0.3547* S92 L 5 2.4259 0.9667 0.5977 1.0332 0.6174*** CF1 4 2.9109 0.8571 0.6725 1.1475 0.2745 CF2 3 2.3897 0.9688 0.5908 0.9514 0.6398*** CF3 3 2.3226 1.0000 0.5791 0.9184 0.7268*** S162 L 2 1.3006 0.2667 0.2350 0.3927 0.1349 CF1 2 1.0488 0.0476 0.0476 0.1125 0.0000 CF2 2 1.1683 0.1562 0.1463 0.2742 0.0677 CF3 2 1.0339 0.0333 0.0333 0.0848 0.0000 BL5 L 13 8.5308 0.9000 0.8977 2.2915 0.0026 CF1 11 6.7328 0.9524 0.8722 2.1379 0.0920 CF2 11 6.8040 0.9062 0.8666 2.1148 0.0457 CF3 10 6.9231 0.5333 0.8701 2.0618 –0.3871 BL52 L 6 3.1915 0.6667 0.6983 1.3975 –0.0453 CF1 5 2.1832 0.7143 0.5552 1.0700 0.2866 CF2 4 1.9636 0.6875 0.4985 0.8220 0.3791* CF3 3 1.9481 0.6000 0.4949 0.7689 0.2124 BL55 L 11 6.2937 0.7667 0.8554 2.0284 –0.1037*** CF1 9 4.5231 0.6667 0.7979 1.7688 –0.1644 CF2 7 4.9113 0.5625 0.8090 1.7173 –0.3047* CF3 10 5.2023 0.5333 0.8215 1.9009 –0.3508*** BL56 L 7 4.9046 0.7333 0.8096 1.7087 –0.0942 CF1 6 2.8544 0.8571 0.6655 1.3127 0.2879 CF2 5 2.5957 0.4375 0.6245 1.1850 –0.2994* CF3 4 2.3529 0.3000 0.5847 1.0691 –0.4869*** BL58 L 8 4.0541 0.8000 0.7661 1.6651 0.0443 CF1 4 2.0512 0.4762 0.5250 0.9124 –0.093 CF2 3 2.1245 0.5000 0.5377 0.8100 –0.0701 CF3 3 2.1251 0.5667 0.5384 0.8139 0.0526 BL62 L 6 3.2967 0.7333 0.7085 1.4305 0.0350 CF1 6 3.2910 0.8571 0.7131 1.3570 0.2019 CF2 7 3.5556 0.7188 0.7302 1.5372 –0.0156 CF3 5 3.2907 0.6000 0.7079 1.3472 –0.1524 BL82 L 6 3.8544 0.8333 0.7531 1.5263 0.1065* CF1 4 2.5491 0.6667 0.6225 1.0789 0.0710* CF2 3 2.6089 0.6562 0.6265 1.0281 0.0474* CF3 4 3.0252 0.4667 0.6808 1.1533 –0.3145 BL101 L 7 2.6572 0.6897 0.6346 1.2132 0.0868 CF1 4 2.4033 0.7143 0.5981 0.9939 0.1943 CF2 3 2.7234 0.5312 0.6429 1.0434 –0.1737 CF3 4 3.1304 0.5000 0.6921 1.1975 –0.2776 BL106-2 L 8 6.0811 0.8000 0.8497 1.8993 –0.0585 CF1 8 4.8462 0.9524 0.8130 1.7261 0.1715 CF2 7 5.0819 0.7500 0.8160 1.7506 –0.0809 CF3 7 4.0000 0.7333 0.7627 1.5389 –0.0385 BL109 L 12 8.6538 0.6333 0.8994 2.2844 –0.2959*** CF1 12 5.9195 0.9524 0.8513 2.0372 0.1188 CF2 11 5.0319 0.7812 0.8140 1.9019 –0.0403*** CF3 10 5.2632 0.8000 0.8237 1.8420 –0.0288** BL116 L 5 2.9221 0.5667 0.6689 1.2323 –0.1528 CF1 3 2.0656 0.5714 0.5285 0.7819 0.0812 CF2 3 2.4294 0.5312 0.5977 0.9806 –0.1113 CF3 3 2.4557 0.6000 0.6028 0.9923 –0.0046 注: L. 鲢; CF1. 长丰鲢子一代; CF2. 长丰鲢子二代; CF3. 长丰鲢子三代; * 数值间差异显著(P<0.05); ** 数值间差异显著(P<0.01); *** 数值间差异显著(P<0.001); 下同Note: L. silver carp; CF1. the first generation of Changfeng silver carp; CF2. the second generation of Changfeng silver carp; CF3. the third generation of Changfeng silver carp. * indicate significant difference with P<0.05; ** indicate significant difference with P<0.01; *** indicate significant difference with P<0.001. The same applies below 表 4 Hardy-Weinberg平衡检测d值及P值
Table 4 The d and P value of each locus by Hardy-Weinberg test
位点
LocusL CF1 CF2 CF3 d P-value d P-value d P-value d P-value H111 –0.1656 0.4625 –0.1058 0.4663 0.0589 0.8750 –0.1315 0.1296 H121 –0.5280 0.0002 –0.4730 0.0221 –0.7136 0.0001 –0.3883 0.0478 H129 0.2534 0.0000 –0.3834 0.0003 –0.0699 0.0000 –0.6194 0.0000 S65 0.0221 0.2899 0.3221 0.0044 –0.2476 0.0019 –0.2870 0.0015 S78 0.2198 0.0240 0.3432 0.0194 0.2667 0.0277 0.3547 0.0157 S92 0.6174 0.0000 0.2745 0.0686 0.6398 0.0000 0.7268 0.0000 S162 0.1349 1.0000 0.0000 1.0000 0.0677 1.0000 0.0000 1.0000 BL5 0.0026 0.2866 0.0920 0.7415 0.0457 0.7032 –0.3871 0.0000 BL52 –0.0453 0.2493 0.2866 0.7198 0.3791 0.0432 0.2124 0.3929 BL55 –0.1037 0.0000 –0.1644 0.4377 –0.3047 0.0282 –0.3508 0.0000 BL56 –0.0942 0.3072 0.2879 0.1830 –0.2994 0.0184 –0.4869 0.0004 BL58 0.0443 0.8536 –0.0930 0.8803 –0.0701 0.5635 0.0526 0.4731 BL62 0.0350 0.9016 0.2019 0.1932 –0.0156 0.8856 –0.1524 0.1016 BL82 0.1065 0.0108 0.0710 0.0322 0.0474 0.0302 –0.3145 0.0660 BL101 0.0868 0.5506 0.1943 0.5647 –0.1737 0.1935 –0.2776 0.1149 BL106-2 –0.0585 0.2240 0.1715 0.4786 –0.0809 0.0798 –0.0385 0.2237 BL109 –0.2959 0.0000 0.1188 0.9990 –0.0403 0.0003 –0.0288 0.0023 BL116 –0.1528 0.4126 0.0812 0.8020 –0.1113 0.6691 –0.0046 0.5119 Mean 0.0044 0.3096 0.0681 0.4230 –0.0345 0.2844 –0.1178 0.1712 St.Dev 0.2396 0.3323 0.2349 0.3677 0.2899 0.3759 0.3252 0.2668 表 5 鲢、长丰鲢各世代间Fis和Fst值比较
Table 5 Comparison of Fis and Fst values among different generations of silver carp and Changfeng silver carp
位点
LocusL∶ CF1 L∶ CF2 L∶ CF3 CF1∶ CF2 CFL1∶ CFL3 CF2∶ CF3 Fis Fst Fis Fst Fis Fst Fis Fst Fis Fst Fis Fst H111 0.1155 0.0257 0.0499 0.0701 0.1350 0.0495 0.0156 0.0359 0.0979 0.0252 0.0244 0.0043 H121 0.4968 0.0534 0.5923 0.0547 0.4701 0.0778 0.5830 0.0007 0.4223 0.0033 0.5547 0.0048 H129 0.0277 0.0580 –0.1162 0.0237 0.1425 0.0727 0.2086 0.0104 0.4906 0.0369 0.3282 0.0392 S65 –0.1911 0.0178 0.0959 0.0283 0.1110 0.0056 –0.0524 0.0104 –0.0409 0.0200 0.2548 0.0362 S78 –0.3047 0.0264 –0.2625 0.0243 –0.3043 0.0209 –0.3314 0.0283 –0.3768 0.0294 –0.3314 0.0096 S92 –0.4658 0.0363 –0.6552 0.0068 –0.6995 0.0041 –0.4749 0.0298 –0.5149 0.0284 –0.7105 0.0012 S162 –0.1322 0.0518 –0.1273 0.0081 –0.1368 0.0566 –0.0700 0.0246 –0.0213 0.0056 –0.0722 0.0279 BL5 –0.0681 0.0336 –0.0406 0.0228 0.1755 0.0344 –0.0904 0.0091 0.1296 0.0527 0.1574 0.0474 BL52 –0.1240 0.0273 –0.1501 0.0477 –0.0795 0.0491 –0.3574 0.0270 –0.2777 0.0350 –0.3173 0.0009 BL55 0.1152 0.0408 0.1883 0.0499 0.2116 0.0324 0.2197 0.0100 0.2437 0.0623 0.3169 0.0647 BL56 –0.1001 0.0391 0.1701 0.0421 0.2464 0.0562 –0.0239 0.0031 0.0551 0.0039 0.3801 0.0017 BL58 –0.0082 0.0310 –0.0135 0.0408 –0.0654 0.0486 0.0629 0.0318 –0.0009 0.0490 –0.0075 0.0023 BL62 –0.1419 0.0209 –0.0259 0.0429 0.0427 0.0211 –0.1138 0.0115 –0.0466 0.0031 0.0679 0.0083 BL82 –0.1125 0.0200 –0.0975 0.0198 0.0780 0.0289 –0.0805 0.0013 0.1126 0.0382 0.1269 0.0284 BL101 –0.1626 0.0035 0.0283 0.0110 0.0878 0.0280 –0.0237 0.0099 0.0397 0.0229 0.2148 0.0083 BL106-2 –0.0756 0.0398 0.0542 0.0226 0.0329 0.0216 –0.0661 0.0142 –0.0920 0.0331 0.0450 0.0156 BL109 0.0757 0.0360 0.1608 0.0169 0.1541 0.0133 –0.0621 0.0223 –0.0678 0.0438 0.0186 0.0161 BL116 0.0303 0.0121 0.1190 0.0090 0.0671 0.0123 0.0014 0.0156 –0.0566 0.0228 0.0423 0.0007 Mean –0.0570 0.0319 –0.0017 0.0301 0.0372 0.0352 –0.0364 0.0164 0.0053 0.0286 0.0607 0.0176 表 6 鲢、长丰鲢各世代间Fst值(下三角)和Fis值(上三角)
Table 6 Comparing pairwise values of Fst (below) and Fis (above) among different generations of silver carp and Changfeng silver carp
Pop ID L CF1 CF2 CF3 L –0.0570 –0.0017 0.0372 CF1 0.0319 –0.0364 0.0053 CF2 0.0301 0.0164 0.0607 CF3 0.0352 0.0286 0.0176 **** 表 7 鲢、长丰鲢各世代18对微卫星位点的F-检验
Table 7 F-statistics for different generations of silver carp and Changfeng silver carp at 18 microsatellite loci
位点
Locus近交系数FisInbreeding coefficient Fis 4个群体FisFour
populations Fis4个群体FstFour
populations Fst3个群体FisThree
populations Fis3个群体FstThree
populations FstL CF1 CF2 CF3 H111 0.1510 0.0829 –0.0756 0.1168 0.0749 0.0525 0.0475 0.0300 H121 0.5200 0.4602 0.7091 0.3778 0.5208 0.0552 0.5212 0.0039 H129 –0.2745 0.3684 0.0552 0.6129 0.1749 0.0600 0.3414 0.0383 S65 –0.0395 –0.3559 0.2357 0.2749 0.0299 0.0295 0.0546 0.0296 S78 –0.2405 –0.3760 –0.2869 –0.3777 –0.3176 0.0345 –0.3464 0.0299 S92 –0.6446 –0.3057 –0.6658 –0.7561 –0.5834 0.0274 –0.5635 0.0270 S162 –0.1538 –0.0244 –0.0847 –0.0169 –0.1088 0.0466 –0.0622 0.0299 BL5 –0.0195 –0.1185 –0.0624 0.3766 0.0438 0.0495 0.0657 0.0485 BL52 0.0291 –0.3180 –0.4010 –0.2329 –0.2096 0.0477 –0.3175 0.0285 BL55 0.0885 0.1441 0.2937 0.3398 0.2156 0.0642 0.2604 0.0610 BL56 0.0789 –0.3194 0.2883 0.4783 0.1167 0.0389 0.1331 0.0039 BL58 –0.0619 0.0708 0.0554 –0.0703 –0.0079 0.0512 0.0180 0.0369 BL62 –0.0526 –0.2313 0.0000 0.1381 –0.0362 0.0270 –0.0307 0.0102 BL82 –0.1253 –0.0970 –0.0641 0.3029 0.0044 0.0342 0.0551 0.0307 BL101 –0.1058 –0.2233 0.1605 0.2653 0.0340 0.0211 0.0800 0.0182 BL106-2 0.0426 –0.2000 0.0663 0.0222 –0.0167 0.0365 –0.0379 0.0277 BL109 0.2839 –0.1460 0.0250 0.0123 0.0480 0.0369 –0.0374 0.0365 BL116 0.1385 –0.1077 0.0971 –0.0122 0.0363 0.0178 –0.0033 0.0170 Mean –0.0214 –0.0943 0.0192 0.1029 0.0011 0.0406 0.0099 0.0282 表 8 鲢、长丰鲢共4个群体的Nei氏遗传距离(下三角)及遗传相似性系数(上三角)
Table 8 Nei’s standard genetic distance (below) and genetic identity (above) between 4 populations of silver carp and Changfeng silver carp
Pop ID L CF1 CF2 CF3 L **** 0.8946 0.8945 0.8892 CF1 0.1114 **** 0.9616 0.9093 CF2 0.1115 0.0392 **** 0.9470 CF3 0.1174 0.0951 0.0544 **** -
[1] 唐启升, 水产养殖业可持续发展战略研究课题组. 水产养殖业可持续发展战略研究 [J]. 中国家禽, 2012, 34(11): 13-15. doi: 10.3969/j.issn.1004-6364.2012.11.005 Tang Q S, Research group on sustainable development strategy of aquaculture. Research on sustainable development strategy of aquaculture [J]. China Poultry, 2012, 34(11): 13-15. doi: 10.3969/j.issn.1004-6364.2012.11.005
[2] 农业农村部渔业渔政管理局, 全国水产技术推广总站, 中国水产学会编制. 中国渔业统计年鉴-2018 [M]. 北京: 中国农业出版社, 2018. Bureau of Fisheries of Ministry of Agriculture and Rural Affairs, National Fisheries Technology Extension Center, China Society of Fisheries. China Fishery Statistics Yearbook 2018 [M]. Beijing: Chinese Agriculture Press, 2018.
[3] 梁宏伟, 李忠, 罗相忠, 等. 长丰鲢与长江鲢形态差异与判别分析 [J]. 水生生物学报, 2015, 39(5): 1059-1064. doi: 10.7541/2015.139 Liang H W, Li Z, Luo X Z, et al. Morphological differences and discriminant analysis between Changfeng and Yangtze River silver carp [J]. Acta Hydrobiologica Sinica, 2015, 39(5): 1059-1064. doi: 10.7541/2015.139
[4] 唐首杰, 毕详, 王成辉, 等. 团头鲂3个选育群体遗传纯度的微卫星分析 [J]. 水产科技情报, 2016, 43(5): 225-230. Tang S J, Bi X, Wang C H, et al. Microsatellite analysis of genetic purity in three selected populations of Megalobrama amblycephala [J]. Fisheries Science & Technology Information, 2016, 43(5): 225-230.
[5] 李耀国, 孙彤, 张雅剑, 等. 三亚卵形鲳鲹微卫星遗传多态性及性状关联分析 [J]. 海南热带海洋学院学报, 2018, 25(2): 1-5,12. Li Y G, Sun T, Zhang Y J, et al. Genetic diversity and trait association analysis of Trachinotus ovatus from Sanya by microsatellite markers [J]. Journal of Hainan Tropical Ocean University, 2018, 25(2): 1-5,12.
[6] 王 丰, 张家华, 沈玉帮, 等. 青鱼野生与养殖群体遗传变异的微卫星分析 [J]. 水生生物学报, 2019, 43(5): 939-944. doi: 10.7541/2019.111 Wang F, Zhang J H, Shen Y B, et al. Microsatellite analysis of genetic variation of wild and cultural populations in black carp Mylopharyngodon piceus [J]. Acta Hydrobiologica Sinica, 2019, 43(5): 939-944. doi: 10.7541/2019.111
[7] 李镕, 白俊杰, 李胜杰, 等. 大口黑鲈选育群体遗传结构的微卫星分析 [J]. 广东海洋大学学报, 2010, 30(3): 11-15. doi: 10.3969/j.issn.1673-9159.2010.03.003 Li R, Bai J J, Li S J, et al. Analysis on genetic structure of selected population of largemouth bass by microsatellite DNA markers [J]. Journal of Guangdong Ocean University, 2010, 30(3): 11-15. doi: 10.3969/j.issn.1673-9159.2010.03.003
[8] 阙延福, 胥贤, 徐念, 等. 基于微卫星标记的唇䱻亲子鉴定技术研究 [J]. 大连海洋大学学报, 2019, 34(5): 643-648. Que Y F, Xu X, Xu N, et al. Parentage analysis of Hemibarbus labeo based on microsatellite markers [J]. Journal of Dalian Ocean University, 2019, 34(5): 643-648.
[9] 武世雄, 姜欣彤, 王伟, 等. 大泷六线鱼6个群体遗传多样性的微卫星分析 [J]. 中国渔业质量与标准, 2018, 8(3): 52-60. doi: 10.3969/j.issn.2095-1833.2018.03.007 Wu S X, Jiang X T, Wang W, et al. Genetic diversity among 6 populations of Hexagrammos otakii based on microsatellite analysis [J]. Chinese Fishery Quality and Standards, 2018, 8(3): 52-60. doi: 10.3969/j.issn.2095-1833.2018.03.007
[10] 唐首杰, 毕详, 王成辉, 等. 团头鲂3个选育群体遗传潜力的微卫星分析 [J]. 南方水产科学, 2017, 13(2): 59-68. doi: 10.3969/j.issn.2095-0780.2017.02.008 Tang S J, Bi X, Wang C H, et al. Genetic potential analysis of three selective breeding populations of blunt snout bream (Megalobrama amblycephala) using microsatellite markers [J]. South China Fisheries Science, 2017, 13(2): 59-68. doi: 10.3969/j.issn.2095-0780.2017.02.008
[11] 史君洁, 石连玉, 李池陶, 等. 微卫星标记对4种鲤鱼种质鉴定技术的研究 [J]. 基因组学与应用生物学, 2018, 37(2): 748-755. Shi J J, Shi L Y, Li C T, et al. Research on germplasm identification technology for four species of common carp by microsatellite markers [J]. Genomics and Applied Biology, 2018, 37(2): 748-755.
[12] 王广宁, 马爱军, 李猛, 等. 大菱鲆微卫星标记在亲本后代中的分离分析 [J]. 海洋科学, 2016, 40(4): 1-10. Wang G N, Ma A J, Li M, et al. Separation and analysis of turbot microsatellite markers in progeny [J]. Marine Science, 2016, 40(4): 1-10.
[13] 曾珍. 松江鲈不同种群间的分子标记和遗传多样性分析 [D]. 上海: 上海海洋大学, 2013. Zeng Z. Molecular markers and genetic diversity analysis in populations of Trachidermus fasciatus [D]. Shanghai: Shanghai Ocean University, 2013.
[14] 王成龙, 郑国栋, 陈 杰, 等. ENU诱变草鱼及其雌核发育后代的微卫星遗传分析 [J]. 中国水产科学, 2017, 24(5): 1013-1019. doi: 10.3724/SP.J.1118.2017.16323 Wang C L, Zheng G D, Chen J, et al. Microsatellite genetic analysis of ENU mutagenesis grass carp and gynogenesis offspring group [J]. Journal of Fishery Sciences of China, 2017, 24(5): 1013-1019. doi: 10.3724/SP.J.1118.2017.16323
[15] 张新辉, 高泽霞, 罗 伟, 等. 雌核发育团头鲂的形态和遗传特征分析 [J]. 水生生物学报, 2015, 39(1): 126-132. doi: 10.7541/2015.16 Zhang X H, Gao Z X, Luo W, et al. Studies on morphological characteristics and genetic analysis of the gynogenesis blunt snout bream (Megalobrama amblycephala) [J]. Acta Hydrobiologica Sinica, 2015, 39(1): 126-132. doi: 10.7541/2015.16
[16] 叶香尘, 韦玲静, 梁克, 等. 广西本地鲢和长丰鲢群体遗传多样性分析 [J]. 基因组学与应用生物学, 2019, 38(1): 100-108. Ye X C, Wei L J, Liang K, et al. Genetic diversity analysis in Changfeng silver carp and Guangxi local silver carp [J]. Genomics and Applied Biology, 2019, 38(1): 100-108.
[17] Noble L. Microsatellites-evolution and applications [J]. Heredity, 1999, 83(5): 633-634.
[18] 全迎春, 孙效文, 梁利群. 应用微卫星多态分析四个鲤鱼群体的遗传多样性 [J]. 动物学研究, 2005, 26(6): 595-602. doi: 10.3321/j.issn:0254-5853.2005.06.004 Quan Y C, Sun X W, Liang L Q. Microsatellite variation among four breeding populations of common carps [J]. Zoological Research, 2005, 26(6): 595-602. doi: 10.3321/j.issn:0254-5853.2005.06.004
[19] 牛东红, 陈 慧, 林国文, 等. 缢蛏群体微卫星分析中样本量对遗传多样性指标的影响 [J]. 海洋科学进展, 2010, 28(2): 203-208. doi: 10.3969/j.issn.1671-6647.2010.02.010 Niu D H, Chen H, Lin G W, et al. Effects of sample size on genetic diversity index for population of Sinonovacula constricta in use of microsatellite DNA marker [J]. Advances in Marine Science, 2010, 28(2): 203-208. doi: 10.3969/j.issn.1671-6647.2010.02.010
[20] 陈虹均. 长江三峡库区渔业资源现状调查及鲢的遗传多样性分析 [D]. 重庆: 西南大学, 2017. Chen H J. The fisheries resources survey of the Three Gorges Reservoir in Yangtze River and genetic diversity analysis of silver carp (Hypophthalmichthys molitrix) [D]. Chongqing: Southwest University, 2017.
[21] 张天时, 王清印, 刘 萍, 等. 中国对虾(Fenneropenaeus chinensis)人工选育群体不同世代的微卫星分析 [J]. 海洋与湖沼, 2005, 36(1): 72-80. doi: 10.3321/j.issn:0029-814X.2005.01.011 Zhang T S, Wang Q Y, Liu P, et al. Genetic diversity analysis on selected populations of shrimp Fenneropenaeus chinensis by microsatellites [J]. Oceanologia et Limnologia Sinica, 2005, 36(1): 72-80. doi: 10.3321/j.issn:0029-814X.2005.01.011
[22] Botstein D, White R L, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms [J]. Journal of Pediatric Surgery, 1980, 32(3): 314-331.
[23] 苏钰玲, 李 敏, 杨永春, 等. 南方拟䱗微卫星标记筛选及遗传多样性分析 [J]. 水产科学, 2020, 39(2): 224-233. Su Y L, Li M, Yang Y C, et al. Microsatellite screening and genetic diversity analysis of gudgeon Pseudohemiculter dispar in Pearl River Basin [J]. Fisheries Science, 2020, 39(2): 224-233.
[24] 崔 蕾, 谢从新, 李艳和, 等. 斑点叉尾鮰4个群体遗传多样性的微卫星分析 [J]. 华中农业大学学报, 2012, 31(6): 744-751. doi: 10.3969/j.issn.1000-2421.2012.06.015 Cui L, Xie C X, Li Y H, et al. Analysis of genetic diversity among four different channel catfish populations by using microsatellite markers [J]. Journal of Huazhong Agricultural University, 2012, 31(6): 744-751. doi: 10.3969/j.issn.1000-2421.2012.06.015
[25] 徐湛宁, 李福贵, 郑国栋, 等. 团头鲂耐低氧新品系雌核发育群体遗传结构的微卫星分析 [J]. 水产学报, 2017, 41(3): 330-338. Xu Z N, Li F G, Zheng G D, et al. Analysis of genetic structure of gynogenetic population in new strain of hypoxia-tolerant Megalobrama amblycephala using microsatellite markers [J]. Journal of Fisheries of China, 2017, 41(3): 330-338.
[26] 李超, 鲁翠云, 曹顶臣, 等. 微卫星标记对德国镜鲤选育群体的遗传分析 [J]. 天津农学院学报, 2012, 19(4): 5-8. doi: 10.3969/j.issn.1008-5394.2012.04.003 Li C, Lu C Y, Cao D C, et al. Genetic analysis on selective population of German mirror carp (Cyprinus carpio L.) using microsatellite markers [J]. Journal of Tianjin Agricultural University, 2012, 19(4): 5-8. doi: 10.3969/j.issn.1008-5394.2012.04.003
[27] 樊佳佳, 白俊杰, 李胜杰, 等. 驯食配合饲料的大口黑鲈3个选育世代的遗传多样性分析 [J]. 渔业科学进展, 2019, 40(4): 57-64. Fan JJ, Bai JJ, Li SJ, et al. Analysis on genetic diversity of three breeding populations of largemouth bass using formulated feeds [J]. Progress in Fishery Sciences, 2019, 40(4): 57-64.
[28] McConnell S, Hamilton L, Morris D, et al. Isolation of salmonid microsatellite loci and their application to the population genetics of Canadian east coast stocks of Atlantic salmon [J]. Aquaculture, 1995, 137(1/2/3/4): 19-30.
[29] 马冬梅, 苏换换, 朱华平, 等. 华南鲤选育群体不同世代遗传多样性与遗传结构的微卫星分析 [J]. 水生生物学报, 2018, 42(5): 887-895. doi: 10.7541/2018.109 Ma D M, Su H H, Zhu H P, et al. Genetic diversity and genetic structure analysis of different selective breeding generations in Cyprinus carpio rubrofuscus using microsatellite markers [J]. Acta Hydrobiologica Sinica, 2018, 42(5): 887-895. doi: 10.7541/2018.109
-
期刊类型引用(6)
1. 吴晓东,陈菁菁,徐佳晟,李海燕,梁镇邦,初庆柱,吴仁协. 南海北部近海5种石斑鱼的骨骼系统比较研究. 广西科学院学报. 2024(01): 93-102 .  百度学术
百度学术
2. 何浩斌,张国庆,郜梓涵,胡凯红,林佳欣,周萌,黄燕华,梁日深. 基于ND1基因石斑鱼属鱼类分子系统进化关系研究. 水产科学. 2024(05): 746-756 .  百度学术
百度学术
3. 王乙婷,朱文斌,张亚洲,王业辉,贾程豪,陈治,高天翔. 浙江沿岸日本鳀幼鱼渔业兼捕鱼类的DNA条形码鉴定. 水产学报. 2024(10): 96-108 .  百度学术
百度学术
4. 黄琼叶,张建明,朱友芳,许秋龙. 丁香酚对细点石斑鱼麻醉效果及肝脏生理生化指标的影响. 河北渔业. 2024(10): 24-29 .  百度学术
百度学术
5. 陈治,蔡杏伟,张清凤,李高俊,马春来,申志新. 海南岛淡水鱼类环境DNA宏条形码参考数据库的初步构建及比较分析. 南方水产科学. 2022(03): 1-12 .  百度学术
百度学术
6. 孙军,宋煜尧,施义锋,翟键,燕文卓. 近十年中国海洋生物多样性研究进展. 生物多样性. 2022(10): 181-195 .  百度学术
百度学术
其他类型引用(1)



 下载:
下载: