GENETIC DIVERSITY AND POPULATION HISTORICAL DYNAMICS OF OCHETOBIUS ELONGATUS IN THE MIDDLE REACHES OF THE YANGTZE RIVER
-
摘要:
为研究长江中游鳤群体的遗传多样性, 利用两个线粒体基因和两个核基因序列作为分子标记, 分析了汉江下游、南洞庭湖、藕池河和长江公安段4个鳤群体的遗传多样性、遗传分化和种群历史动态。研究结果显示, 长江中游鳤单倍型多样性较高, 核苷酸多样性较低, 整体遗传多样性处于中等水平。分子方差变异分析(AMOVA)和遗传分化指数FST显示长江中游鳤群体间不存在遗传分化现象。种群历史动态分析表明长江中游鳤群体在历史上可能发生过种群扩张现象, 扩张时间约在16.54万年前, 处于庐山冰期和大理冰期间较为温暖的间冰期。STRUCTURE分析表明长江中游和西江中下游鳤群体间存在较明显的分化, 推算其分化时间约在5.55万年前, 建议在制定保护策略时将这两个水系的鳤群体视作两个保护单元。
Abstract:After the implementation of 10-year ban fishing in the Yangtze River, the rare and endangered fish species Ochetobius elongatus has reappeared in the middle reaches of the Yangtze River. However, the genetic diversity and population structure of O. elongatus in this region remain unclear, hampering the development of effective conservation strategies. In this study, two mitochondrial genes and two nuclear gene sequences were used as molecular markers to analyze the genetic diversity, genetic differentiation, and population historical dynamics of four O. elongatus populations: the Hanjiang River, South Dongting Lake, Ouchi River, and the Gongan section of the Yangtze River. The results showed that haplotype diversity of O. elongatus in the middle reaches of the Yangtze River was high, the nucleotide diversity was low, and the overall genetic diversity was at a medium level. Molecular variance analysis (AMOVA) and genetic differentiation index (FST) showed that there was no significant genetic differentiation among O. elongatus populations in the middle reaches of the Yangtze River. The analysis of population historical dynamics indicated a possible population expansion of O. elongatus in the middle reaches of the Yangtze River approximately 165400 years ago, during the warm interglacial period between Lushan Glacial and Dali glacial. Furthermore, clear differentiation was observed between O. elongatus populations in the middle reaches of the Yangtze River and those in the middle and lower reaches of the Xijiang River, with an estimated divergence time was about 55500 years ago. Based on these findings, we propose considering O. elongatus populations in these two river basins as distinct conservation units when formulating conservation strategies.
-
遗传多样性是生物多样性的基础, 一个物种遗传多样性的高低决定了其适应环境变化的能力和进化的潜力, 当一个物种的遗传多样性较高时, 其对环境变化的适应能力和抵抗疾病的能力就较强; 然而当一个物种的遗传多样较低时, 就会削弱其抵抗疾病的能力, 最终导致物种灭绝的概率增加[1]。开展濒危物种的遗传多样性、遗传分化、种群历史动态等研究, 从遗传角度解析其濒危机制, 可为濒危物种保护对策的制定提供理论依据[2]。
鳤(Ochetobius elongatus), 隶属于鲤形目, 鲤科, 鳤属, 曾是长江、珠江等南方水系的重要经济鱼类[3, 4]。近几十年来, 受过度捕捞、江湖阻隔、栖息地破坏等因素的影响, 其资源量急剧萎缩, 在很多河流已多年未被监测到[5]。2016年修订的《中国脊椎动物红色名录》已将鳤的保护等级上升为极度濒危[6], 被湖北、湖南等地列为省级重点保护水生野生动物。
由于难以获得样本, 目前关于鳤遗传多样性方面的研究较少。范凤娟等[7]分析了长江水系汨罗江4尾鳤与珠江水系柳江2尾鳤间Cyt b基因序列的差异; 杨计平等[8]研究了西江中下游鳤(桂平1尾、梧州1尾、肇庆50尾)的遗传多样性和种群历史动态; 陈蔚涛等[9]利用多基因标记揭示了长江公安江段7尾鳤与杨计平论文中52尾珠江鳤间存在高度遗传分化。这3篇论文使我们对鳤的遗传状况有了初步了解, 但由于样本量太少, 或局限于单个种群, 很难准确掌握鳤的遗传现状。
令人欣慰的是, 在长江“十年禁渔”政策实施后, 长江流域的水生生物资源得以休养生息, 鳤也重现长江中游。本课题组及合作单位于2022年在汉江下游、长江支流藕池河、洞庭湖等水域多次监测到鳤, 表明长江中游鳤资源正在逐步恢复。然而, 目前长江中游鳤群体的遗传多样性和遗传结构尚不明确, 不利于长江中游鳤保护策略的制定。
本研究利用2个线粒体基因细胞色素b (Cyt b)和酰胺腺嘌呤二核苷酸(NADH)脱氢酶亚单位2 (ND2), 2个核基因肌球蛋白重链6 (myh6)和重组激活基因2 (RAG2)序列作为分子标记, 分析了长江中游鳤群体的遗传多样性、遗传分化和种群历史动态, 同时分析了长江中游和西江中下游鳤群体间的遗传差异, 研究结果可为鳤的资源保护提供科学依据。
1. 材料与方法
1.1 样本采集
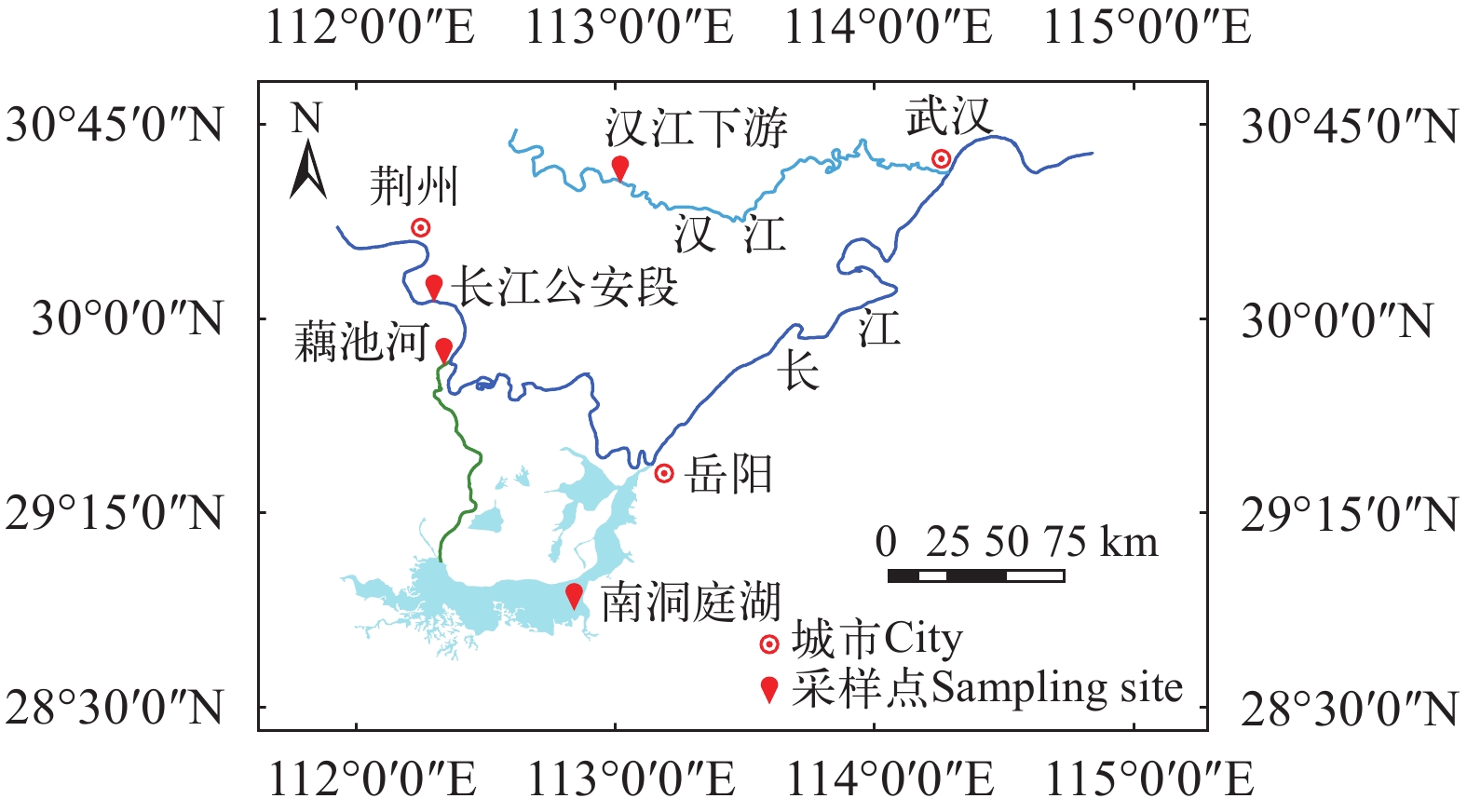
鳤样本采集于长江中游流域3个位点, 分别为汉江下游潜江段(14尾, 2022年7月至8月和2022年10月至11月)、藕池河河口段(7尾, 2023年4月至6月)和南洞庭湖(11尾, 2023年5月至6月), 另外从GenBank数据库中下载了长江干流公安段7尾鳤的基因序列, 合计有4个样点的39尾样本用于分析(图 1)。调查网具为定置刺网, 网目规格为2、6、10和14 cm, 每张刺网长50 m, 高2 m。下网时间为傍晚18:00, 收网时间为次日早上06:00。参考《中国动物志 硬骨鱼纲鲤形目(中卷)》[3]进行物种鉴定, 若采集到鳤, 存活个体剪取少量臀鳍条后放生, 鳍条样本使用95%酒精固定, 编号留存; 死亡个体在剪取鳍条后, 用甲醛将残体固定, 然后带回江汉大学生命科学学院制成标本用以科研和科普。
1.2 基因组DNA的提取、基因片段的扩增与测序
利用试剂盒法(成都福际生物技术有限公司)提取基因组DNA, 并使用1%的琼脂糖凝胶电泳检验总DNA的质量。扩增细胞色素b (Cyt b)、酰胺腺嘌呤二核苷酸(NADH)脱氢酶亚单位2 (ND2)、肌球蛋白重链6 (myh6)和重组激活基因2 (RAG2)基因片段的引物参考杨计平等[8]的研究(表 1)。聚合酶链式反应(Polymerase Chain Reaction, PCR)的反应体系总体积为30 μL, 包含15 μL 2×PrimeSTAR Max Premix (包含dNTPs、MgCl2、DNA 聚合酶、反应缓冲液, 宝日医生物技术有限公司)、2 μL模板DNA、各2 μL的正反引物(10 mmol/L)和9 μL的ddH2O。PCR扩增程序为97℃预变性3min; 98℃变性15s, 退火15s, 72℃延伸20s, 循环35次; 最后72℃末延伸5min, 4℃保存, 完成PCR过程。对PCR产物进行琼脂糖凝胶(1%)电泳检验, 选取目的条带清晰且明亮的样品送往天一辉远生物科技有限公司, 完成纯化回收和测序工作。
表 1 本研究所用引物信息Table 1. Primer information used in the present study基因
Gene引物名称
Primer引物序列
Primer sequence (5′—3′)退火温度
Annealed
temperature (℃)Cyt b L14724 GACTTGAAA AACCACCGTTG 58 H15915 CTCCGATCTCCGGATTACAAGAC ND2 AFND2L AAGCTYTYGGGCCCATACC 58 AFND2R TCCYGCTTAGGGCTTTGAAGG myh6 myh6_F459 CATMTTYTCCATCTCAGATAATGC 53 myh6_R1325 ATTCTCACCACCATCCAGTTGAA RAG2 RAG2-f2a AARCGCTCMTGTCCMACTGG 55 RAG2-R6a TGRTCCARGCAGAAGTACTTG 1.3 数据分析
在GenBank数据库中下载了7尾鳤(2020年采集自长江干流公安江段)的4个基因序列[9], 和本研究获得的32尾鳤的4个基因序列一起用于数据分析。首先, 将所得测序结果参照序列峰图进行编辑和校正, 使用MEGA 7软件对序列进行对比[10], 并裁剪至陈蔚涛等[9]研究所用序列相同的长度。为获得更多遗传信息, 将2个线粒体基因和2个核基因分别合并进行后续分析。
使用DnaSP 5.10软件计算长江中游鳤群体的单倍型数(H)、多态位点数(S)、单倍型多样性(Hd)和核苷酸多样性(Pi)[11]。使用Popart 1.7软件中的中接法(Median-joining)构建长江中游鳤群体的单倍型网络图[12]。使用jModeltest 2.1.7软件以贝叶斯信息准则(BIC)计算出鳤单倍型的最佳核苷酸进化模型[13]。利用MrBayes 3.2.7软件构建长江中游鳤群体单倍型的BI系统发育树[14]。外类群选用GenBank数据库中公开的在珠江肇庆江段采集的鳡(Elopichthys bambusa), 运算马尔卡夫链(Markov chain Monte Carlo, MCMC)1000万代, 舍去前25%的数据, BI系统发育树的可视化和美化使用FigTree v 1.44软件进行。
使用Arlequin 3.0软件对鳤群体进行分子方差变异分析(AMOVA)并计算成对遗传分化指数(FST值)[15]。使用DnaSP 5.10软件对鳤群体进行错配分析, 并使用Arlequin 3.0软件对鳤群体进行中性检验(Tajima’s D 检验和 Fu’s Fs 检验)来检验长江中游鳤群体在历史上是否发生过种群扩张现象[11, 15]。如果群体不能拒绝种群扩张模型, 则可以利用公式τ =2ut计算出种群扩张的大致时间。式中, τ为Tau, 即扩张时间参数, 可由Arelequin软件计算得到, t为扩张后经历的世代数, u代表每世代每条序列的变异速率, 可根据公式u=2μk计算得到; 式中, μ为每个碱基的变异速率, 联合基因的进化速率由Cyt b基因的进化速率估算获得, k是所研究序列长度(bp)[15, 16]。鳤初次性成熟时间约为3龄, 故本研究中世代时间为3年/世代, 扩张时间T=t×每世代的时间[3, 4]。
为了解长江中游和西江中下游鳤群体间的遗传差异, 在GenBank数据库中下载了52尾采集至西江中下游鳤的4个基因序列, 具体采集信息参考杨计平等[8]。将4个基因合并, 使用STRUCTURE v2.3.4分析群体的遗传组成及个体聚类情况。假定类群K从1到7, 在混合模型和相关基因频率下, 每个K值运行15次, 运行参数为(250000 iterations with 50000 burn-in periods)[17]。最佳K值通过在Structure Harvester上运行得出的LnP(K)和∆K值来确认(可在http://taylor0.biology.ucla.edu/structureHarvester/获取)[18]。
使用BEAST v2.7.0软件估算长江中游和西江中下游鳤群体间的分化时间[19]。由于缺乏有效的化石数据, 故采用严格的分子钟模型, 基于Yule先验模型, 最佳核苷酸进化模型使用jModeltest 2.1.7软件以贝叶斯信息准则(BIC)推算[13]。联合基因的进化速率由Cyt b的进化速率估算获得。1%是鲤科鱼类Cyt b基因普遍接受的一个进化速率[20—22], 联合基因的K2P平均遗传距离(0.7%)为Cyt b基因(1.1%)的0.633倍, 因此联合基因的进化速率约为0.633%[9]。马尔卡夫链(MCMC)设置108代, 舍弃前50%老化数据。运算结束后使用TreeAnnotator v.1.8.1软件总结出一致树, 并在FigTree v 1.44软件中显示节点的分化时间和后验概率[9]。
2. 结果
2.1 序列变异特征和单倍型系统发育分析
在序列经过比对后, 删减两端噪音序列, 最终获得Cyt b基因序列895 bp、ND2基因序列935 bp、myh6基因序列763 bp、RAG2基因序列857 bp, 线粒体联合基因序列(Cyt b+ND2)全长1830 bp, 核联合基因序列(myh6+RAG2)全长1620 bp。核基因的碱基组成比较平均(T=24.1%、C=25.3%、A=26.4%、G=24.2%), 而线粒体基因则存在向碱基A的偏倚(T=25.7%、C=23.2%、A=30.6%、G=20.4)。
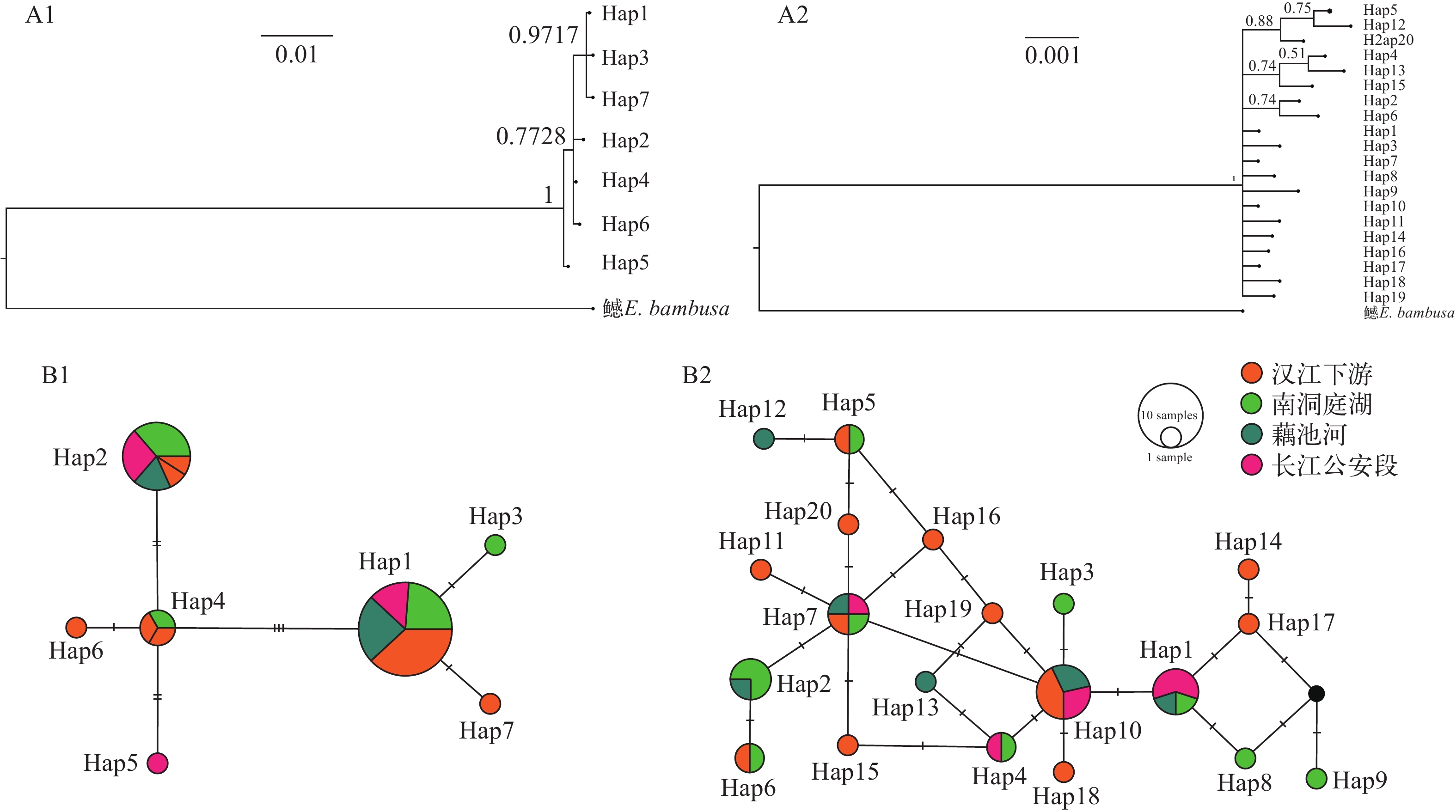
39条线粒体联合基因序列共计有10个变异位点, 分离出7个单倍型; 核联合基因共计有11个变异位点, 分离出20个单倍型(表 2)。无论是基于线粒体基因还是核基因的BI系统发育树, 均能看出长江中游鳤群体的单倍型间没有明显的分支趋势, 各单倍型间差异较小, 一般只有1—3个碱基对的差异, 同时基于单倍型网络图也可看出长江中游鳤群体的单倍型分布没有明显的系统地理格局(图 2)。
表 2 长江中游鳤群体单倍型序列的变异位点Table 2. Variation loci of haplotype sequence from O. elongatus population in the middle reaches of the Yangtze River序列
SequenceCyt b ND2 122 203 323 400 281 317 364 372 741 877 Hap1 T A A G G T C G G G Hap2 C G T A C Hap3 A Hap4 C T A Hap5 C T A T A Hap6 C T A A Hap7 A 序列
SequenceRAG2 myh6 30 277 353 398 523 669 24 186 228 507 606 Hap1 C G T C C C C G C G C Hap2 A A A Hap3 A T Hap4 A G Hap5 A A A T Hap6 A A Hap7 A A Hap8 T Hap9 T C T Hap10 A Hap11 T A A Hap12 A A A A T Hap13 A G T Hap14 C A Hap15 A G A Hap16 A A T Hap17 C Hap18 A T Hap19 A T Hap20 A A A ![]() 图 2 基于线粒体基因序列和核基因序列构建的长江中游鳤的单倍型关系图A1. 基于线粒体基因序列构建的长江中游鳤BI系统发育树; A2. 基于核基因序列构建的长江中游鳤BI系统发育树; B1. 基于线粒体基因序列构建的鳤单倍型网络图, 每个圆圈代表一种单倍型, 圆圈的大小与单倍型所含的个体数成正比例; B2. 基于核基因序列构建的鳤单倍型网络图Figure 2. Haplotypes relationship of O. elongatus from the middle reaches of the Yangtze River based on mitochondrial gene sequences and nuclear gene sequencesA1. Bayesian inference phylogenetic tree of haplotypes of O. elongatus from the middle reaches of the Yangtze River based on mitochondrial gene sequences; A2. Bayesian inference phylogenetic tree of haplotypes of O. elongatus from the middle reaches of the Yangtze River based on nuclear gene sequences; B1. The haplotype network map of O. elongatus based on mitochondrial gene sequence, Each circle represents a haplotype, and its size is proportional to its total frequency; B2. The haplotype network map of O. elongatus based on nuclear gene sequence
图 2 基于线粒体基因序列和核基因序列构建的长江中游鳤的单倍型关系图A1. 基于线粒体基因序列构建的长江中游鳤BI系统发育树; A2. 基于核基因序列构建的长江中游鳤BI系统发育树; B1. 基于线粒体基因序列构建的鳤单倍型网络图, 每个圆圈代表一种单倍型, 圆圈的大小与单倍型所含的个体数成正比例; B2. 基于核基因序列构建的鳤单倍型网络图Figure 2. Haplotypes relationship of O. elongatus from the middle reaches of the Yangtze River based on mitochondrial gene sequences and nuclear gene sequencesA1. Bayesian inference phylogenetic tree of haplotypes of O. elongatus from the middle reaches of the Yangtze River based on mitochondrial gene sequences; A2. Bayesian inference phylogenetic tree of haplotypes of O. elongatus from the middle reaches of the Yangtze River based on nuclear gene sequences; B1. The haplotype network map of O. elongatus based on mitochondrial gene sequence, Each circle represents a haplotype, and its size is proportional to its total frequency; B2. The haplotype network map of O. elongatus based on nuclear gene sequence2.2 遗传多样性
基于线粒体基因计算, 长江中游鳤群体的整体单倍型多样性为0.638, 整体核苷酸多样性为0.141%。其中长江公安段群体的单倍型多样性和核苷酸多样性最高, 藕池河群体的单倍型多样性最低, 汉江下游群体的核苷酸多样性最低。基于核基因计算, 长江中游鳤群体的整体单倍型多样性为0.938, 整体核苷酸多样性为0.143%。其中汉江下游群体的单倍型多样性最高, 南洞庭湖群体的核苷酸多样性最高, 长江公安段群体的单倍型多样性和核苷酸多样性最低(表 3)。
表 3 长江中游4个鳤群体的遗传多样性Table 3. The genetic diversity of 4 O. elongatus populations from the middle reaches of the Yangtze River基因
Gene群体
Population序列数
Number of
sequence单倍型数
Number of
haplotype多态位点数
Number of
polymorphic site单倍型多样性
Haplotype
diversity核苷酸多样性
Nucleotide
diversity (%)线粒体基因
Mitochondrial gene汉江下游 14 5 7 0.670±0.126 0.126±0.026 南洞庭湖 11 4 6 0.709±0.099 0.155±0.021 藕池河 7 2 5 0.476±0.171 0.130±0.047 长江公安段 7 3 7 0.714±0.127 0.187±0.032 整体 39 7 10 0.638±0.063 0.141±0.013 核基因
Nuclear gene汉江下游 14 12 9 0.967±0.044 0.145±0.017 南洞庭湖 11 9 10 0.945±0.066 0.184±0.030 藕池河 7 6 6 0.952±0.096 0.147±0.032 长江公安段 7 4 3 0.810±0.130 0.071±0.016 整体 39 20 11 0.938±0.021 0.143±0.013 2.3 遗传分化
AMOVA分析表明, 无论是基于线粒体基因还是核基因, 群体内的遗传变异是长江中游鳤群体遗传变异的主要来源, 且固定系数FST值均不显著, 表明长江中游鳤群体间不存在遗传分化现象(表 4)。群体间成对遗传分化指数FST值表明, 无论是基于线粒体基因还是核基因, FST值所对应的P值均大于0.05, 表明长江中游4个鳤群体间不存在遗传分化现象(表 5和表 6)。
表 4 基于线粒体和核基因的4个鳤群体的分子方差变异分析Table 4. Analysis of molecular variance (AMOVA) for 4 O. elongatus populations based on mitochondrial and nuclear gene sequence基因
Gene变异来源
Source of variation自由度
df平方和
Sum of
square变异分量
Variance
component变异百分比
Percentage of
variationFST FST- P 线粒体基因
Mitochondrial gene群体间Among group 3 2.435 –0.05479 Va –4.30 –0.04297 0.65885 群体内
Among population within group35 46.539 1.32968 Vb 104.30 总计Total 38 48.974 1.27490 核基因
Nuclear gene群体间Among group 3 3.285 –0.00738 Va –0.64 –0.00637 0.49658 群体内
Among population within group35 40.766 1.16475 Vb 100.64 总计Total 38 44.051 1.15737 表 5 基于线粒体基因分析的鳤群体的成对FST值(对角线下)和校正P值(对角线上)Table 5. Pairwise FST values (below diagonal) and corrected P-values (above diagonal) among O. elongatus populations based on mitochondrial gene sequence汉江下游 南洞庭湖 藕池河 长江公安段 汉江下游 0.51367 0.82227 0.23047 南洞庭湖 –0.02471 0.62891 0.75195 藕池河 –0.08530 –0.08360 0.59863 长江公安段 0.02486 –0.09251 –0.03140 表 6 基于核基因分析的鳤群体的成对FST (对角线下)和校正P值(对角线上)Table 6. Pairwise FST values (below diagonal) and corrected P-values (above diagonal) among O. elongatus populations based on nuclear gene sequence汉江下游 南洞庭湖 藕池河 长江公安段 汉江下游 0.63770 0.92578 0.11914 南洞庭湖 0.01939 0.84863 0.22656 藕池河 0.06338 0.05857 0.24121 长江公安段 0.05997 0.04487 0.05128 2.4 历史动态
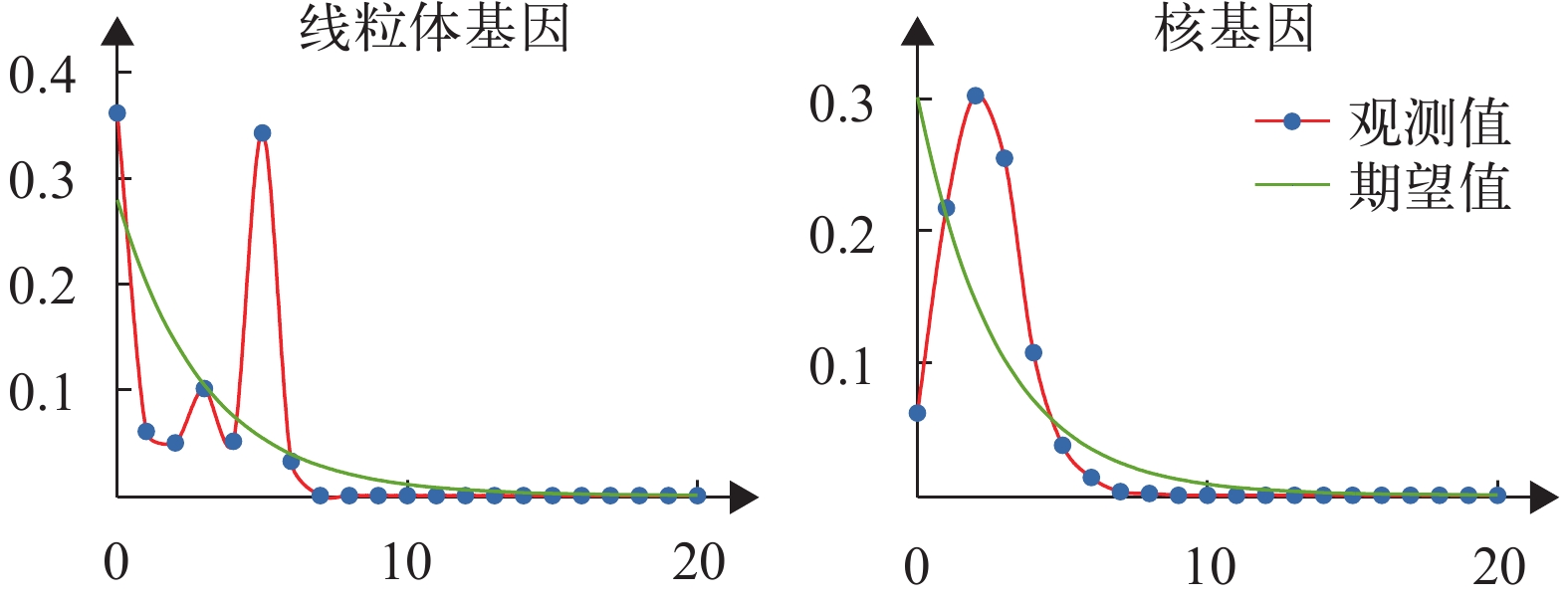
基于线粒体基因的中性检验显示, 无论是Tajima’s D检验还是Fu’s Fs检验的结果均为不显著的正值, 说明长江中游鳤群体的突变没有偏离中性模型, 暗示长江中游鳤群体在历史上可能没有发生过种群扩张现象(表 7); 同时错配分析结果呈现双峰分布, 也支持长江中游鳤群体在历史上没有发生过种群扩张这一观点(图 3)。
表 7 基于线粒体基因和核基因的长江中游鳤群体的中性检验Table 7. Neutrality test of O. elongatus populations in the middle reaches of the Yangtze River based on mitochondrial and nuclear gene指标
Index线粒体基因
Mitochondrial
gene核基因
Nuclear
gene样本量Number of sample 39 39 多态位点Number of polymorphic site 10 11 Tajima’s D 0.26912 –0.33201 Tajima’s D-P 0.66200 0.40700 Fu’s Fs 0.82408 –15.38510 Fu’s Fs- P 0.67600 0.00000 扩张时间(万年前) Expansion time(ten thousand years B.P.) 16.54 而基于核基因的中性检验显示, Tajima’s D检验结果为负值, 但结果不显著, Fu’s Fs检验结果为较大的负值, 且达到极显著的水平, 暗示长江中游鳤群体可能存在一个种群扩张的历史(表 7)。同时错配分析的观测值为单峰的泊松分布(图 3), 表明长江中游鳤群体在历史上可能发生过种群扩张现象, 根据公式推算出扩张时间约为16.54万年前(进化速率为0.67%每百万年, 世代时间为3年/世代)。
2.5 分化时间推算
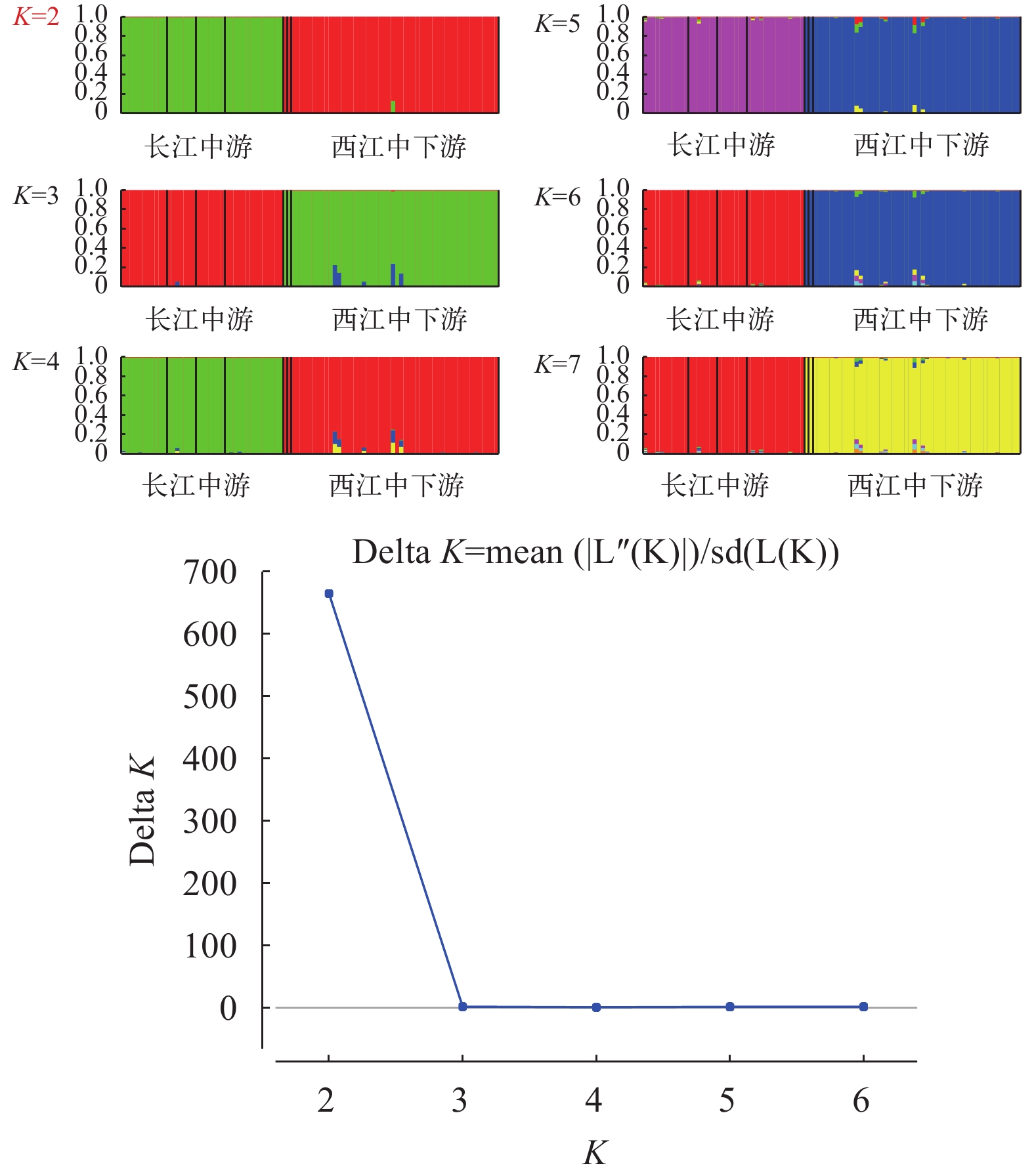
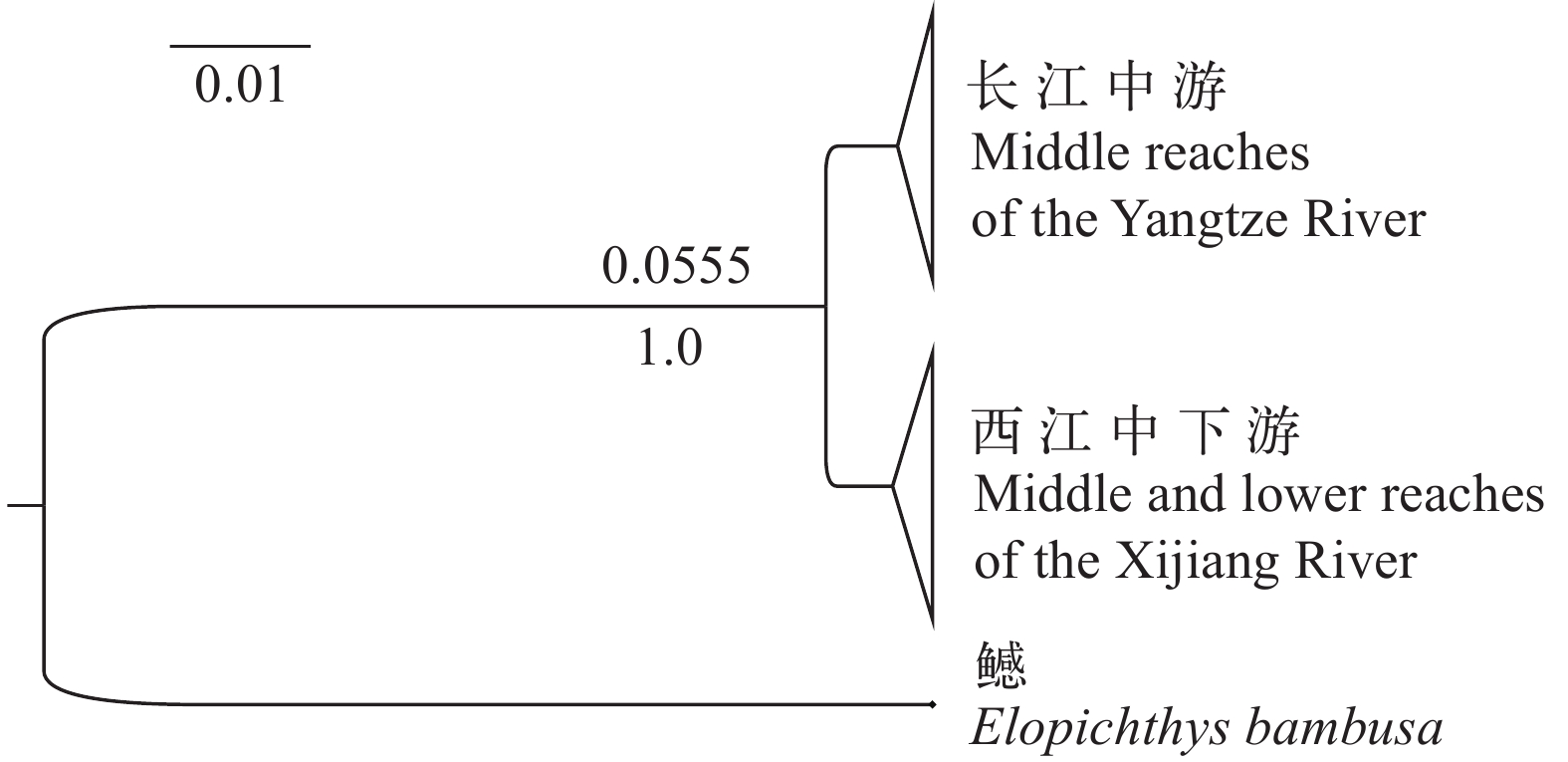
图 4展示了K=2—7时鳤群体遗传簇的分布情况, 可以清晰地看到, 无论K值为多少时, 长江中游和西江中下游的鳤群体存在显著的遗传差异。ΔK在K=2时达到最大值, 表明长江中游和西江中下游的鳤应划分为两个遗传簇, 即长江中游群体和西江中下游群体各自形成了一个遗传簇。BEAST最终运算的有效取样大小(Effect sample size, ESS)均大于200, 表明运算是收敛的。基于0.633%每百万年进化速率估算可知长江中游鳤群体和西江中下游鳤群体的分化时间约在0.0555百万年前(即5.55万年前, 图 5)。
![]() 图 5 基于联合基因的长江中游和西江中下游鳤群体分化时间推算分支上方数值表示分化时间, 分支下方数值表示后验概率Figure 5. Estimation of divergence time of O. elongatus in the middle reaches of the Yangtze River, and the middle and lower reaches of the Xijiang River on mitochondrial and nuclear geneThe value above the branch represents the divergence time, and the value below the branch represents the posterior probability
图 5 基于联合基因的长江中游和西江中下游鳤群体分化时间推算分支上方数值表示分化时间, 分支下方数值表示后验概率Figure 5. Estimation of divergence time of O. elongatus in the middle reaches of the Yangtze River, and the middle and lower reaches of the Xijiang River on mitochondrial and nuclear geneThe value above the branch represents the divergence time, and the value below the branch represents the posterior probability3. 讨论
3.1 遗传多样性
较高的遗传多样性是生物能长期存在的必要条件, 单倍型多样性和核苷酸多样性是评估生物遗传多样性的两个重要指标[23, 24]。无论是基于线粒体基因还是核基因标记, 长江中游鳤群体的单倍型多样性均处于较高水平(>0.5), 而核苷酸多样性均处于较低水平(<0.5%)[25]。长江中游鳤的遗传多样性高于三峡库区似鳊(基于线粒体Cyt b基因, Hd=0.213、Pi=0.022%)[16], 但低于长江上游鲢、蛇鮈(基于线粒体Cyt b基因, Hd=0.849、Pi=1.438%; Hd=0.872、Pi=0.400%)[26, 27]等群体, 处于中等水平。和珠江水系相比, 基于线粒体基因, 长江中游鳤的单倍型多样性和核苷酸多样性均低于西江中下游鳤(Hd=0.940, Pi=0.219%)[8]; 但基于核基因, 长江中游鳤的单倍型多样性和核苷酸多样性明显高于西江中下游鳤(Hd=0.546, Pi=0.040%)[8]。
长江中游鳤群体线粒体基因和核基因的多态位点数和核苷酸多样性相差不大, 但基于线粒体基因的单倍型多样性明显低于核基因, 这可能和线粒体基因标记的母系遗传有关。线粒体基因组是典型的单亲标记, 其有效种群大小比常染色体DNA小, 在瓶颈效应、遗传漂变的作用下, 其单倍型较常染色体DNA更容易丢失[28, 29]。鳤曾是长江流域的重要的经济鱼类, 种群数量较大; 但近些年长江中游鳤资源急剧萎缩, 甚至一度在长江中游流域绝迹, 遭受了瓶颈效应和遗传漂变的作用。这些作用使线粒体基因丢失的单倍型多于核基因, 造成了长江中游鳤群体线粒体基因标记的单倍型多样性明显低于核基因标记的现象[28]。
3.2 遗传分化
群体间成对遗传分化指数(FST值)和AMOVA分析表明长江中游4个鳤群体间不存在遗传分化现象, 这可能和各采样点间相距较近, 且鳤迁移能力较强有关[30]。本研究中各采样点间相距约几十公里, 且无水电站等地理阻隔, 群体之间可以自由交流。其次, 鳤作为江湖洄游性鱼类, 游泳能力较强, 这为鳤在各群体间进行基因交流提供了可能[3, 4]。同时, 鳤产漂流性鱼卵, 受精卵在发育过程中会随水流漂流较远的距离, 这也可能加强了长江中游各鳤群体间的基因交流[31—33]。
基于STRUCTURE分析可以看出长江中游鳤群体和西江中下游鳤群体间存在明显的遗传差异, 说明长江中游和西江中下游的鳤群体是两个独立遗传的谱系, 这和陈蔚涛等[7]、范凤娟等[9]的研究相吻合。基于BEAST推算出的两水系鳤的分化时间约在5.55万年前, 说明长江中游和西江中下游的鳤群体间经历了较长时间的隔离[7]。
3.3 种群历史动态
中性检验可以检验一个种群内稀有等位基因是否以高频存在, 进而判断种群是否经历过定向选择或种群扩张的历史。基于线粒体基因序列和核基因序列得到了不同的长江中游鳤的种群历史动态, 出现了核质不统一现象, 线粒体基因分析结果表明长江中游鳤没有发生过种群扩张, 而核基因分析结果表明长江中游鳤发生过种群扩张。线粒体基因组的有效种群大小往往比常染色体DNA小, 更容易丢失稀有等位基因, 使我们对种群历史动态可能做出错误的判断[28]。其次线粒体基因组为严格的母系遗传, 其结果在一定程度上不能完整代表物种的历史动态[28]。综合线粒体基因和核基因标记的结果, 我们认为长江中游鳤群体可能在16.54万年前发生过种群扩张现象。
在进入第四纪后, 全球气候出现明显的冰期和间冰期交替的模式, 大致划分为大理冰期(0.07—0.01 MaB.P.)、庐山冰期(0.37—0.24 MaB.P.)、大姑冰期(0.8—0.68 MaB.P.)和鄱阳冰期(1.2—0.9 MaB.P.)四个寒冷的时期[34]。而在两个冰期间的间冰期气候较为温暖, 一般而言间冰期和冰期气温相差可达12℃, 间冰期温暖的气候往往会促进生物的扩张[35, 16]。长江中游鳤群体可能在16.54万年前发生过种群扩张现象, 此时正处于庐山冰期和大理冰期间的间冰期, 气候较为温暖, 生物普遍开始从冰期避难所向外扩张, 如长江中游䱗群体也是在这一时间段内发生过种群扩张现象(0.17和0.24 MaB.P.)[36]。
4. 结论
鳤曾经是长江流域的重要经济鱼类, 但近年来长江流域鳤资源严重衰退, 已经多年未被监测到。长江十年禁渔政策实施后, 长江鳤资源有所回升。本研究利用两个线粒体基因和两个核基因标记初步解析了长江中游鳤群体的遗传结构, 发现长江中游鳤群体的遗传多样性处于中等水平, 这说明恢复长江流域鳤资源有良好的遗传基础。同时也印证了长江中游鳤的资源衰退并不是因为低遗传多样性造成的, 而可能是因为过度捕捞、江湖阻隔、栖息地破坏等人为因素的影响所致。
陈蔚涛等[9]的研究发现长江公安段和西江中下游鳤群体间存在较大的遗传差异, 提出在保护策略制定和实施上应该区别对待这两个鳤群体。本研究将长江中游4个鳤群体和西江中下游鳤群体进行对比, 发现长江中游鳤群体间不存在遗传分化现象, 而长江中游群体和西江中下游群体间存在较大的遗传差异, 这印证了这两水系的鳤群体已经形成了独立的谱系, 应当视作两个进化显著单元(Evolutionary Significant Unit, ESU), 在制定保护策略时应当设立长江中游和西江中下游两个保护单元。
本研究虽初步解析了长江中游鳤群体的遗传多样性和遗传结构, 但受限于样本数量, 研究结果仍然存在一些局限性。待未来长江流域鳤资源恢复、种群数量增加后, 应扩大研究区域、增大样本量、使用SNP等分辨率更高的分子标记, 进一步深入探讨长江中游鳤群体的遗传多样性和遗传结构。
-
图 2 基于线粒体基因序列和核基因序列构建的长江中游鳤的单倍型关系图
A1. 基于线粒体基因序列构建的长江中游鳤BI系统发育树; A2. 基于核基因序列构建的长江中游鳤BI系统发育树; B1. 基于线粒体基因序列构建的鳤单倍型网络图, 每个圆圈代表一种单倍型, 圆圈的大小与单倍型所含的个体数成正比例; B2. 基于核基因序列构建的鳤单倍型网络图
Figure 2. Haplotypes relationship of O. elongatus from the middle reaches of the Yangtze River based on mitochondrial gene sequences and nuclear gene sequences
A1. Bayesian inference phylogenetic tree of haplotypes of O. elongatus from the middle reaches of the Yangtze River based on mitochondrial gene sequences; A2. Bayesian inference phylogenetic tree of haplotypes of O. elongatus from the middle reaches of the Yangtze River based on nuclear gene sequences; B1. The haplotype network map of O. elongatus based on mitochondrial gene sequence, Each circle represents a haplotype, and its size is proportional to its total frequency; B2. The haplotype network map of O. elongatus based on nuclear gene sequence
图 5 基于联合基因的长江中游和西江中下游鳤群体分化时间推算
分支上方数值表示分化时间, 分支下方数值表示后验概率
Figure 5. Estimation of divergence time of O. elongatus in the middle reaches of the Yangtze River, and the middle and lower reaches of the Xijiang River on mitochondrial and nuclear gene
The value above the branch represents the divergence time, and the value below the branch represents the posterior probability
表 1 本研究所用引物信息
Table 1 Primer information used in the present study
基因
Gene引物名称
Primer引物序列
Primer sequence (5′—3′)退火温度
Annealed
temperature (℃)Cyt b L14724 GACTTGAAA AACCACCGTTG 58 H15915 CTCCGATCTCCGGATTACAAGAC ND2 AFND2L AAGCTYTYGGGCCCATACC 58 AFND2R TCCYGCTTAGGGCTTTGAAGG myh6 myh6_F459 CATMTTYTCCATCTCAGATAATGC 53 myh6_R1325 ATTCTCACCACCATCCAGTTGAA RAG2 RAG2-f2a AARCGCTCMTGTCCMACTGG 55 RAG2-R6a TGRTCCARGCAGAAGTACTTG 表 2 长江中游鳤群体单倍型序列的变异位点
Table 2 Variation loci of haplotype sequence from O. elongatus population in the middle reaches of the Yangtze River
序列
SequenceCyt b ND2 122 203 323 400 281 317 364 372 741 877 Hap1 T A A G G T C G G G Hap2 C G T A C Hap3 A Hap4 C T A Hap5 C T A T A Hap6 C T A A Hap7 A 序列
SequenceRAG2 myh6 30 277 353 398 523 669 24 186 228 507 606 Hap1 C G T C C C C G C G C Hap2 A A A Hap3 A T Hap4 A G Hap5 A A A T Hap6 A A Hap7 A A Hap8 T Hap9 T C T Hap10 A Hap11 T A A Hap12 A A A A T Hap13 A G T Hap14 C A Hap15 A G A Hap16 A A T Hap17 C Hap18 A T Hap19 A T Hap20 A A A 表 3 长江中游4个鳤群体的遗传多样性
Table 3 The genetic diversity of 4 O. elongatus populations from the middle reaches of the Yangtze River
基因
Gene群体
Population序列数
Number of
sequence单倍型数
Number of
haplotype多态位点数
Number of
polymorphic site单倍型多样性
Haplotype
diversity核苷酸多样性
Nucleotide
diversity (%)线粒体基因
Mitochondrial gene汉江下游 14 5 7 0.670±0.126 0.126±0.026 南洞庭湖 11 4 6 0.709±0.099 0.155±0.021 藕池河 7 2 5 0.476±0.171 0.130±0.047 长江公安段 7 3 7 0.714±0.127 0.187±0.032 整体 39 7 10 0.638±0.063 0.141±0.013 核基因
Nuclear gene汉江下游 14 12 9 0.967±0.044 0.145±0.017 南洞庭湖 11 9 10 0.945±0.066 0.184±0.030 藕池河 7 6 6 0.952±0.096 0.147±0.032 长江公安段 7 4 3 0.810±0.130 0.071±0.016 整体 39 20 11 0.938±0.021 0.143±0.013 表 4 基于线粒体和核基因的4个鳤群体的分子方差变异分析
Table 4 Analysis of molecular variance (AMOVA) for 4 O. elongatus populations based on mitochondrial and nuclear gene sequence
基因
Gene变异来源
Source of variation自由度
df平方和
Sum of
square变异分量
Variance
component变异百分比
Percentage of
variationFST FST- P 线粒体基因
Mitochondrial gene群体间Among group 3 2.435 –0.05479 Va –4.30 –0.04297 0.65885 群体内
Among population within group35 46.539 1.32968 Vb 104.30 总计Total 38 48.974 1.27490 核基因
Nuclear gene群体间Among group 3 3.285 –0.00738 Va –0.64 –0.00637 0.49658 群体内
Among population within group35 40.766 1.16475 Vb 100.64 总计Total 38 44.051 1.15737 表 5 基于线粒体基因分析的鳤群体的成对FST值(对角线下)和校正P值(对角线上)
Table 5 Pairwise FST values (below diagonal) and corrected P-values (above diagonal) among O. elongatus populations based on mitochondrial gene sequence
汉江下游 南洞庭湖 藕池河 长江公安段 汉江下游 0.51367 0.82227 0.23047 南洞庭湖 –0.02471 0.62891 0.75195 藕池河 –0.08530 –0.08360 0.59863 长江公安段 0.02486 –0.09251 –0.03140 表 6 基于核基因分析的鳤群体的成对FST (对角线下)和校正P值(对角线上)
Table 6 Pairwise FST values (below diagonal) and corrected P-values (above diagonal) among O. elongatus populations based on nuclear gene sequence
汉江下游 南洞庭湖 藕池河 长江公安段 汉江下游 0.63770 0.92578 0.11914 南洞庭湖 0.01939 0.84863 0.22656 藕池河 0.06338 0.05857 0.24121 长江公安段 0.05997 0.04487 0.05128 表 7 基于线粒体基因和核基因的长江中游鳤群体的中性检验
Table 7 Neutrality test of O. elongatus populations in the middle reaches of the Yangtze River based on mitochondrial and nuclear gene
指标
Index线粒体基因
Mitochondrial
gene核基因
Nuclear
gene样本量Number of sample 39 39 多态位点Number of polymorphic site 10 11 Tajima’s D 0.26912 –0.33201 Tajima’s D-P 0.66200 0.40700 Fu’s Fs 0.82408 –15.38510 Fu’s Fs- P 0.67600 0.00000 扩张时间(万年前) Expansion time(ten thousand years B.P.) 16.54 -
[1] Pinsky M L, Palumbi S R. Meta-analysis reveals lower genetic diversity in overfished populations [J]. Molecular Ecology, 2014, 23(1): 29-39. doi: 10.1111/mec.12509
[2] Willi Y, Kristensen T N, Sgrò C M, et al. Conservation genetics as a management tool: The five best-supported paradigms to assist the management of threatened species [J]. Proceedings of the National Academy of Sciences of the United States of America, 2022, 119(1): e2105076119.
[3] 陈宜瑜. 中国动物志, 硬骨鱼纲, 鲤形目(中卷) [M]. 北京: 科学出版社, 1998: 106-107. Chen Y Y. Fauna Sinica, Osteichthyes Cypriniformes Ⅱ [M]. Beijing: Science Press, 1998: 106-107.
[4] 湖北省水生生物研究所鱼类研究室. 长江鱼类 [M]. 北京: 科学出版社, 1976: 91-93. Fish Laboratory, Hubei Institute of Hydrobiology. The fish of Yangtze River [M]. Beijing: Science Press, 1976: 91-93.
[5] 李捷, 李新辉, 贾晓平, 等. 西江鱼类群落多样性及其演变 [J]. 中国水产科学, 2010, 17(2): 298-311. Li J, Li X H, Jia X P, et al. Evolvement and diversity of fish community in Xijiang River [J]. Journal of Fishery Sciences of China, 2010, 17(2): 298-311.
[6] 蒋志刚, 江建平, 王跃招, 等. 中国脊椎动物红色名录 [J]. 生物多样性, 2016, 24(5): 500-551. doi: 10.17520/biods.2016076 Jiang Z G, Jiang J P, Wang Y Z, et al. Red list of China’s vertebrates [J]. Biodiversity Science, 2016, 24(5): 500-551. doi: 10.17520/biods.2016076
[7] 范凤娟, 章群. 汩罗江与柳江鳤鱼线粒体细胞色素b基因的遗传变异初探 [J]. 生态科学, 2009, 28(5): 457-459. Fan F J, Zhang Q. Preliminary study on genetic variation of mitochondrial DNA cytochrome b gene of Ochetobius elogatus in Miluojiang River and Liujiang River [J]. Ecological Science, 2009, 28(5): 457-459.
[8] 杨计平, 李策, 陈蔚涛, 等. 西江中下游鳤的遗传多样性与种群动态历史 [J]. 生物多样性, 2018, 26(12): 1289-1295. doi: 10.17520/biods.2018121 Yang J P, Li C, Chen W T, et al. Genetic diversity and population demographic history of Ochetobius elongatus in the middle and lower reaches of the Xijiang River [J]. Biodiversity Science, 2018, 26(12): 1289-1295. doi: 10.17520/biods.2018121
[9] 陈蔚涛, 段辛斌, 高雷, 等. 多基因解析长江与珠江鳤的遗传结构 [J]. 南方水产科学, 2022, 18(6): 19-25. Chen W T, Duan X B, Gao L, et al. Genetic structure analysis of Ochetobius elongatus between Yangtze River and Pearl River using multiple loci [J]. South China Fisheries Science, 2022, 18(6): 19-25.
[10] Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets [J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. doi: 10.1093/molbev/msw054
[11] Librado P, Rozas J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data [J]. Bioinformatics, 2009, 25(11): 1451-1452. doi: 10.1093/bioinformatics/btp187
[12] Leigh J W, Bryant D. Popart: full-feature software for haplotype network construction [J]. Methods in Ecology and Evolution, 2015, 6(9): 1110-1116. doi: 10.1111/2041-210X.12410
[13] Darriba D, Taboada G L, Doallo R, et al. jModelTest 2: more models, new heuristics and parallel computing [J]. Nature Methods, 2012, 9(8): 772.
[14] Ronquist F, Teslenko M, Van der Mark P, et al. MrBayes 3.2: efficient Bayesian phylogenetic inference and model choice across a large model space [J]. Systematic Biology, 2012, 61(3): 539-542. doi: 10.1093/sysbio/sys029
[15] Excoffier L, Laval G, Schneider S. Arlequin (version 3.0): an integrated software package for population genetics data analysis [J]. Evolutionary Bioinformatics Online, 2007, 1: 47-50.
[16] 王东, 罗进勇, 熊飞, 等. 三峡库区干支流似鳊群体的遗传结构 [J]. 水生态学杂志, 2023, 44(4): 124-131. Wang D, Luo J Y, Xiong F, et al. Genetic structure of Pseudobrama simoni populations in the mainstream and tributaries of Three Gorges Reservoir [J]. Journal of Hydroecology, 2023, 44(4): 124-131.
[17] Pritchard J K, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data [J]. Genetics, 2000, 155(2): 945-959. doi: 10.1093/genetics/155.2.945
[18] Earl D A, VonHoldt B M. STRUCTURE HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method [J]. Conservation Genetics Resources, 2012, 4(2): 359-361. doi: 10.1007/s12686-011-9548-7
[19] Suchard M A, Lemey P, Baele G, et al. Bayesian phylogenetic and phylodynamic data integration using BEAST 1.10 [J]. Virus Evolution, 2018, 4(1): vey016.
[20] Ketmaier V, Bianco P G, Cobolli M, et al. Molecular phylogeny of two lineages of Leuciscinae cyprinids (Telestes and Scardinius) from the peri-Mediterranean area based on cytochrome b data [J]. Molecular Phylogenetics and Evolution, 2004, 32(3): 1061-1071. doi: 10.1016/j.ympev.2004.04.008
[21] Durand J D, Tsigenopoulos C S, Unlü E, et al. Phylogeny and biogeography of the family Cyprinidae in the middle East inferred from cytochrome b DNA-evolutionary significance of this region [J]. Molecular Phylogenetics and Evolution, 2002, 22(1): 91-100. doi: 10.1006/mpev.2001.1040
[22] 高嘉昕, 俞丹, 刘焕章. 基于cyt b基因的长江中上游大鳞马口鱼遗传多样性及谱系生物地理学过程分析 [J]. 四川动物, 2021, 40(4): 361-373. Gao J X, Yu D, Liu H Z. Genetic diversity and phylogeography of Opsariichthys macrolepis from the upper and middle Yangtze River based on cytochrome b sequences [J]. Sichuan Journal of Zoology, 2021, 40(4): 361-373.
[23] Féral J P. How useful are the genetic markers in attempts to understand and manage marine biodiversity [J]? Journal of Experimental Marine Biology and Ecology, 2002, 268(2): 121-145. doi: 10.1016/S0022-0981(01)00382-3
[24] Vrijenhoek R C. Genetic diversity and fitness in small populations [J]. Conservation Genetics, 1994(68): 37-53.
[25] Grant W S, Bowen B W. Shallow population histories in deep evolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation [J]. Journal of Heredity, 1998, 89(5): 415-426. doi: 10.1093/jhered/89.5.415
[26] 翟东东, 蔡金, 喻记新, 等. 长江上游鲢群体遗传多样性和遗传分化 [J]. 北京师范大学学报(自然科学版), 2021, 57(2): 274-282. Zhai D D, Cai J, Yu J X, et al. Genetic diversity and population differentiation of silver carp (Hypophthalmichthys molitrix) in the upper reaches of the Yangtze River [J]. Journal of Beijing Normal University (Natural Science), 2021, 57(2): 274-282.
[27] 李小兵, 唐琼英, 俞丹, 等. 长江上游干流及赤水河蛇鮈遗传多样性与种群历史分析 [J]. 动物学杂志, 2016, 51(5): 833-843. Li X B, Tang Q Y, Yu D, et al. Genetic diversity and population history of longnose gudgeon (Saurogobio dabryi) in the upper Yangtze River and Chishui River based on cytochrome b gene sequences [J]. Chinese Journal of Zoology, 2016, 51(5): 833-843.
[28] Joanna R. F, Heather K, Stephen D. P, 等. 分子生态学: 第二版 [M]. 北京: 高等教育出版社, 2015: 37-40. Joanna R. F, Heather K, Stephen D. P, et al. Molecular Ecology. second Edittion [M]. Beijing, Higher Education Press, 2015: 37-40.
[29] Allio R, Donega S, Galtier N, et al. Large variation in the ratio of mitochondrial to nuclear mutation rate across animals: implications for genetic diversity and the use of mitochondrial DNA as a molecular marker [J]. Molecular Biology and Evolution, 2017, 34(11): 2762-2772. doi: 10.1093/molbev/msx197
[30] Suárez D, Arribas P, Jiménez-García E, et al. Dispersal ability and its consequences for population genetic differentiation and diversification [J]. Proceedings Biological Sciences, 2022, 289(1975): 20220489.
[31] Araujo-Lima C A R M, Oliveira E C. Transport of larval fish in the Amazon [J]. Journal of Fish Biology, 1998(53): 297-306. doi: 10.1111/j.1095-8649.1998.tb01033.x
[32] Tyus H M, Haines G B. Distribution, habitat use, and growth of age-0 Colorado squawfish in the green river basin, Colorado and Utah [J]. Transactions of the American Fisheries Society, 1991, 120(1): 79-89. doi: 10.1577/1548-8659(1991)120<0079:DHUAGO>2.3.CO;2
[33] Zitek A, Schmutz S, Unfer G, et al. Fish drift in a Danube sidearm-system: Ⅰ. Site-, inter- and intraspecific patterns [J]. Journal of Fish Biology, 2004, 65(5): 1319-1338. doi: 10.1111/j.0022-1112.2004.00533.x
[34] 景才瑞, 刘会平. 论中国第四纪冰期与间冰期 [J]. 成都理工学院学报, 1999, 26(1): 97-100. Jing C R, Liu H P. On the glacial and interglacial stages in quaternary of China [J]. Journal of Chengdu University of Technology (Science & Technology Edition), 1999, 26(1): 97-100.
[35] 刘嘉麒, 倪云燕, 储国强. 第四纪的主要气候事件 [J]. 第四纪研究, 2001, 21(3): 239-248. Liu J Q, Ni Y Y, Chu G Q. Main palaeoclimatic events in the quaternary [J]. Quaternary Sciences, 2001, 21(3): 239-248.
[36] 范启, 何舜平. 长江流域䱗种群遗传多样性和遗传结构分析 [J]. 水生生物学报, 2014, 38(4): 627-635. Fan Q, He S P. The pattern of upper and middle Yangtze drainages shapes the genetic structure and diversity of Hemiculter leucisculus revealed by mitochondrial DNA locus [J]. Acta Hydrobiologica Sinica, 2014, 38(4): 627-635.



 下载:
下载: