MOLECULAR DIVERSITIES OF PLANKOTIC MICROBIAL EUKARYOTES IN THE PEARL RIVER AND THEIR RELATIONSHIP WITH WATER ENVIRONMENT
-
摘要: 文章分别在丰水期和枯水期于珠江广州段和西江广东段43个样点采集水样, 基于末端限制性长度多态性分析, 研究了珠三角地区珠江水体的浮游真核微型生物的分子多样性及其群落结构和水体理化因子的关系。结果表明, 西江广东段及珠江广州段水体氮和磷严重超标, 水质状况堪忧。枯水期样本浮游真核微型生物香农威尔多样性指数普遍高于丰水期样本, 西江广东段样本浮游真核微型生物多样性指数普遍高于珠江广州段样本。不同水期, 不同区域的浮游真核微型生物群落结构均存在显著差异。珠江样点的浮游真核微型生物群落结构均与化学需氧量、高锰酸盐指数、氨氮、总氮、总磷高度相关; 但不同水期、不同区域的浮游真核微型生物群落结构与理化因子相关系数有所差异。此外, 分别筛选出了1个、8个末端限制性片段代表了潜在的敏感(Cystobasidium sp.或Protostelium nocturnum)、耐污(Acanthamoeba hatchetti、Babesia bicornis、Blastocystis sp.、Botryosphaerella sudetica、Candida caryicola、Coccomyxa simplex、Cryptomonas ovata、Filos agilis、Stenophora robusta、Sulfonecta uniserialis、Theileria sp.等)物种/类群。Abstract: To analyze the molecular diversity of planktonic microbial eukaryotes as well as relationships between the community structures and physicochemical factors based on the terminal restriction fragment length polymorphism (T-RFLP), water samples were collected from 43 sites in Guangzhou Reach of Pearl River and Guangdong Reach of Xijiang River during wet season and dry season, respectively. The results revealed that water bodies of Guangzhou Reach of Pearl River and Guangdong Reach of Xijiang River were seriously polluted by nitrogen and phosphorus, and the water were in poor quality. The diversity indexes of planktonic microbial eukaryotes in Guangdong Reach of Xijiang River were higher than those in Guangzhou Reach of Pearl River. The Shannon-Wiener indexes of samples collected in wet season were lower than those in dry season. There were significant differences in community structures of microbial eukaryotes from different seasons and regions. The community structures of planktonic microbial eukaryotes in Pearl River were affected by chemical oxygen demand, permanganate index, ammonia nitrogen, total nitrogen and total phosphorus. However, correlation coefficients between community structures and physicochemical factors were different depending on seasons and regions. In addition, one T-RF and six T-RFs were selected as possible sensitive species (Cystobasidium sp. or Protostelium nocturnum) and pollution-tolerant species (Acanthamoeba hatchetti, Babesia bicornis, Blastocystis sp., Botryosphaerella sudetica, Candida caryicola, Coccomyxa simplex, Cryptomonas ovata, Filos agilis, Stenophora robusta, Sulfonecta uniserialis and Theileria sp. etc.), respectively.
-
Keywords:
- The Pearl River /
- Microbial eukaryote /
- 18S rDNA /
- T-RFLP /
- Molecular diversity
-
珠江水资源总量丰富, 在全国七大江河中排名第二, 仅次于长江[1]。然而伴随着珠三角地区经济高速发展、城市规模不断扩大, 各种工业和生活污水、有机污染物不断迁移入珠江水域, 珠江下游水污染问题严重[2]。珠江主要支流西江和北江在流经广东省后污染状况同样不容乐观, 西江水质总体较差, 支流贺江水体整体处于中营养水平, 发生藻类水华的潜在风险较高[3]。珠江广州段和西江广东段承载着珠三角地区重要的城市供水任务, 与人民生活质量以及当地经济发展息息相关, 日趋严重的水污染所导致的水质性缺水, 已成为制约城市可持续发展的一个重要因素[4]。此外, 珠江流域处于热带-亚热带地区, 丰水期(4—9月)径流量大, 占全年径流总量的80%, 而枯水期(10月至翌年3月)的水量较少, 仅占全年径流总量的24%[5], 这意味着对珠江水质状况的研究有必要考虑丰水期及枯水期之间的差异, 但目前相关研究较少[6, 7]。
真核微型生物在水生生态系统中普遍存在, 主要包括原生动物、真核微藻、真菌及线虫等小型动物[8], 由于生命周期短、敏感性强等特点, 能迅速反映水环境变化[9, 10]。例如, 浮游藻类群落分布与温度和营养盐变化显著相关[11], 某些原生动物类群丰度与氨氮浓度呈显著正相关[12]。徐润林等[6]对珠江广州市段的原生动物群落组成和群集过程的研究发现, 原生动物群落特征的变化与水质变化相吻合, 珠江在流经广州市区后水质发生了明显地恶化。20世纪90年代, 沈韫芬等[13]便建立了适用于我国淡水生态环境的原生生物(真核微型生物)群落监测方法, 并成为我国第一个淡水生物监测标准。近年来, 徐奎栋等[14]将其改良并应用到海水环境监测中, 取得了较好的效果。
由于个体微小(通常为几十至几百微米), 基于形态鉴定基础的真核微型生物群落研究, 高度依赖于研究人员的鉴定经验, 在一定程度上限制了原生动物多样性研究及其在水质监测中的广泛应用。这也是珠江水体真核微型生物群落结构与环境因子的关系研究较少的重要原因之一[3, 6, 15]。末端限制性片段长度多态性分析(Terminal restriction fragment length polymorphism analysis, T-RFLP)可以定性和定量的进行微生物群落结构分析及不同群落结构间的比较[16]。目前, T-RFLP被广泛应用于水体微生物多样性及其与水环境关系的研究中, 例如: 环境因素对微生物的群落结构和功能的影响[17], 河流生态系统的健康评价[18], 指示生物的筛选[19]。
本文通过T-RFLP研究了珠三角地区珠江丰水期及枯水期浮游真核微型生物多样性, 对比分析了不同时期、不同区域真核微型生物的群落结构及其与环境因子的关系, 并筛选了潜在的敏感种和耐污种。以期较为全面地了解珠三角地区珠江水体中真核微型生物的动态变化, 为后续珠三角地区珠江水质监测提供可靠的数据参考。
1. 材料与方法
1.1 水样采集
采样点分为珠江广州段(G)和西江广东段(X), 分别在其丰水期(2016年6月—2016年9月)和枯水期(2016年10月—2017年1月)采集水样, 使用有机玻璃采水器采集表层水(30 cm—1 m)。其中, 丰水期设置了40个样点(西江广东段21个, 珠江广州段19个), 枯水期设置了43个样点(西江广东段21个, 珠江广州段22个, 图 1)。
附表 1 采样点信息 Appendix Tab. 1 Information of sampling sites 样品名Sample name 经度Longitude 纬度Latitude 样本描述Sample description 地点描述Location description GC1 113°47′37.211′′ 23°45′8.2548′′ 广州参照点1 流溪河国家森林公园 GC2 113°5′27.650′′ 23°9′2.988′′ 广州参照点2 思贤窖 GC3 112°53′8.001′′ 23°9′39.5604′′ 广州参照点3 三水区东平水道 GC4 113°20′45.229′′ 23°4′56.1755′′ 广州参照点4 海珠湿地公园 GC5 113°17′44.88″ 22°53′35.6855′′ 广州参照点5 顺德水道 GC6 113°37′26.4936′′ 23°6′44.5932′′ 广州参照点6 刘屋洲水道 GC7 113°11′46.7088′′ 23°26′20.0543′′ 广州参照点7 秀全水库 GG1 113°13′23.4875′′ 23°12′1.7748′′ 广州工业点1 石门水厂工业区 GG2 113°13′55.9344′′ 23°9′51.6455′′ 广州工业点2 松北工业区 GG3 113°18′13.2228′′ 23°3′45.2556″ 广州工业点3 南华西工业区 GJ1 113°14′42.0972″ 23°6′31.6691′′ 广州交通点1 芳村码头 GJ2 113°18′22.4604″ 23°6′35.3339′′ 广州交通点2 中大码头 GJ3 113°26′17.1095′′ 23°5′59.8524′′ 广州交通点3 嘉利码头 GN1 113°18′58.5503′′ 23°5′2.85′′ 广州农田点1 上冲果树公园 GN2 113°28′4.1016″ 23°7′9.966″ 广州农田点2 大沙北 GN3 113°29′8.7756″ 22°53′23.838″ 广州农田点3 东涌 GR1 113°37′48.2051′′ 22°45′34.9128″ 广州入海口1 南沙天后宫 GR2 113°29′30.4224′′ 23°3′55.134′′ 广州入海口2 黄埔大桥 GR3 113°40′18.1272″ 22°38′29.1623′′ 广州入海口3 新龙特大桥 GS1 113°13′52.1508″ 23°7′42.4739′′ 广州生活点1 珠岛花园 GS2 113°15′28.0835′′ 23°5′48.5411′′ 广州生活点2 太古仓 GS3 113°20′13.4484′′ 23°7′4.5552′′ 广州生活点3 猎德涌 XG1 112°48′9.0108″ 23°5′34.0475′′ 西江工业点1 肇庆市高要区 XG2 112°56′33.0539′′ 22°55′15.6792″ 西江工业点2 佛山市高明区 XG3 112°55′54.0156″ 22°44′4.4592″ 西江工业点3 江门市鹤山市 XG4 113°7′31.5372′′ 22°33′17.0352″ 西江工业点4 江门市江海区 XG5 113°30′21.0923′′ 22°16′44.4575′′ 西江工业点5 中山市坦洲镇 XJ1 112°48′17.7551′′ 23°5′58.2792′′ 西江交通点1 肇庆市高要区 XJ2 112°54′26.3556′′ 22°54′0.864″ 西江交通点2 佛山市高明区 XJ3 113°1′57.5436″ 22°48′23.2271′′ 西江交通点3 江门市鹤山市 XJ4 113°11′6.8459′′ 22°35′9.3227′′ 西江交通点4 江门市江海区 XJ5 113°25′28.9488′′ 22°13′8.1012′′ 西江交通点5 中山市坦洲镇 XN1 112°47′54.4019′′ 23°5′24.6911′′ 西江农田点1 肇庆市高要区 XN2 112°51′48.8592′′ 22°57′21.5136′′ 西江农田点2 佛山市高明区 XN3 113°4′52.176′′ 22°41′48.8112′′ 西江农田点3 江门市鹤山市 XN4 113°5′25.1592′′ 22°32′52.908′′ 西江农田点4 江门市江海区 XN5 113°24′40.5684′′ 22°13′34.6008′′ 西江农田点5 中山市坦洲镇 XR1 113°25′14.4587′′ 22°12′55.5731′′ 西江入海口 中山市坦洲镇 XS1 112°47′47.5008′′ 23°5′22.1316′′ 西江生活点1 肇庆市高要区 XS2 112°54′22.7664″ 22°54′5.4756″ 西江生活点2 佛山市高明区 XS3 112°57′38.0952′′ 22°46′33.6″ 西江生活点3 江门市鹤山市 XS4 113°5′27.6504′′ 22°32′26.9843′′ 西江生活点4 江门市江海区 XS5 113°31′16.4387′′ 22°16′25.4568′′ 西江生活点5 中山市坦洲镇 1.2 水质分析
现场使用便携式多参数测量仪(ORION 520M-01A)测定采样位点的水温(Temperature, Temp)、pH、溶解氧(Dissolved oxygen, DO)和盐度(Salinity)。其余理化指标参照《Water Analysis Handbook》(美国哈希公司)于实验室测定, 总氮(Total nitrogen, TN)的测定采用过硫酸盐氧化法(0.5—25.0 mg/L); 氨氮(Ammonia nitrogen, AN)的测定采用USEPA纳氏试剂法(0.02—2.5 mg/L)或水杨酸法(0.4—50 mg/L); 总磷(Total phosphorus, TP)的测定采用USEPA PhosVer 3抗坏血酸法(0.06—3.5 mg/L);化学需氧量(Chemical oxygen demand, CODCr)的测定采用USEPA消解比色法(3—150 mg/L); 高锰酸盐指数(Permanganate index, CODMn)的测定采用高锰酸盐法(0.5—5.0 mg/L)。
附表 2 理化因子数据 Appendix Tab. 2 Physicochemical factors 样本Sample 温度Temp (℃) pH 盐度Salinity (‰) 溶氧量DO (mg/L) 化学需氧量CODCr (mg/L) 高锰酸盐指数CODMn (mg/L) 氨氮AN (mg/L) 总氮TN (mg/L) 总磷TP (mg/L) 水质类别The rank of water quality GC1_1 27.4 7.82 0.088 8.2 8 1.46 0.12 0.2 0.037 Ⅰ、Ⅱ类 GC2_1 26.2 6.87 0.13 7.9 5 1.03 0.24 1.4 0.046 Ⅳ类 GC3_1 26.7 6.97 0.134 7.7 7 1.31 0.28 1.3 0.062 Ⅳ类 GC4_1 33 8.75 0.196 7.8 8 1.25 0.11 0.5 0.02 Ⅰ、Ⅱ类 GG1_1 27.9 7.07 0.232 1.2 70 15 9.9 12.2 0.435 劣Ⅴ类 GG2_1 27.8 7.13 0.265 0.2 101 30.9 18.5 20.4 0.705 劣Ⅴ类 GG3_1 31.1 7.44 0.191 5.4 23 8.46 3.4 5.1 0.186 劣Ⅴ类 GJ1_1 28.5 7.07 0.17 2.7 18 5.08 2.36 3.5 0.104 劣Ⅴ类 GJ2_1 30.5 7.06 0.167 3 8 3.3 2.6 3.6 0.092 劣Ⅴ类 GJ3_1 29.9 7.02 0.177 3.6 7 2.58 2.7 5.6 0.106 劣Ⅴ类 GN1_1 29.9 7 0.265 0.1 17 7.92 19.8 23.8 0.745 劣Ⅴ类 GN2_1 29.4 6.64 0.292 4.4 23 5.32 5.6 7.4 0.178 劣Ⅴ类 GN3_1 32 7.1 0.208 3.6 12 4.58 1.76 3.6 0.104 劣Ⅴ类 GR1_1 28.8 7.29 0.171 4.9 3.5 1.68 0.56 2.1 0.076 劣Ⅴ类 GR2_1 30.3 7.04 0.092 2.7 14 1.7 0.56 1.8 0.064 Ⅴ类 GR3_1 32.9 8 0.207 7 16 1.92 0.6 2 0.044 Ⅴ类 GS1_1 30.7 6.95 0.177 0.7 18 6.2 3.1 4.2 0.116 劣Ⅴ类 GS2_1 28.9 7.19 0.147 4.1 5 2.66 1.3 1.6 0.064 Ⅴ类 GS3_1 30 6.3 0.189 0.9 11 6.18 2 5.3 0.114 劣Ⅴ类 XG1_1 29.1 6.88 0.31 3.1 38 9.68 2.7 8 0.272 劣Ⅴ类 XG2_1 31.6 7.45 0.283 5.6 18 6.34 4.7 6.8 0.22 劣Ⅴ类 XG3_1 28 7.22 0.173 3.2 42 19.4 4 8.2 0.256 劣Ⅴ类 XG4_1 26.1 7.01 0.174 6.5 10 2.8 0.2 1.6 0.269 Ⅴ类 XG5_1 27.6 6.93 0.031 1.8 25 10.44 8.7 10 0.49 劣Ⅴ类 XJ1_1 26.9 7.6 0.206 7.6 4 1.3 0.21 3.6 0.086 劣Ⅴ类 XJ2_1 29.4 7.54 0.146 6.6 4.5 2.06 0.49 1.2 0.027 Ⅳ类 XJ3_1 27.3 7.51 0.155 6.8 2.8 1.38 0.5 0.6 0.037 Ⅲ类 XJ4_1 25.6 7.28 0.16 7.4 3.3 1.42 0.87 1.7 0.042 Ⅴ类 XJ5_1 27 7.03 0.152 6.5 16 2.31 0.48 1.1 0.042 Ⅳ类 XN1_1 29.5 6.87 0.418 2.8 32 9.16 3.5 8.6 0.342 劣Ⅴ类 XN2_1 30 4.4 0.175 6.88 14 4.08 1.02 3.2 0.082 劣Ⅴ类 XN3_1 27.6 6.02 0.191 7.7 12 1.76 0.4 0.8 0.056 Ⅲ类 XN4_1 26.8 7.42 0.172 5.6 2 1.5 1.3 2.7 0.061 劣Ⅴ类 XN5_1 29.4 6.94 0.252 6.6 14 2.93 0.52 1.4 0.09 Ⅳ类 XR1_1 26.5 6.7 0.151 6.5 12 3.26 0.42 1.4 0.078 Ⅳ类 XS1_1 28.9 6.81 0.473 3.2 25 8.72 2.2 8 0.304 劣Ⅴ类 XS2_1 29.2 7.52 0.144 6.5 10.6 2.42 0.61 1.4 0.035 Ⅳ类 XS3_1 29.8 7.04 0.219 2.8 36 8.06 7 9.6 0.262 劣Ⅴ类 XS4_1 26.2 7.56 0.163 6.7 9 1.66 0.1 0.8 0.03 Ⅲ类 XS5_1 29.6 7.13 0.217 0.1 150 52.2 33.5 37 0.95 劣Ⅴ类 GC1_2 20.8 7.23 0.083 7.6 3 1.04 0.06 2.3 0.055 劣Ⅴ类 GC2_2 18.8 7.71 0.155 8.4 8 2.06 0.32 2.2 0.104 劣Ⅴ类 GC3_2 18.6 7.69 0.152 8.1 5 1.52 0.43 2.4 0.098 劣Ⅴ类 GC4_2 19.1 8.31 0.214 9.8 3 1.74 0.01 0.5 0.055 Ⅰ、Ⅱ类 GC5_2 20.2 7.31 0.31 6.1 11.5 4.68 1.62 3.1 0.137 劣Ⅴ类 1.3 生物富集与DNA提取
将200 mL水体抽滤到0.8 μm滤膜(美国颇尔公司)上, 用灭菌镊子将滤膜取下, 在超净工作台上用灭菌剪刀将滤膜剪碎, 使用PowerSoil DNA Isolation Kit (美国Mobio)进行环境总DNA提取。
1.4 末端限制性片段长度多态性分析(T-RFLP)
利用引物TAReuk454FWD1 (5′-CCAGCA (G/C)C(C/T)GCGGTAATTCC-3′), TAReukREV3 (5′-ACTTTCGTTCTTGAT(C/T)(A/G)A-3′)[20]扩增真核微型生物18S rDNA的V4区。PCR体系为: 5 × FastPfu Buffer, 2.5 mmol/L dNTPs, 1U FastPfu DNA聚合酶(北京全式金生物技术有限公司), 引物各5 μmol/L, 约10 ng基因组DNA, 反应体系总量为20 μL。PCR反应程序: 95℃预变性5min; 95℃变性30s, 55℃退火30s, 72℃延伸45s, 共27个循环; 最后72℃延伸10min, 终止于4℃。PCR产物用1%琼脂糖电泳检验扩增效果, 并采用超薄DNA产物纯化试剂盒DP203-02(北京天根生化科技有限公司)纯化。
预实验选用了Apa Ⅰ、BamH Ⅰ、EcoR Ⅰ、Hind Ⅲ、Hinf Ⅰ、Hae Ⅲ等限制性核酸内切酶进行处理, 根据酶切效率、区分能力等因素, 最终选取Hae Ⅲ构建酶切体系: 10 μL回收产物, 2 μL QC缓冲液, 0.4 μL Hae Ⅲ酶(日本TaKaRa公司)和7.6 μL灭菌双蒸水。放置水浴锅中37℃温浴1h, 1%琼脂糖电泳检验酶切效果。最后将剩余酶切产物进行毛细管电泳并检测荧光和荧光强度(上海生工生物工程有限公司)。
1.5 数据分析
数据取整, 保留50—500 bp内的末端限制性片段数据(T-RFs), 看作是物种种类数。计算同一样品的所有峰度面积之和, 以及各片段面积占所在样品总面积的比例, 看作是物种丰度[21]。
使用R语言(R-3.2.3)程序包Vegan[22]计算多样性指数, 基于Bray-Curtis距离[23]量化样本间群落结构差异, 使用Adonis进行组间差异显著性检验[24], 并进行典范对应分析[25]、蒙特卡罗检验(Monte Carlo permutation test)[26]。多样性指数包括物种数(S), Shannon-Wiener指数(H′)[27]和Pielou指数(J′)[28]。典范对应分析反映物种或样点与环境因子间的关系。蒙特卡罗检验用于检验群落结构与理化因子相关性是否显著(重复数为999)。
参考地表水环境质量标准《GB 3838—2002》, 综合丰水期及枯水期数据, 按水质类别将水质状况分为优(Ⅰ、Ⅱ类水质)、良(Ⅲ类水)、轻度污染(Ⅳ类水质)、中度污染(Ⅴ类水质)及重度污染(劣Ⅴ类水质)。使用R语言indicspecies程序包[29]进行组间指示种分析, 分别筛选了代表优(Ⅰ、Ⅱ类水)及重度污染(劣Ⅴ类水)潜在的敏感种和耐污种的末端限制性片段。并使用MICA工具进行末端限制性片段对应物种预测[30]。使用R语言vegan[22]程序包中的corr.test功能对物种(T-RFs)和理化因子进行Spearman秩相关分析, 并使用corr.test检验其相关性是否显著(重复数为999)。
2. 结果
2.1 真核微型生物多样性
丰水期40个样本中共发现133个不同的末端限制性片段, 代表了至少133个不同的物种。样点末端限制性片段数目最少为2个(GG2_1), 最多为25个(XG5_1); 香农威尔指数最小也为0.6734(GG2_1), 最大为3.0611 (XG5_1); 均匀度指数最小为0.5564 (XS5_1), 最大为0.9715 (GG2_1, 表 1)。由此可见, 珠江广州段样本GG2_1物种组成单一, 且末端限制性片段分布不均匀; 而西江广东段样本XG5_1物种组成较为多样化, 且末端限制性片段分布较为均匀。
表 1 真核微型生物多样性指数Table 1. Diversity index of microbial eukaryotes丰水期
Wet season末端限制性
片段数目S香农威尔指数H′ 均匀度指数J′ 枯水期
Dry season末端限制性
片段数目S香农威尔指数H′ 均匀度指数J′ GC1_1 9 1.608 0.7318 GC1_2 10 2.0652 0.8969 GC2_1 16 2.216 0.7993 GC2_2 13 2.1221 0.8273 GC3_1 9 1.3739 0.6253 GC3_2 15 1.9987 0.738 GC4_1 12 1.8551 0.7465 GC4_2 14 1.5333 0.581 — — — — GC5_2 17 2.4159 0.8527 — — — — GC6_2 20 2.7689 0.9243 — — — — GC7_2 21 2.6763 0.879 GG1_1 23 2.8175 0.8986 GG1_2 13 2.1689 0.8456 GG2_1 2 0.6734 0.9715 GG2_2 17 2.0793 0.7339 GG3_1 9 1.6692 0.7597 GG3_2 10 1.7488 0.7595 GJ1_1 12 2.068 0.8322 GJ1_2 22 2.2546 0.7294 GJ2_1 11 1.4899 0.6213 GJ2_2 18 1.8902 0.654 GJ3_1 10 1.8355 0.7971 GJ3_2 15 2.1987 0.8119 GN1_1 22 2.6622 0.8612 GN1_2 12 1.8717 0.7532 GN2_1 12 1.8629 0.7497 GN2_2 10 1.2377 0.5375 GN3_1 14 2.1524 0.8156 GN3_2 22 2.729 0.8829 GR1_1 16 2.0206 0.7288 GR1_2 12 1.4302 0.5756 GR2_1 12 1.7408 0.7005 GR2_2 18 2.4772 0.8571 GR3_1 12 1.9956 0.8031 GR3_2 12 1.7861 0.7188 GS1_1 9 1.4595 0.6643 GS1_2 16 1.5581 0.562 GS2_1 11 1.7814 0.7429 GS2_2 17 1.7799 0.6282 GS3_1 11 1.4508 0.605 GS3_2 16 2.2194 0.8005 XG1_1 12 2.04 0.821 XG1_2 29 3.1966 0.9493 XG2_1 21 2.7365 0.8988 XG2_2 12 1.9619 0.7895 XG3_1 15 2.1675 0.8004 XG3_2 22 2.7692 0.8959 XG4_1 14 2.055 0.7787 XG4_2 19 2.4304 0.8254 XG5_1 25 3.0611 0.951 XG5_2 15 1.9894 0.7346 XJ1_1 17 2.2633 0.7988 XJ1_2 14 2.2346 0.8467 XJ2_1 19 2.5793 0.876 XJ2_2 11 1.8417 0.768 XJ3_1 18 2.1775 0.7534 XJ3_2 12 1.9432 0.782 XJ4_1 13 1.8523 0.7222 XJ4_2 15 2.2064 0.8148 XJ5_1 12 1.9305 0.7769 XJ5_2 21 2.6664 0.8758 XN1_1 17 2.3056 0.8138 XN1_2 21 2.7569 0.9055 XN2_1 16 2.4022 0.8664 XN2_2 10 1.7982 0.7809 XN3_1 18 2.5455 0.8807 XN3_2 15 2.2528 0.8319 XN4_1 15 2.1885 0.8082 XN4_2 18 2.4169 0.8362 XN5_1 16 2.564 0.9248 XN5_2 13 2.0353 0.7935 XR1_1 17 2.3402 0.826 XR1_2 12 2.0757 0.8353 XS1_1 11 1.9654 0.8196 XS1_2 29 3.1842 0.9456 XS2_1 16 2.3648 0.8529 XS2_2 12 1.9814 0.7974 XS3_1 11 2.1321 0.8892 XS3_2 17 2.1603 0.7625 XS4_1 16 2.1631 0.7802 XS4_2 18 2.3741 0.8214 XS5_1 6 0.997 0.5564 XS5_2 16 2.4252 0.8747 注: S代表末端限制性片段数目, H′代表香农威尔指数, J′代表均匀度指数, —. 丰水期缺失采样点Note: S represents T-RF number, H′ represents Shannon-Wiener index, J′ represents evenness index, —. sampling sites absent in wet season 枯水期43个样本中共发现167个不同的末端限制性片段, 末端限制性片段数目最少为10个(GC1_2、GG3_2、GN2_2、XN2_2), 最多为29个(XG1_2和XS1_2)。珠江广州段样本GN2_2的均匀度指数最小(0.5375), 香农威尔指数也最小(1.2377), 说明该样本物种组成单一, 且T-RFs分布不均匀。西江广东段样本XG1_2的均匀度指数最大(0.9493), 香农威尔指数也最大(3.1966), 其末端限制性片段数目虽多(29), 但各个物种的数量相差不大, 物种组成较为多样化。
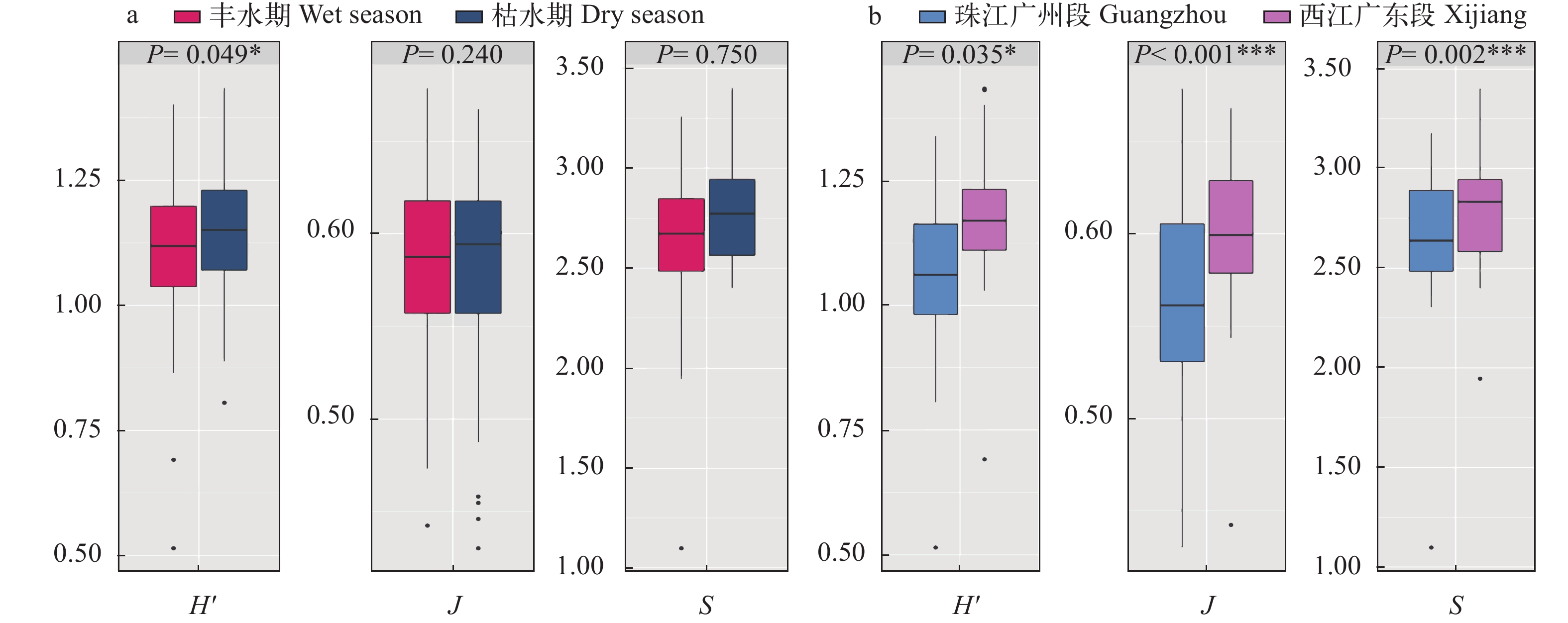
丰水期样本与枯水期样本的末端限制性片段数目与均匀度指数无显著差异, 但是香农威尔指数存在显著差异(P<0.05, 图 2a), 枯水期样本多样性指数普遍高于丰水期样本(表 1)。珠江广州段样本与西江广东段样本多样性指数均存在显著差异(P<0.05, 图 2b), 西江广东段样本多样性指数普遍高于珠江广州段样本(表 1)。
![]() 图 2 不同水期(a)和采样区域(b)多样性指数箱线图横轴表示多样性指数, 纵轴代表lg (X+1) 转化后的多样性指数值, P代表检验值, * 表示显著性水平P<0.05Figure 2. The boxplots for diversity index of different water seasons or regionsThe horizontal axis represents diversity indexes, and vertical axis represents the values of diversity index transformed by lg (X+1), P represents test value, * indicates P<0.05
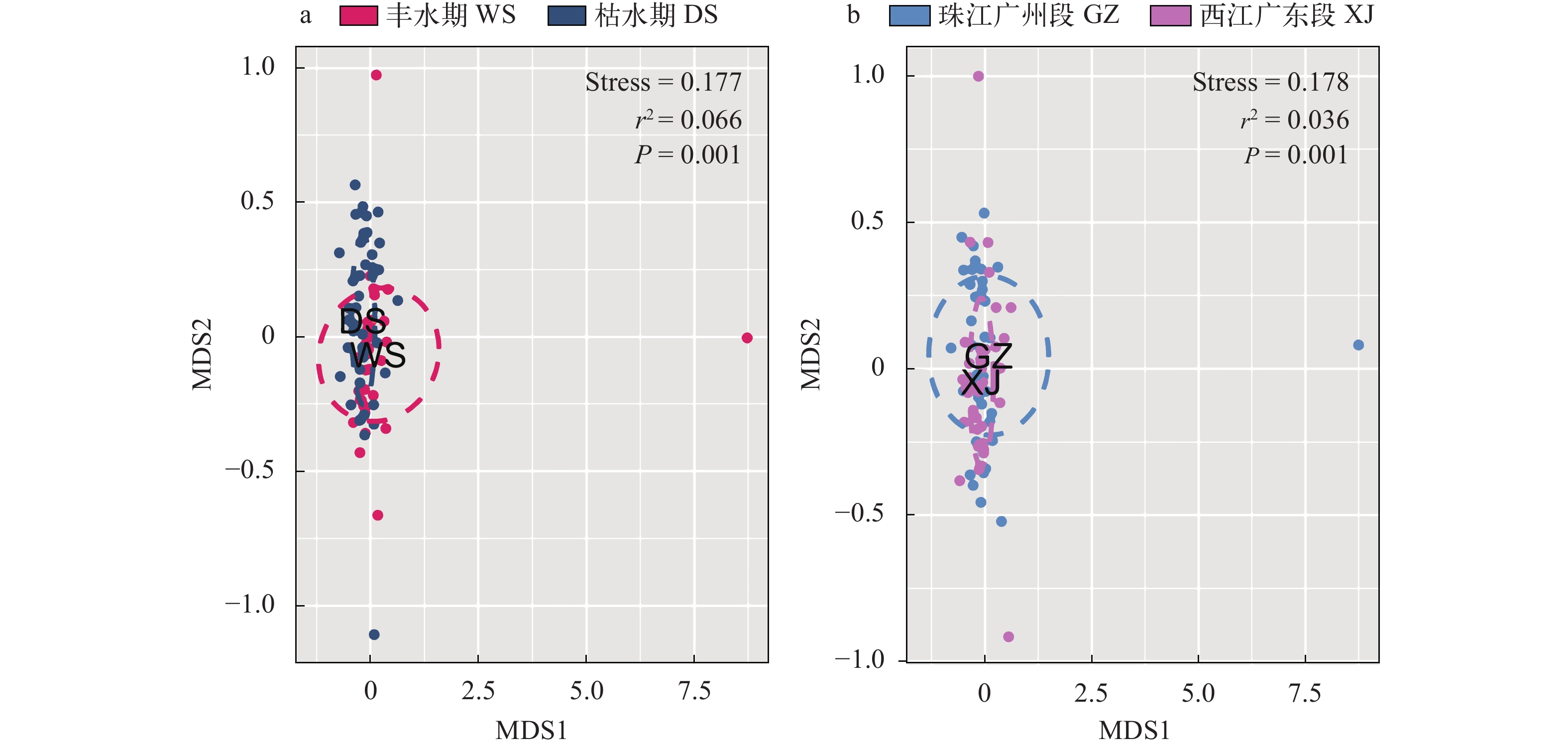
图 2 不同水期(a)和采样区域(b)多样性指数箱线图横轴表示多样性指数, 纵轴代表lg (X+1) 转化后的多样性指数值, P代表检验值, * 表示显著性水平P<0.05Figure 2. The boxplots for diversity index of different water seasons or regionsThe horizontal axis represents diversity indexes, and vertical axis represents the values of diversity index transformed by lg (X+1), P represents test value, * indicates P<0.05真核微型生物非度量多维尺度分析(图 3)显示, 丰水期40个样本(WS)较枯水期43个样本(DS)的分布更为分散, 珠江广州段41个样本(GZ)较西江广东段42个样本(XJ)的分布更为分散。Adonis分析表明丰水期与枯水期样本之间、珠江广州段与西江广东段样本之间的真核微型生物群落结构均存在显著差异(P<0.05, 图 3)。
![]() 图 3 不同水期(a)和不同区域(b)真核微型生物非度量多维尺度分析WS代表丰水期, DS代表枯水期, GZ代表珠江广州段, XJ代表西江广东段Figure 3. The nMDS for microbial eukaryotes of different water seasons (a) and regions (b)WS represents wet season, DS represents dry season, GZ represents Guangzhou Reach of the Pearl River, XJ represents Guangdong Reach of the Xijiang River
图 3 不同水期(a)和不同区域(b)真核微型生物非度量多维尺度分析WS代表丰水期, DS代表枯水期, GZ代表珠江广州段, XJ代表西江广东段Figure 3. The nMDS for microbial eukaryotes of different water seasons (a) and regions (b)WS represents wet season, DS represents dry season, GZ represents Guangzhou Reach of the Pearl River, XJ represents Guangdong Reach of the Xijiang River2.2 真核微型生物群落结构及其与理化因子的关系
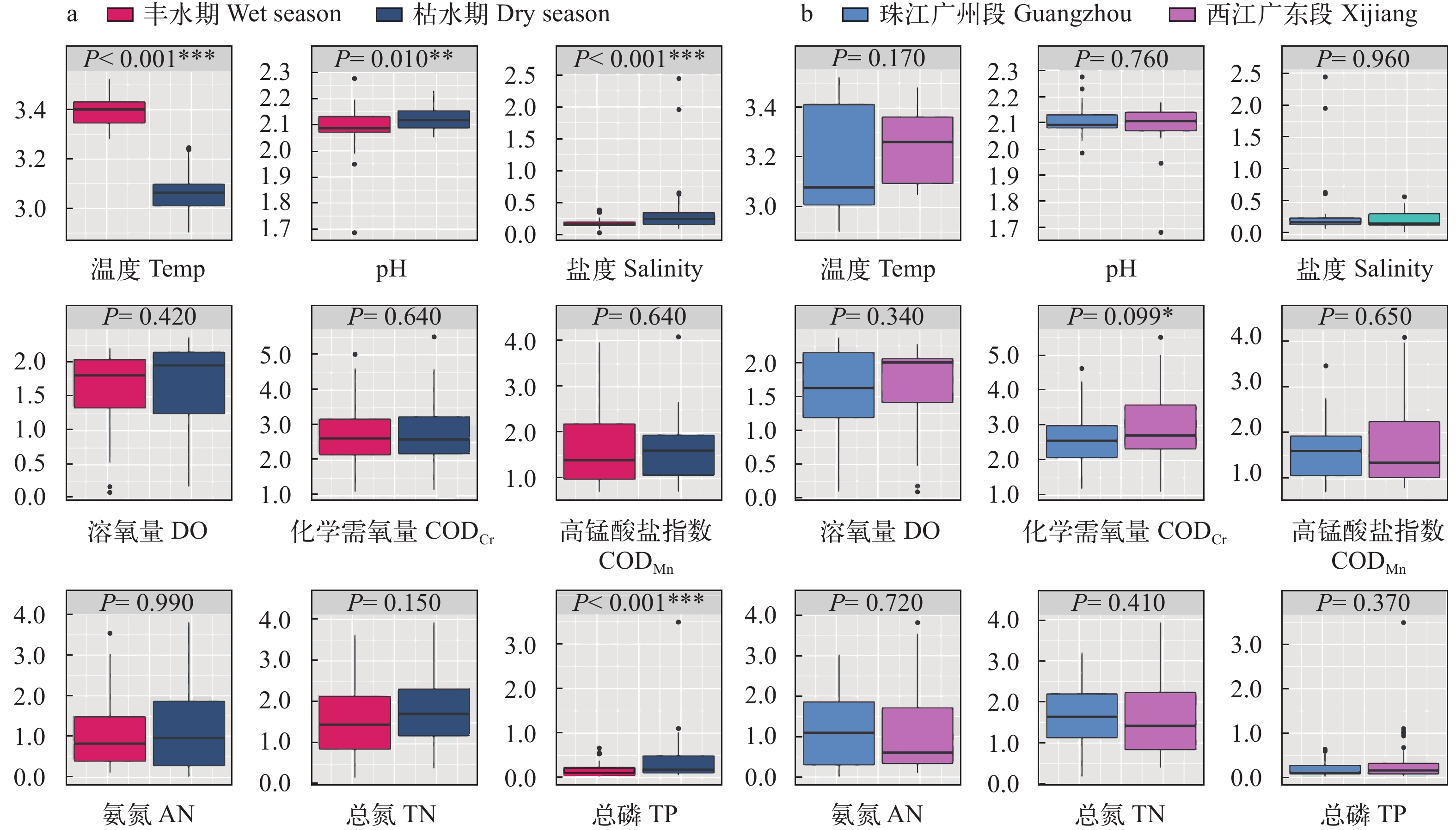
调查区域83个样本中, 水质类别为优、良、轻度污染、中度污染和重度污染的样本分别为3、7、9、7和57个。57个样本的总氮(TN)、氨氮(AN)和总磷(TP)指标均超过地表水劣Ⅴ类水标准, 为珠江水体主要污染指标。枯水期样本温度、pH、盐度和总磷显著高于丰水期样本(P<0.05, 图 4a), 其余所测理化因子在枯水期和丰水期无显著差异。西江广东段样本化学需氧量显著高于珠江广州段样本(P<0.05, 图 4b), 其余所测理化因子在西江广东段和珠江广州段均无显著差异。
![]() 图 4 不同水期(a)和不同区域(b)的理化因子箱线图。横轴代表理化因子, 纵轴代表lg (X+1)转化后的理化因子值, P代表检验值, * 表示显著性水平P<0.05Figure 4. The boxplots for physicochemical factors of different water seasons (a) and regions (b)The horizontal axis represents physicochemical factors, and vertical axis represents the values of physicochemical factors transformed by lg (X+1), P represents test value. * indicates P<0.05
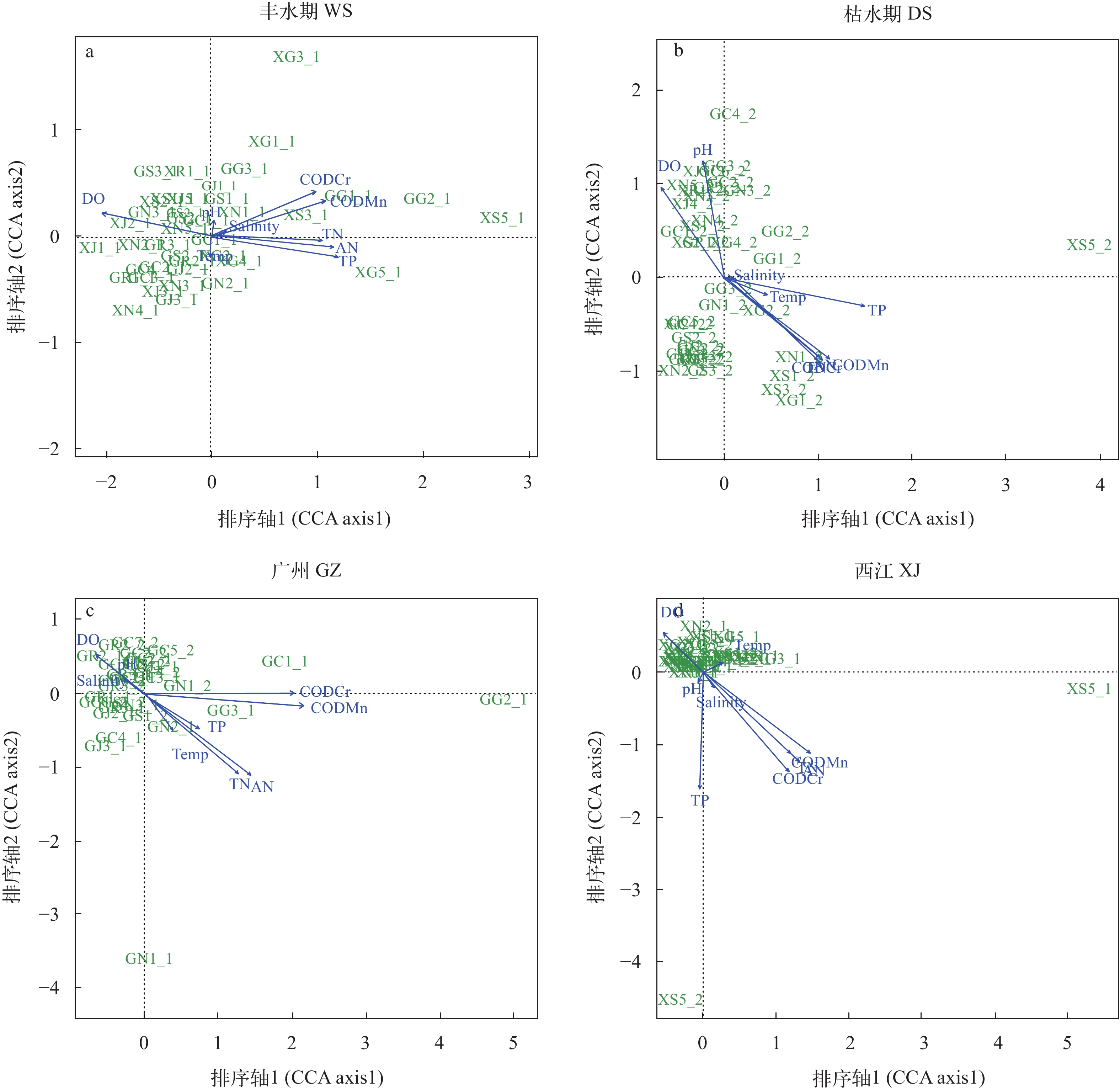
图 4 不同水期(a)和不同区域(b)的理化因子箱线图。横轴代表理化因子, 纵轴代表lg (X+1)转化后的理化因子值, P代表检验值, * 表示显著性水平P<0.05Figure 4. The boxplots for physicochemical factors of different water seasons (a) and regions (b)The horizontal axis represents physicochemical factors, and vertical axis represents the values of physicochemical factors transformed by lg (X+1), P represents test value. * indicates P<0.05丰水期样本典范对应分析(图 5a)和相关性检验(表 2)表明, 丰水期样本真核微型生物群落结构与溶氧量(DO)、化学需氧量(CODCr)、高锰酸盐指数(CODMn)、氨氮(AN)、总氮(TN)和总磷(TP)显著相关(P<0.05), 其中对群落结构影响最大的理化因子是总磷(TP), 相关系数(r2)为0.7885, 其次是氨氮(AN), 相关系数为0.6976。其余理化因子相关系数由大到小分别是高锰酸盐指数(CODMn, 0.6797)、化学需氧量(CODCr, 0.6170)、溶氧量(DO, 0.5599)、总氮(TN, 0.5397)。枯水期样本典范对应分析(图 5b)和相关性检验(表 2)表明, 枯水期样本真核微型生物群落结构受温度(Temp)、化学需氧量(CODCr)、高锰酸盐指数(CODMn)、氨氮(AN)、总氮(TN)和总磷(TP)的显著影响, 其中受总磷(TP)和高锰酸盐指数(CODMn)影响最大, 相关系数分别是0.9289和0.7950, 其次是化学需氧量(CODCr, 0.6815)、总氮(TN, 0.6638)、氨氮(AN, 0.6352)、溶氧量(DO, 0.4449)和温度(TP, 0.1695)。与丰水期相比, 枯水期样本真核微生物群落结构与造成污染的理化因子如总磷(TP)、总氮(TN)和化学需氧量(CODCr)等的相关系数要大于丰水期, 且温度(Temp)对枯水期样本真核微型生物群落结构有显著影响。
表 2 真核微型生物群落结构与理化因子相关性检验Table 2. Correlation test between the microbial eukaryotic community structures of samples and physicochemical factors指标Index 丰水期Wet season 枯水期Dry season 珠江广州段Guangzhou 西江广东段Xijiang 温度Temp 0.0021 0.1695* 0.1329* 0.0351 pH 0.0001 0.7469 0.0061 0.0089 盐度Salinity 0.0026 0.0005*** 0.0026 0.0576 溶氧DO 0.5599*** 0.4449*** 0.1294* 0.2305*** 化学需氧量CODCr 0.6170*** 0.6815*** 0.7687*** 0.8200*** 高锰酸盐指数CODMn 0.6797*** 0.7950*** 0.8313*** 0.8722*** 氨氮AN 0.6976*** 0.6352*** 0.6086*** 0.8681*** 总氮TN 0.5397*** 0.6638*** 0.4973*** 0.8047*** 总磷TP 0.7885*** 0.9289*** 0.1925* 0.9285* 注: 数字代表相关系数, * 表示显著性水平P<0.05Note: Number represents correlation coefficient, * indicates P<0.05 ![]() 图 5 丰水期样本(a)、枯水期样本(b)、珠江广州段(c)和西江广东段(d)样本真核微型群落结构与理化因子典范对应分析绿色代表样本, 蓝线代表理化因子, CCA axis 1代表排序轴1, CCA axis 2代表排序轴2。Temp代表温度, DO代表溶氧量, pH代表酸碱度, CODCr代表化学需氧量, CODMn代表高锰酸盐指数, AN代表氨氮, TN代表总氮, TP代表总磷Figure 5. Canonical correspondence analysis (CCA) between physicochemical factors and microbial eukaryotic community structures of wet season (a), dry season (b), Guangzhou Reach of the Pearl River (c) and Guangdong Reach of the Xijiang River (d)Samples are in green, physicochemical factors are represented by blue lines, the first axis is represented by CCA axis 1, the second axis is represented by CCA axis 2. Temp represents temperature, DO represents dissolved oxygen, pH represents acidity and alkalinity, CODCr represents chemical oxygen demand, CODMn represents permanganate index, AN represents ammonia nitrogen, TN represents total nitrogen, TP represents total phosphorus
图 5 丰水期样本(a)、枯水期样本(b)、珠江广州段(c)和西江广东段(d)样本真核微型群落结构与理化因子典范对应分析绿色代表样本, 蓝线代表理化因子, CCA axis 1代表排序轴1, CCA axis 2代表排序轴2。Temp代表温度, DO代表溶氧量, pH代表酸碱度, CODCr代表化学需氧量, CODMn代表高锰酸盐指数, AN代表氨氮, TN代表总氮, TP代表总磷Figure 5. Canonical correspondence analysis (CCA) between physicochemical factors and microbial eukaryotic community structures of wet season (a), dry season (b), Guangzhou Reach of the Pearl River (c) and Guangdong Reach of the Xijiang River (d)Samples are in green, physicochemical factors are represented by blue lines, the first axis is represented by CCA axis 1, the second axis is represented by CCA axis 2. Temp represents temperature, DO represents dissolved oxygen, pH represents acidity and alkalinity, CODCr represents chemical oxygen demand, CODMn represents permanganate index, AN represents ammonia nitrogen, TN represents total nitrogen, TP represents total phosphorus珠江广州段样本典范对应分析(图 5c)和相关性检验(表 2)表明, 珠江广州段样本真核微型生物群落结构与温度(Temp)、溶氧量(DO)、化学需氧量(CODCr)、高锰酸盐指数(CODMn)、氨氮(AN)、总氮(TN)和总磷(TP)显著相关(P <0.05), 其中对群落结构影响最大的理化因子是高锰酸盐指数(CODMn), 相关系数(r2)为0.8313, 其次是化学需氧量(CODCr), 相关系数为0.7687。其余理化因子相关系数由大到小分别是氨氮(AN, 0.6086)、总氮(TN, 0.4973)、总磷(TP, 0.1925)、温度(Temp, 0.1329)、溶氧量(DO, 0.1294)。西江广东段样本典范对应分析(图 5d)和相关性检验(表 2)表明, 西江广东段样本真核微型生物群落结构与溶氧量(DO)、化学需氧量(CODCr)、高锰酸盐指数(CODMn)、氨氮(AN)、总氮(TN)和总磷(TP)显著相关(P <0.05), 其中对群落结构影响最大的理化因子是总磷(TP), 相关系数(r2)为0.9285, 其次是高锰酸盐指数(CODMn), 相关系数为0.8722。其余理化因子相关系数由大到小分别是氨氮(AN, 0.8681)、化学需氧量(CODCr, 0.8200)、总氮(TN, 0.8047)、溶氧量(DO, 0.2305)。与珠江广州段样本相比, 西江广东段样本真核微型生物群落结构与理化因子相关系数普遍更大, 但是温度对西江广东段样本真核微型生物群落结构无显著影响(P>0.05)。
2.3 潜在指示物种筛选
为筛选潜在的对应水质指示物种, 参考地表水环境质量标准《GB 3838—2002》, 在水质类为优(I、II类水)、良(III类水)、轻度污染(IV类水质)、中度污染(V类水质)、重度污染(劣V类水)的样本组间进行组间指示末端限制性片段筛选(表 3)。筛选出1个末端限制性片段, 片段长度为76 bp, 可能只存在于“优”级水质, 而在水质为其余类别的水体中基本不存在或数量非常少。筛选出了8个末端限制性片段, 只存在或大量存在于“重度污染”水体中, 片段长度分别为83、117、192、235、376、377、391和406 bp。基于数据库比对, 潜在的敏感物种可能为后鞭类(Opisthokonta)的Cystobasidium sp. 或变形类(Amoebozoa)的Protostelium nocturnum; 潜在的耐污物种可能为后鞭类(Opisthokonta)的Candida caryicola、梅奇酵母科(Metschnikowiaceae)或酿酒酵母目的未定科(Saccharomycetales incertae sedis)物种; 原始色素体生物(Archaeplastida)的Coccomyxa simplex、Botryosphaerella sudetica以及轮藻科(Characeae)、衣藻科(Chlamydomonadaceae)物种; 变形类(Amoebozoa)的Acanthamoeba hatchetti; 囊泡类(Alveolata)的Sulfonecta uniserialis、Stenophora robusta、Theileria sp.、Babesia bicornis以及钟形虫科(Vorticellidae)和隐孢科(Cryptosporidiidae)物种; 隐藻类(Cryptophyceae)的Cryptomonas ovata; 不等鞭毛类(Stramenopiles)的Filos agilis和Blastocystis sp.。
表 3 潜在的水质指示片段Table 3. Potential indicative T-RFs片段长度The length of T-RF A B P 优 76 0.9465 0.2500 0.009** 重度污染 83 0.9851 0.2222 0.033* 117 0.9934 0.2222 0.028* 192 1.0000 0.2222 0.032* 235 0.8198 0.2222 0.045* 376 1.0000 0.2222 0.027* 377 1.0000 0.2222 0.027* 391 1.0000 0.2222 0.035* 406 0.7552 0.3333 0.022* 注: A代表该片段仅在水质类别为优/重度污染的样本组间存在的概率, B代表该片段在水质类别为优/重度污染的样本组内存在的概率。P代表检验值, * 表示显著性水平P<0.05Note: A represents the probability that the T-RF were only present in high water quality samples/poor quality samples, B represents the probability that the T-RFs were detected among all high water quality samples/poor quality samples. P represents test value. * indicates P<0.05 附表 3 基于MICA工具的末端限制性片段对应物种预测 Appendix Tab. 3 Species prediction based on terminal restriction fragments and MICA tool 片段长度The length of T-RF GenBank登录号Accession number 物种分类Taxonomy 76 AB055190 Opisthokonta, Fungi, Dikarya, Basidiomycota, Pucciniomycotina, Cystobasidiomycetes, Cystobasidiales, Cystobasidiaceae, Cystobasidium, Cystobasidium sp. 76 FJ766481 Amoebozoa, Mycetozoa, Protosteliales, Protosteliaceae, Protostelium, Protostelium nocturnum 83 DQ504334 Alveolata, environmental samples 83 FJ648512 Archaeplastida, Viridiplantae, Chlorophyta, Trebouxiophyceae, Trebouxiophyceae incertae sedis, Coccomyxaceae, Coccomyxa, Coccomyxa simplex 83 EU446335 environmental samples 83 AB023471 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina,Saccharomycetes, Saccharomycetales, Saccharomycetales incertae sedis, Candida, Candida agrestis 83 AB013503 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Saccharomycetales incertaesedis, Candida melibiosica 83 AY242231 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Saccharomycetales incertaesedis, Candida, Candida sp. 83 AB013514 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 AY640202 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 DQ655699 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 AB013571 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 EF152415 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 EF152412 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 EF408189 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 X89518 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 AB013545 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 AB013547 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 AY611608 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Candida, Candida sp. 83 AY520164 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Metschnikowia, Metschnikowia chrysomelidarum 83 AY452053 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Metschnikowia, Metschnikowia picachoensis 83 AY452055 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Metschnikowia, Metschnikowia pimensis 83 AJ508272 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Metschnikowia, Metschnikowia rancensis 117 U57695 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chlamydomonas, Chlamydomonas mutabilis 117 U57696 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chlamydomonas, Chlamydomonas nivalis 117 DQ009749 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Microglena, Microglena reginae 117 AF517100 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chlamydomonas, unclassified Chlamydomonas 117 AJ410445 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chloromonas, Chloromonas actinochloris 117 U70783 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chloromonas, Chloromonas asteroidea 117 AF517095 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chloromonas, Chloromonas augustae 117 AF517092 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chloromonas, Chloromonas brevispina 117 GU117585 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chloromonas, Chloromonas insignis 117 AF514402 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chloromonas, Chloromonas rostafinskii 117 AF514401 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chloromonas, Chloromonas cf. platystigma 117 GU117584 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chloromonas, Chloromonas cf. schussnigii 3. 讨论
本研究T-RFLP数据表明, 丰水期与枯水期水体中的真核微型生物香农威尔指数和群落结构存在显著差异(图 2a、图 3a)。前人研究也表明季节是影响真核微型生物群落组成的重要因素, 例如: Countway等[31]报道同一样点不同季节的真核微型生物物种组成有显著差异; 北太平洋东部海域的真核微型生物群落存在显著的季节和年度差异[32]。与此同时, 本研究发现珠江广州段和西江广东段真核微型生物多样性指数、群落结构也存在显著差异(图 2b、图 3b), 表明地域甚至是小范围的地理位置差异对真核微型生物多样性有着显著的影响。同样, 先前文献报道, 青岛、厦门和湛江沿岸滩涂真核微型生物群落结构存在显著差异[33]; 美国黄石公园不同样点间真核微型生物群落相似性较低[34]。考虑到时间和地域的差异对物种多样性有显著的影响, 我们建议在利用真核微型生物监测水质时, 必须建立在全面了解该地区不同时期、不同地点的真核微型生物多样性的基础上。
我们的研究数据表明, 珠江广州段和西江广东段水体受到氮、磷的严重污染, 普遍处于劣V类(57个样本, 占据了69%); 丰水期总磷浓度普遍低于枯水期, 西江广东段化学需氧量浓度普遍高于珠江广州段(图 4)。排序分析和相关性检验(图 5、表 2)表明, 真核微型生物群落结构与化学需氧量、高锰酸盐指数、氨氮、总氮、总磷等水质监测相关理化因子显著相关, 能够较好地反映水质状况。化学需氧量和高锰酸盐指数代表着样点受有机物污染的程度[35], 其对真核微型生物群落结构有着显著的影响[36, 37]。过量的氨氮会影响细胞膜稳定性及酶水解反应, 导致生物代谢缓慢, 严重时可导致生物死亡[38], 文献报道原生生物、金藻、硅藻与氨氮之间呈现显著正相关[12]。总氮和总磷通过影响浮游藻类的光合作用对其生长进行调控, 从而又影响了异养真核微型生物类群[39—43]。总氮和总磷是导致水体富营养化的主要理化因子[44], 此类营养盐主要来源于工业废水和生活污水, 而西江广东段与珠江广州段周边工业废水和生活污水排放量巨大[15]。此外, 本研究发现, 研究区域总氮和总磷的季节性变化不明显, 但枯水期高于丰水期, 这可能是由于丰水期水量大, 物理稀释作用导致总氮和总磷浓度降低, 也可能是因为丰水期水生生物活动频繁, 大量消耗了水体中的营养盐[35]。
某些真核微型生物, 会出现或大量存在于在不同污染程度的水域中, 从而可以作为水质评判的指示物种。许恒龙等[12]发现Peridinium(多甲藻虫属)的某些物种在较为清洁的水体中丰度较高, 可作为潜在的敏感类群。杨金鹏等[45]发现Uronemella (尾丝虫)在富营养水体中有较高的丰度, 可作为潜在的耐污类群。本研究分别筛选出1个、8个末端限制性片段作为候选的敏感和耐污片段, 并根据数据库给出了大致的物种/类群信息, 但由于缺乏具体的碱基信息, 这些片段具体代表哪些物种还有待进一步研究。
致谢:
感谢华南师范大学本科生张宝丹、陈静慧、陈俊宇在部分样品采集和实验操作中给予的帮助。
-
图 2 不同水期(a)和采样区域(b)多样性指数箱线图
横轴表示多样性指数, 纵轴代表lg (X+1) 转化后的多样性指数值, P代表检验值, * 表示显著性水平P<0.05
Figure 2. The boxplots for diversity index of different water seasons or regions
The horizontal axis represents diversity indexes, and vertical axis represents the values of diversity index transformed by lg (X+1), P represents test value, * indicates P<0.05
图 3 不同水期(a)和不同区域(b)真核微型生物非度量多维尺度分析
WS代表丰水期, DS代表枯水期, GZ代表珠江广州段, XJ代表西江广东段
Figure 3. The nMDS for microbial eukaryotes of different water seasons (a) and regions (b)
WS represents wet season, DS represents dry season, GZ represents Guangzhou Reach of the Pearl River, XJ represents Guangdong Reach of the Xijiang River
图 4 不同水期(a)和不同区域(b)的理化因子箱线图。
横轴代表理化因子, 纵轴代表lg (X+1)转化后的理化因子值, P代表检验值, * 表示显著性水平P<0.05
Figure 4. The boxplots for physicochemical factors of different water seasons (a) and regions (b)
The horizontal axis represents physicochemical factors, and vertical axis represents the values of physicochemical factors transformed by lg (X+1), P represents test value. * indicates P<0.05
图 5 丰水期样本(a)、枯水期样本(b)、珠江广州段(c)和西江广东段(d)样本真核微型群落结构与理化因子典范对应分析
绿色代表样本, 蓝线代表理化因子, CCA axis 1代表排序轴1, CCA axis 2代表排序轴2。Temp代表温度, DO代表溶氧量, pH代表酸碱度, CODCr代表化学需氧量, CODMn代表高锰酸盐指数, AN代表氨氮, TN代表总氮, TP代表总磷
Figure 5. Canonical correspondence analysis (CCA) between physicochemical factors and microbial eukaryotic community structures of wet season (a), dry season (b), Guangzhou Reach of the Pearl River (c) and Guangdong Reach of the Xijiang River (d)
Samples are in green, physicochemical factors are represented by blue lines, the first axis is represented by CCA axis 1, the second axis is represented by CCA axis 2. Temp represents temperature, DO represents dissolved oxygen, pH represents acidity and alkalinity, CODCr represents chemical oxygen demand, CODMn represents permanganate index, AN represents ammonia nitrogen, TN represents total nitrogen, TP represents total phosphorus
附表 1 采样点信息 Appendix Tab. 1 Information of sampling sites 样品名Sample name 经度Longitude 纬度Latitude 样本描述Sample description 地点描述Location description GC1 113°47′37.211′′ 23°45′8.2548′′ 广州参照点1 流溪河国家森林公园 GC2 113°5′27.650′′ 23°9′2.988′′ 广州参照点2 思贤窖 GC3 112°53′8.001′′ 23°9′39.5604′′ 广州参照点3 三水区东平水道 GC4 113°20′45.229′′ 23°4′56.1755′′ 广州参照点4 海珠湿地公园 GC5 113°17′44.88″ 22°53′35.6855′′ 广州参照点5 顺德水道 GC6 113°37′26.4936′′ 23°6′44.5932′′ 广州参照点6 刘屋洲水道 GC7 113°11′46.7088′′ 23°26′20.0543′′ 广州参照点7 秀全水库 GG1 113°13′23.4875′′ 23°12′1.7748′′ 广州工业点1 石门水厂工业区 GG2 113°13′55.9344′′ 23°9′51.6455′′ 广州工业点2 松北工业区 GG3 113°18′13.2228′′ 23°3′45.2556″ 广州工业点3 南华西工业区 GJ1 113°14′42.0972″ 23°6′31.6691′′ 广州交通点1 芳村码头 GJ2 113°18′22.4604″ 23°6′35.3339′′ 广州交通点2 中大码头 GJ3 113°26′17.1095′′ 23°5′59.8524′′ 广州交通点3 嘉利码头 GN1 113°18′58.5503′′ 23°5′2.85′′ 广州农田点1 上冲果树公园 GN2 113°28′4.1016″ 23°7′9.966″ 广州农田点2 大沙北 GN3 113°29′8.7756″ 22°53′23.838″ 广州农田点3 东涌 GR1 113°37′48.2051′′ 22°45′34.9128″ 广州入海口1 南沙天后宫 GR2 113°29′30.4224′′ 23°3′55.134′′ 广州入海口2 黄埔大桥 GR3 113°40′18.1272″ 22°38′29.1623′′ 广州入海口3 新龙特大桥 GS1 113°13′52.1508″ 23°7′42.4739′′ 广州生活点1 珠岛花园 GS2 113°15′28.0835′′ 23°5′48.5411′′ 广州生活点2 太古仓 GS3 113°20′13.4484′′ 23°7′4.5552′′ 广州生活点3 猎德涌 XG1 112°48′9.0108″ 23°5′34.0475′′ 西江工业点1 肇庆市高要区 XG2 112°56′33.0539′′ 22°55′15.6792″ 西江工业点2 佛山市高明区 XG3 112°55′54.0156″ 22°44′4.4592″ 西江工业点3 江门市鹤山市 XG4 113°7′31.5372′′ 22°33′17.0352″ 西江工业点4 江门市江海区 XG5 113°30′21.0923′′ 22°16′44.4575′′ 西江工业点5 中山市坦洲镇 XJ1 112°48′17.7551′′ 23°5′58.2792′′ 西江交通点1 肇庆市高要区 XJ2 112°54′26.3556′′ 22°54′0.864″ 西江交通点2 佛山市高明区 XJ3 113°1′57.5436″ 22°48′23.2271′′ 西江交通点3 江门市鹤山市 XJ4 113°11′6.8459′′ 22°35′9.3227′′ 西江交通点4 江门市江海区 XJ5 113°25′28.9488′′ 22°13′8.1012′′ 西江交通点5 中山市坦洲镇 XN1 112°47′54.4019′′ 23°5′24.6911′′ 西江农田点1 肇庆市高要区 XN2 112°51′48.8592′′ 22°57′21.5136′′ 西江农田点2 佛山市高明区 XN3 113°4′52.176′′ 22°41′48.8112′′ 西江农田点3 江门市鹤山市 XN4 113°5′25.1592′′ 22°32′52.908′′ 西江农田点4 江门市江海区 XN5 113°24′40.5684′′ 22°13′34.6008′′ 西江农田点5 中山市坦洲镇 XR1 113°25′14.4587′′ 22°12′55.5731′′ 西江入海口 中山市坦洲镇 XS1 112°47′47.5008′′ 23°5′22.1316′′ 西江生活点1 肇庆市高要区 XS2 112°54′22.7664″ 22°54′5.4756″ 西江生活点2 佛山市高明区 XS3 112°57′38.0952′′ 22°46′33.6″ 西江生活点3 江门市鹤山市 XS4 113°5′27.6504′′ 22°32′26.9843′′ 西江生活点4 江门市江海区 XS5 113°31′16.4387′′ 22°16′25.4568′′ 西江生活点5 中山市坦洲镇 附表 2 理化因子数据 Appendix Tab. 2 Physicochemical factors 样本Sample 温度Temp (℃) pH 盐度Salinity (‰) 溶氧量DO (mg/L) 化学需氧量CODCr (mg/L) 高锰酸盐指数CODMn (mg/L) 氨氮AN (mg/L) 总氮TN (mg/L) 总磷TP (mg/L) 水质类别The rank of water quality GC1_1 27.4 7.82 0.088 8.2 8 1.46 0.12 0.2 0.037 Ⅰ、Ⅱ类 GC2_1 26.2 6.87 0.13 7.9 5 1.03 0.24 1.4 0.046 Ⅳ类 GC3_1 26.7 6.97 0.134 7.7 7 1.31 0.28 1.3 0.062 Ⅳ类 GC4_1 33 8.75 0.196 7.8 8 1.25 0.11 0.5 0.02 Ⅰ、Ⅱ类 GG1_1 27.9 7.07 0.232 1.2 70 15 9.9 12.2 0.435 劣Ⅴ类 GG2_1 27.8 7.13 0.265 0.2 101 30.9 18.5 20.4 0.705 劣Ⅴ类 GG3_1 31.1 7.44 0.191 5.4 23 8.46 3.4 5.1 0.186 劣Ⅴ类 GJ1_1 28.5 7.07 0.17 2.7 18 5.08 2.36 3.5 0.104 劣Ⅴ类 GJ2_1 30.5 7.06 0.167 3 8 3.3 2.6 3.6 0.092 劣Ⅴ类 GJ3_1 29.9 7.02 0.177 3.6 7 2.58 2.7 5.6 0.106 劣Ⅴ类 GN1_1 29.9 7 0.265 0.1 17 7.92 19.8 23.8 0.745 劣Ⅴ类 GN2_1 29.4 6.64 0.292 4.4 23 5.32 5.6 7.4 0.178 劣Ⅴ类 GN3_1 32 7.1 0.208 3.6 12 4.58 1.76 3.6 0.104 劣Ⅴ类 GR1_1 28.8 7.29 0.171 4.9 3.5 1.68 0.56 2.1 0.076 劣Ⅴ类 GR2_1 30.3 7.04 0.092 2.7 14 1.7 0.56 1.8 0.064 Ⅴ类 GR3_1 32.9 8 0.207 7 16 1.92 0.6 2 0.044 Ⅴ类 GS1_1 30.7 6.95 0.177 0.7 18 6.2 3.1 4.2 0.116 劣Ⅴ类 GS2_1 28.9 7.19 0.147 4.1 5 2.66 1.3 1.6 0.064 Ⅴ类 GS3_1 30 6.3 0.189 0.9 11 6.18 2 5.3 0.114 劣Ⅴ类 XG1_1 29.1 6.88 0.31 3.1 38 9.68 2.7 8 0.272 劣Ⅴ类 XG2_1 31.6 7.45 0.283 5.6 18 6.34 4.7 6.8 0.22 劣Ⅴ类 XG3_1 28 7.22 0.173 3.2 42 19.4 4 8.2 0.256 劣Ⅴ类 XG4_1 26.1 7.01 0.174 6.5 10 2.8 0.2 1.6 0.269 Ⅴ类 XG5_1 27.6 6.93 0.031 1.8 25 10.44 8.7 10 0.49 劣Ⅴ类 XJ1_1 26.9 7.6 0.206 7.6 4 1.3 0.21 3.6 0.086 劣Ⅴ类 XJ2_1 29.4 7.54 0.146 6.6 4.5 2.06 0.49 1.2 0.027 Ⅳ类 XJ3_1 27.3 7.51 0.155 6.8 2.8 1.38 0.5 0.6 0.037 Ⅲ类 XJ4_1 25.6 7.28 0.16 7.4 3.3 1.42 0.87 1.7 0.042 Ⅴ类 XJ5_1 27 7.03 0.152 6.5 16 2.31 0.48 1.1 0.042 Ⅳ类 XN1_1 29.5 6.87 0.418 2.8 32 9.16 3.5 8.6 0.342 劣Ⅴ类 XN2_1 30 4.4 0.175 6.88 14 4.08 1.02 3.2 0.082 劣Ⅴ类 XN3_1 27.6 6.02 0.191 7.7 12 1.76 0.4 0.8 0.056 Ⅲ类 XN4_1 26.8 7.42 0.172 5.6 2 1.5 1.3 2.7 0.061 劣Ⅴ类 XN5_1 29.4 6.94 0.252 6.6 14 2.93 0.52 1.4 0.09 Ⅳ类 XR1_1 26.5 6.7 0.151 6.5 12 3.26 0.42 1.4 0.078 Ⅳ类 XS1_1 28.9 6.81 0.473 3.2 25 8.72 2.2 8 0.304 劣Ⅴ类 XS2_1 29.2 7.52 0.144 6.5 10.6 2.42 0.61 1.4 0.035 Ⅳ类 XS3_1 29.8 7.04 0.219 2.8 36 8.06 7 9.6 0.262 劣Ⅴ类 XS4_1 26.2 7.56 0.163 6.7 9 1.66 0.1 0.8 0.03 Ⅲ类 XS5_1 29.6 7.13 0.217 0.1 150 52.2 33.5 37 0.95 劣Ⅴ类 GC1_2 20.8 7.23 0.083 7.6 3 1.04 0.06 2.3 0.055 劣Ⅴ类 GC2_2 18.8 7.71 0.155 8.4 8 2.06 0.32 2.2 0.104 劣Ⅴ类 GC3_2 18.6 7.69 0.152 8.1 5 1.52 0.43 2.4 0.098 劣Ⅴ类 GC4_2 19.1 8.31 0.214 9.8 3 1.74 0.01 0.5 0.055 Ⅰ、Ⅱ类 GC5_2 20.2 7.31 0.31 6.1 11.5 4.68 1.62 3.1 0.137 劣Ⅴ类 表 1 真核微型生物多样性指数
Table 1 Diversity index of microbial eukaryotes
丰水期
Wet season末端限制性
片段数目S香农威尔指数H′ 均匀度指数J′ 枯水期
Dry season末端限制性
片段数目S香农威尔指数H′ 均匀度指数J′ GC1_1 9 1.608 0.7318 GC1_2 10 2.0652 0.8969 GC2_1 16 2.216 0.7993 GC2_2 13 2.1221 0.8273 GC3_1 9 1.3739 0.6253 GC3_2 15 1.9987 0.738 GC4_1 12 1.8551 0.7465 GC4_2 14 1.5333 0.581 — — — — GC5_2 17 2.4159 0.8527 — — — — GC6_2 20 2.7689 0.9243 — — — — GC7_2 21 2.6763 0.879 GG1_1 23 2.8175 0.8986 GG1_2 13 2.1689 0.8456 GG2_1 2 0.6734 0.9715 GG2_2 17 2.0793 0.7339 GG3_1 9 1.6692 0.7597 GG3_2 10 1.7488 0.7595 GJ1_1 12 2.068 0.8322 GJ1_2 22 2.2546 0.7294 GJ2_1 11 1.4899 0.6213 GJ2_2 18 1.8902 0.654 GJ3_1 10 1.8355 0.7971 GJ3_2 15 2.1987 0.8119 GN1_1 22 2.6622 0.8612 GN1_2 12 1.8717 0.7532 GN2_1 12 1.8629 0.7497 GN2_2 10 1.2377 0.5375 GN3_1 14 2.1524 0.8156 GN3_2 22 2.729 0.8829 GR1_1 16 2.0206 0.7288 GR1_2 12 1.4302 0.5756 GR2_1 12 1.7408 0.7005 GR2_2 18 2.4772 0.8571 GR3_1 12 1.9956 0.8031 GR3_2 12 1.7861 0.7188 GS1_1 9 1.4595 0.6643 GS1_2 16 1.5581 0.562 GS2_1 11 1.7814 0.7429 GS2_2 17 1.7799 0.6282 GS3_1 11 1.4508 0.605 GS3_2 16 2.2194 0.8005 XG1_1 12 2.04 0.821 XG1_2 29 3.1966 0.9493 XG2_1 21 2.7365 0.8988 XG2_2 12 1.9619 0.7895 XG3_1 15 2.1675 0.8004 XG3_2 22 2.7692 0.8959 XG4_1 14 2.055 0.7787 XG4_2 19 2.4304 0.8254 XG5_1 25 3.0611 0.951 XG5_2 15 1.9894 0.7346 XJ1_1 17 2.2633 0.7988 XJ1_2 14 2.2346 0.8467 XJ2_1 19 2.5793 0.876 XJ2_2 11 1.8417 0.768 XJ3_1 18 2.1775 0.7534 XJ3_2 12 1.9432 0.782 XJ4_1 13 1.8523 0.7222 XJ4_2 15 2.2064 0.8148 XJ5_1 12 1.9305 0.7769 XJ5_2 21 2.6664 0.8758 XN1_1 17 2.3056 0.8138 XN1_2 21 2.7569 0.9055 XN2_1 16 2.4022 0.8664 XN2_2 10 1.7982 0.7809 XN3_1 18 2.5455 0.8807 XN3_2 15 2.2528 0.8319 XN4_1 15 2.1885 0.8082 XN4_2 18 2.4169 0.8362 XN5_1 16 2.564 0.9248 XN5_2 13 2.0353 0.7935 XR1_1 17 2.3402 0.826 XR1_2 12 2.0757 0.8353 XS1_1 11 1.9654 0.8196 XS1_2 29 3.1842 0.9456 XS2_1 16 2.3648 0.8529 XS2_2 12 1.9814 0.7974 XS3_1 11 2.1321 0.8892 XS3_2 17 2.1603 0.7625 XS4_1 16 2.1631 0.7802 XS4_2 18 2.3741 0.8214 XS5_1 6 0.997 0.5564 XS5_2 16 2.4252 0.8747 注: S代表末端限制性片段数目, H′代表香农威尔指数, J′代表均匀度指数, —. 丰水期缺失采样点Note: S represents T-RF number, H′ represents Shannon-Wiener index, J′ represents evenness index, —. sampling sites absent in wet season 表 2 真核微型生物群落结构与理化因子相关性检验
Table 2 Correlation test between the microbial eukaryotic community structures of samples and physicochemical factors
指标Index 丰水期Wet season 枯水期Dry season 珠江广州段Guangzhou 西江广东段Xijiang 温度Temp 0.0021 0.1695* 0.1329* 0.0351 pH 0.0001 0.7469 0.0061 0.0089 盐度Salinity 0.0026 0.0005*** 0.0026 0.0576 溶氧DO 0.5599*** 0.4449*** 0.1294* 0.2305*** 化学需氧量CODCr 0.6170*** 0.6815*** 0.7687*** 0.8200*** 高锰酸盐指数CODMn 0.6797*** 0.7950*** 0.8313*** 0.8722*** 氨氮AN 0.6976*** 0.6352*** 0.6086*** 0.8681*** 总氮TN 0.5397*** 0.6638*** 0.4973*** 0.8047*** 总磷TP 0.7885*** 0.9289*** 0.1925* 0.9285* 注: 数字代表相关系数, * 表示显著性水平P<0.05Note: Number represents correlation coefficient, * indicates P<0.05 表 3 潜在的水质指示片段
Table 3 Potential indicative T-RFs
片段长度The length of T-RF A B P 优 76 0.9465 0.2500 0.009** 重度污染 83 0.9851 0.2222 0.033* 117 0.9934 0.2222 0.028* 192 1.0000 0.2222 0.032* 235 0.8198 0.2222 0.045* 376 1.0000 0.2222 0.027* 377 1.0000 0.2222 0.027* 391 1.0000 0.2222 0.035* 406 0.7552 0.3333 0.022* 注: A代表该片段仅在水质类别为优/重度污染的样本组间存在的概率, B代表该片段在水质类别为优/重度污染的样本组内存在的概率。P代表检验值, * 表示显著性水平P<0.05Note: A represents the probability that the T-RF were only present in high water quality samples/poor quality samples, B represents the probability that the T-RFs were detected among all high water quality samples/poor quality samples. P represents test value. * indicates P<0.05 附表 3 基于MICA工具的末端限制性片段对应物种预测 Appendix Tab. 3 Species prediction based on terminal restriction fragments and MICA tool 片段长度The length of T-RF GenBank登录号Accession number 物种分类Taxonomy 76 AB055190 Opisthokonta, Fungi, Dikarya, Basidiomycota, Pucciniomycotina, Cystobasidiomycetes, Cystobasidiales, Cystobasidiaceae, Cystobasidium, Cystobasidium sp. 76 FJ766481 Amoebozoa, Mycetozoa, Protosteliales, Protosteliaceae, Protostelium, Protostelium nocturnum 83 DQ504334 Alveolata, environmental samples 83 FJ648512 Archaeplastida, Viridiplantae, Chlorophyta, Trebouxiophyceae, Trebouxiophyceae incertae sedis, Coccomyxaceae, Coccomyxa, Coccomyxa simplex 83 EU446335 environmental samples 83 AB023471 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina,Saccharomycetes, Saccharomycetales, Saccharomycetales incertae sedis, Candida, Candida agrestis 83 AB013503 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Saccharomycetales incertaesedis, Candida melibiosica 83 AY242231 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Saccharomycetales incertaesedis, Candida, Candida sp. 83 AB013514 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 AY640202 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 DQ655699 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 AB013571 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 EF152415 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 EF152412 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 EF408189 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 X89518 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 AB013545 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 AB013547 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Clavispora, Clavispora/Candida clade 83 AY611608 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Candida, Candida sp. 83 AY520164 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Metschnikowia, Metschnikowia chrysomelidarum 83 AY452053 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Metschnikowia, Metschnikowia picachoensis 83 AY452055 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Metschnikowia, Metschnikowia pimensis 83 AJ508272 Opisthokonta, Fungi, Dikarya, Ascomycota, Saccharomycotina, Saccharomycetes, Saccharomycetales, Metschnikowiaceae, Metschnikowia, Metschnikowia rancensis 117 U57695 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chlamydomonas, Chlamydomonas mutabilis 117 U57696 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chlamydomonas, Chlamydomonas nivalis 117 DQ009749 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Microglena, Microglena reginae 117 AF517100 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chlamydomonas, unclassified Chlamydomonas 117 AJ410445 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chloromonas, Chloromonas actinochloris 117 U70783 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chloromonas, Chloromonas asteroidea 117 AF517095 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chloromonas, Chloromonas augustae 117 AF517092 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chloromonas, Chloromonas brevispina 117 GU117585 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chloromonas, Chloromonas insignis 117 AF514402 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chloromonas, Chloromonas rostafinskii 117 AF514401 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chloromonas, Chloromonas cf. platystigma 117 GU117584 Archaeplastida, Viridiplantae, Chlorophyta, Chlorophyceae, Chlamydomonadales, Chlamydomonadaceae, Chloromonas, Chloromonas cf. schussnigii -
[1] 柳青, 马健荣, 苏晓磊, 等. 珠江水系2006-2015年主要水质参数动态变化 [J]. 人民珠江, 2018, 39(12): 54-58, 67. doi: 10.3969/j.issn.1001-9235.2018.12.008 Liu Q, Ma J R, Su X L, et al. Dynamic changes of main water quality parameters in Pearl River (China) water system from 2006 to 2015 [J]. Pearl River, 2018, 39(12): 54-58, 67. doi: 10.3969/j.issn.1001-9235.2018.12.008
[2] 陈明媚. 珠江流域水污染治理的问题与对策 [D]. 广州: 华南理工大学, 2011 Chen M M. On the issues countermeasures to water pollution treatment in the Pearl River Basin [D]. Guangzhou: South China University of Technology, 2011
[3] 苟婷, 许振成, 李杰, 等. 珠江流域西江支流贺江浮游藻类群落特征及水质分析 [J]. 湖泊科学, 2015, 27(3): 412-420. doi: 10.18307/2015.0307 Gou T, Xu Z C, Li J, et al. Phytoplankton community structure and water quality assessment of Hejiang River, a branch of Xijiang River, Pearl River drainage basin [J]. Journal of Lake Sciences, 2015, 27(3): 412-420. doi: 10.18307/2015.0307
[4] 江涛, 黎坤, 柯栋. 珠江广州段水环境问题与保护对策 [J]. 湖泊科学, 2004, 16(3): 282-284. doi: 10.3321/j.issn:1003-5427.2004.03.015 Jiang T, Li K, Ke D. On the water environmental pollution and protection measures in the Guangzhou Section of Pearl River [J]. Journal of Lake Sciences, 2004, 16(3): 282-284. doi: 10.3321/j.issn:1003-5427.2004.03.015
[5] 童娟. 珠江流域概况及水文特性分析 [J]. 水利科技与经济, 2007, 13(1): 31-33. doi: 10.3969/j.issn.1006-7175.2007.01.011 Tong J. The generalizations and hydrological character analysis of Pearl River Basin [J]. Water Conservancy Science and Technology and Economy, 2007, 13(1): 31-33. doi: 10.3969/j.issn.1006-7175.2007.01.011
[6] 徐润林, 白庆笙, 谢瑞文. 珠江广州市段PFU原生动物群落特征及其与水质的关系 [J]. 生态学报, 2002, 22(4): 479-485. doi: 10.3321/j.issn:1000-0933.2002.04.005 Xu R L, Bai Q S, Xie R W. PF unit protozoan community characteristics and the relation with the water quality in the Guangzhou Reach of the Pearl River [J]. Acta Ecologica Sinica, 2002, 22(4): 479-485. doi: 10.3321/j.issn:1000-0933.2002.04.005
[7] 王朝晖, 胡韧, 谷阳光, 等. 珠江广州河段着生藻类的群落结构及其与水质的关系 [J]. 环境科学学报, 2009, 29(7): 1510-1516. doi: 10.3321/j.issn:0253-2468.2009.07.027 Wang Z H, Hu R, Gu Y G, et al. Community structures of periphyticalgae in the Guangzhou reaches of the Pearl River and their relationship to water quality [J]. Acta Scientiae Circumstantiae, 2009, 29(7): 1510-1516. doi: 10.3321/j.issn:0253-2468.2009.07.027
[8] Bik H M, Sung W, De Ley P, et al. Metagenetic community analysis of microbial eukaryotes illuminates biogeographic patterns in deep-sea and shallow water sediments [J]. Molecular Ecology, 2012, 21(5): 1048-1059. doi: 10.1111/j.1365-294X.2011.05297.x
[9] Foissner W. Evaluating water quality using protozoa and saprobity indexes [A]// John J L, Anthony T S (Eds.), Protocols in Protozoology [C]. Lawrence: Allen Press, 1992: B11.1-B11.20
[10] Chen M, Li X, Yang Q, et al. Soil eukaryotic microorganism succession as affected by continuous cropping of peanut-pathogenic and beneficial fungi were selected [J]. PLoS One, 2012, 7(7): e40659. doi: 10.1371/journal.pone.0040659
[11] Muhid P, Davis T W, Bunn S E, et al. Effects of inorganic nutrients in recycled water on freshwater phytoplankton biomass and composition [J]. Water Research, 2013, 47(1): 384-394. doi: 10.1016/j.watres.2012.10.015
[12] 许恒龙. 半封闭式海水养殖水体中浮游原生生物的生态学与毒理学研究 [D]. 青岛: 中国海洋大学, 2003 Xu H L. Ecology and toxicology of planktonic protists in semi-closed mariculture waters [D]. Qingdao: Ocean University of China, 2003
[13] 沈韫芬, 顾曼如, 龚循矩, 等. 微型生物监测新技术 [M]. 北京: 中国建筑工业出版社, 1990: 524 Shen Y F, Gu M R, Gong X J, et al. Modern Biomonitoring Techniques Using Freshwater Microbiota [M]. Beijing: China Architecture & Building Press, 1990: 524
[14] Xu K D, Choi J K, Yang E J, et al. Biomonitoring of coastal pollution status using protozoan communities with a modified PFU method [J]. Marine Pollution Bulletin, 2002, 44(9): 877-886. doi: 10.1016/S0025-326X(02)00090-5
[15] 张俊逸, 蒋江峦, 刘擎, 等. 珠江广州段微生物和浮游植物群落与水质特征研究 [J]. 水生态学杂志, 2011, 32(2): 38-46. Zhang J Y, Jiang J L, Liu Q, et al. The characteristics of microbial and phytoplankton community and water quality in the Guangzhou segment of Pearl River [J]. Journal of Hydroecology, 2011, 32(2): 38-46.
[16] Lueders T, Friedrich M W. Evaluation of PCR amplification bias by terminal restriction fragment length polymorphism analysis of small-subunit rRNA and mcrA genes by using defined template mixtures of methanogenic pure cultures and soil DNA extracts [J]. Applied and Environmental Microbiology, 2003, 69(1): 320-326. doi: 10.1128/AEM.69.1.320-326.2003
[17] 于洋, 王晓燕, 张鹏飞. 北运河水体浮游细菌群落的空间分布特征及其与水质的关系 [J]. 生态毒理学报, 2012, 7(3): 337-344. Yu Y, Wang X Y, Zhang P F. Spatial distribution of planktonic bacterial community and its relationship to water quality in Beiyun River [J]. Asian Journal of Ecotoxicology, 2012, 7(3): 337-344.
[18] 黄艺, 舒中亚. 基于浮游细菌生物完整性指数的河流生态系统健康评价——以滇池流域为例 [J]. 环境科学, 2013, 34(8): 3010-3018. Huang Y, Shu Z Y. Bacterioplankton index of Biotic Integrity (BP-IBI): an approach for assessing river ecosystem health in Dianchi Watershed [J]. Environmental Science, 2013, 34(8): 3010-3018.
[19] 董萍, 孙寓姣, 王红旗, 等. 利用T-RFLP技术对温榆河微生物群落结构研究 [J]. 中国环境科学, 2011, 31(4): 631-636. Dong P, Sun Y J, Wang H Q, et al. Study of micro-ecosystem of the Wenyu River by terminal restriction fragment length polymorphism analysis [J]. China Environmental Science, 2011, 31(4): 631-636.
[20] Stoeck T, Bass D, Nebel M, et al. Multiple marker parallel tag environmental DNA sequencing reveals a highly complex eukaryotic community in marine anoxic water [J]. Molecular Ecology, 2010(19): 21-31. doi: 10.1111/j.1365-294X.2009.04480.x
[21] Angela D K, Dan J S, Barbara J B, et al. Web-based phylogenetic assignment tool for analysis of terminal restriction fragment length polymorphism profiles of microbial communities [J]. Applied and environmental microbiology, 2003, 69(11): 6768-6776. doi: 10.1128/AEM.69.11.6768-6776.2003
[22] Oksanen J, Kindt R, Legendre P, et al. The vegan package [J]. Community Ecology Package, 2007(10): 631-637.
[23] Bray J R, Curtis J T. An ordination of the upland forest communities of southern Wisconsin [J]. Ecological Monographs, 1957(27): 325-349. doi: 10.2307/1942268
[24] Clarke K R, Somerfield P J, Gorley R N. Testing of null hypotheses in exploratory community analyses: similarity profiles and biota-environment linkage [J]. Journal of Experimental Marine Biology and Ecology, 2008, 366(1-2): 56-69. doi: 10.1016/j.jembe.2008.07.009
[25] Ter Braak C J. Canonical correspondence analysis: a new eigenvector technique for multivariate direct gradient analysis [J]. Ecology, 1986, 67(5): 1167-1179. doi: 10.2307/1938672
[26] Manly B F. Randomization, bootstrap and Monte Carlo methods in biology [M]. CRC Press, 1997: 330
[27] Shannon C E. The mathematical theory of communication [J]. Bell System Technical Journal, 1948, 27(3): 379-423. doi: 10.1002/j.1538-7305.1948.tb01338.x
[28] Pielou E C. Species-diversity and pattern-diversity in the study of ecological succession [J]. Journal of Theoretical Biology, 1966, 10(2): 370-383. doi: 10.1016/0022-5193(66)90133-0
[29] Cáceres M D, Legendre P. Associations between species and groups of sites: indices and statistical inference [J]. Ecology, 2009, 90(12): 3566-3574. doi: 10.1890/08-1823.1
[30] Shyu C, Soule T, Bent S J, et al. MiCA: A web-based tool for the analysis of microbial communities based on terminal-restriction fragment length polymorphisms of 16S and 18S rRNA genes [J]. Microbial Ecology, 2007, 53(4): 562-570. doi: 10.1007/s00248-006-9106-0
[31] Countway P D, Vigil P D, Schnetzer A, et al. Seasonal analysis of protistan community structure and diversity at the USC Microbial Observatory (San Pedro Channel, North Pacific Ocean) [J]. Limnology and Oceanography, 2010, 55(6): 2381-2396. doi: 10.4319/lo.2010.55.6.2381
[32] Kim D Y, Countway P D, Jones A C, et al. Monthly to interannual variability of microbial eukaryote assemblages at four depths in the eastern North Pacific [J]. The ISME Journal, 2014, 8(3): 515-530. doi: 10.1038/ismej.2013.173
[33] Zhang W, Pan Y, Yang J, et al. The diversity and biogeography of abundant and rare intertidal marine microeukaryotes explained by environment and dispersal limitation [J]. Environmental Microbiology, 2018, 20(2): 462-476. doi: 10.1111/1462-2920.13916
[34] Meadow J F, Zabinski C A. Spatial heterogeneity of eukaryotic microbial communities in an unstudied geothermal diatomaceous biological soil crust: Yellowstone National Park, WY, USA [J]. FEMS Microbiology Ecology, 2012, 82(1): 182-191. doi: 10.1111/j.1574-6941.2012.01416.x
[35] Sun Z, Li G, Wang C, et al. Community dynamics of prokaryotic and eukaryotic microbes in an estuary reservoir [J]. Scientific reports, 2014, 4: 6966.
[36] Scholz M, Martin R J. Control of bio-regenerated granular activated carbon by spreadsheet modelling [J]. Journal of Chemical Technology and Biotechnology, 1998, 71(3): 253-261. doi: 10.1002/(SICI)1097-4660(199803)71:3<253::AID-JCTB834>3.0.CO;2-Y
[37] Jiang J G, Shen Y F. Development of the microbial communities in Lake Donghu in relation to water quality [J]. Environmental Monitoring and Assessment, 2007, 127(1): 227-236.
[38] Montresor L C, Miranda-Filho K C, Paglia A, et al. Short-term toxicity of ammonia, sodium Hydroxide and a commercial biocide to golden mussel Limnoperna fortunei (Dunker, 1857) [J]. Ecotoxicology and Environmental Safety, 2013, 92: 150-154. doi: 10.1016/j.ecoenv.2013.03.016
[39] Hecky R, Kilham P. Nutrient limitation of phytoplankton in freshwater and marine environments: a review of recent evidence on the effects of enrichment [J]. Limnology and Oceanography, 1988, 33(4): 796-822.
[40] Bode A, Dortch Q. Uptake and regeneration of inorganic nitrogen in coastal waters influenced by the Mississippi River spatial and seasonal variations [J]. Journal of Plankton Research, 1996, 18(12): 2251-2268. doi: 10.1093/plankt/18.12.2251
[41] Lohrenz S E, Fahnenstiel G L, Redalje D G, et al. Variations in primary production of northern Gulf of Mexico continental shelf waters linked to nutrient inputs from the Mississippi River [J]. Marine Ecology Progress Series, 1997, 155: 45-54. doi: 10.3354/meps155045
[42] Yin K, Harrison P J, Beamish R J. Effects of a fluctuation in Fraser River discharge of primary production in the central strait of Georgia, British Columbia, Canada [J]. Canadian Journal of Fisheries and Aquatic Sciences, 1997, 54(5): 1015-1024. doi: 10.1139/f97-010
[43] Yin K, Goldblatt R H, Harrison P J, et al. Importance of wind and river discharge in influencing nutrient dynamics and phytoplankton production in summer in the central Strait of Georgia [J]. Marine Ecology Progress Series, 1997, 161: 173-183. doi: 10.3354/meps161173
[44] Tian C, Tan J, Wu X, et al. Spatiotemporal transition of bacterioplankton diversity in a large shallow hypertrophic freshwater lake, as determined by denaturing gradient gel electrophoresis [J]. Journal of Plankton Research, 2009, 31(8): 885-897. doi: 10.1093/plankt/fbp028
[45] 杨金鹏. 利用BPFU系统中原生生物群落对青岛近岸水质状况的初步评价 [D]. 青岛: 中国海洋大学, 2009 Yang J P. Primary assessment on water quality along the coast of Qingdao by protistan community in BPFU system [D]. Qingdao: Ocean University of China, 2009
-
期刊类型引用(2)
1. 陈家琪,董丽,麻晓梅,田凯,白洁,赵彦伟. 基于eDNA技术的白洋淀微型生物群落监测. 农业环境科学学报. 2021(08): 1773-1786+1839 .  百度学术
百度学术
2. 姚新民,陈云,沈燕,李禹昕,龚美萍,金凤,邵未艾,刁永卿,师乐,胡洁兰. 2013—2019年上海市青浦区出厂水水质状况及趋势分析. 职业与健康. 2021(21): 2976-2978+2982 .  百度学术
百度学术
其他类型引用(3)



 下载:
下载: